
| Sequence ID | X_DroMel_CAF1 |
|---|---|
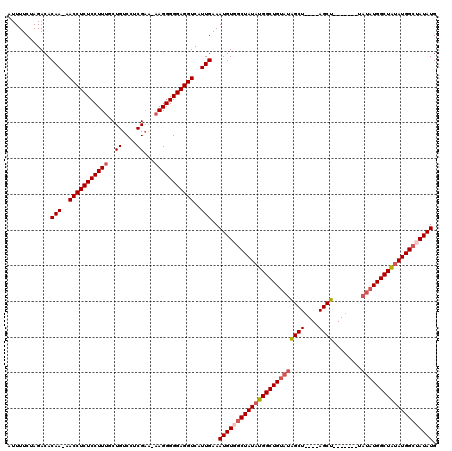
| Location | 15,950,820 – 15,950,968 |
| Length | 148 |
| Max. P | 0.998055 |

| Location | 15,950,820 – 15,950,928 |
|---|---|
| Length | 108 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 86.09 |
| Mean single sequence MFE | -44.04 |
| Consensus MFE | -34.22 |
| Energy contribution | -35.66 |
| Covariance contribution | 1.44 |
| Combinations/Pair | 1.05 |
| Mean z-score | -5.48 |
| Structure conservation index | 0.78 |
| SVM decision value | 2.99 |
| SVM RNA-class probability | 0.998055 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15950820 108 + 22224390 AUUUUCUAGACACAA-AACCUCUCCUUUACUGUCCUCGAA-AAGGGGGAGGUCAUUGAAAUGUGGCUAUAUGGCUGUAUGGCU----AGCUG------UAUAUGGCUAUAUGGCUAUAUG ............(((-.(((((((((((..((....))..-)))))))))))..)))..(((((((((((((((((((((.(.----....)------))))))))))))))))))))). ( -41.50) >DroSec_CAF1 19206 106 + 1 AUUUUCUAGACACAA-AACCUCUCCUUUGCUGUCCUCGAA-AAGGGGGAGGUCAUUGAAAUGUGACUAUAUGGCUGUAUAGCU----AGCU--------AUAUGGCUGUAUGGCUAUAUG ..........((((.-.(((((((((((..((....))..-)))))))))))........))))..(((((((((((((((((----(...--------...)))))))))))))))))) ( -39.10) >DroSim_CAF1 13697 106 + 1 AUUUUCUAGACACAA-AACCUCUCCUUUGCUGUCCUCGAA-AAGGGGGAGGUCAUUGAAAUGUGGCUAUAUGGCUGUAUAGCU----AGCU--------AUAUGGCUGUAUGGCUAUAUG ............(((-.(((((((((((..((....))..-)))))))))))..)))..((((((((((((((((((((((..----..))--------)))))))))))))))))))). ( -45.60) >DroEre_CAF1 17934 99 + 1 AUUUUCUAGACACAA-AACCUCUCCUUUGCUGUCCCCGCC--AGGGGGAGGUCAUUGAAAUGUGGCUAUAUGGCUG------------------UGGCUAUAUGGCUAUAUGGCGAUAUG ...............-.((((((((((.((.......)).--)))))))))).((((..(((((((((((((((..------------------..)))))))))))))))..))))... ( -41.20) >DroYak_CAF1 1282 120 + 1 AUUUUCUAGACACAAAAACCUCUCCUUUACUGUCCUCGAAAAAGGGGGAGGUCAUUGAAAUGUGGCUAUAUGGCUGUAUAGCUGUAUAGCUAUAUGGCUAUAUGGCUACAUGGCGAUAUG .................(((((((((((.............))))))))))).((((..((((((((((((((((((((((((....))))))))))))))))))))))))..))))... ( -52.82) >consensus AUUUUCUAGACACAA_AACCUCUCCUUUGCUGUCCUCGAA_AAGGGGGAGGUCAUUGAAAUGUGGCUAUAUGGCUGUAUAGCU____AGCU_______UAUAUGGCUAUAUGGCUAUAUG ............(((..(((((((((((..((....))...)))))))))))..)))..((((((((((((((((((((((((....))))........)))))))))))))))))))). (-34.22 = -35.66 + 1.44)


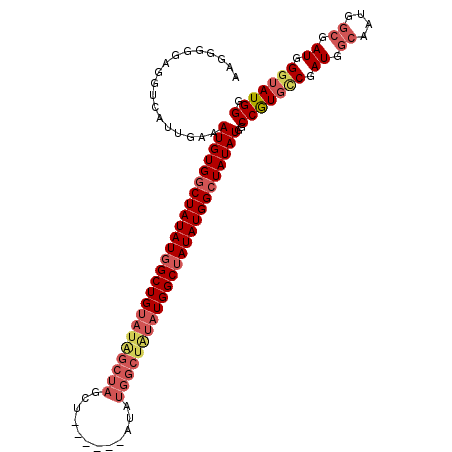
| Location | 15,950,859 – 15,950,968 |
|---|---|
| Length | 109 |
| Sequences | 4 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 85.80 |
| Mean single sequence MFE | -46.67 |
| Consensus MFE | -36.14 |
| Energy contribution | -39.07 |
| Covariance contribution | 2.94 |
| Combinations/Pair | 1.11 |
| Mean z-score | -6.57 |
| Structure conservation index | 0.77 |
| SVM decision value | 1.12 |
| SVM RNA-class probability | 0.917846 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15950859 109 + 22224390 AAGGGGGAGGUCAUUGAAAUGUGGCUAUAUGGCUGUAUGGCUAGCUG----UAUAUGGCUAUAUGGCUAUAUGGCUAUAUGGCCAUGCCGAUGGCAAUGGCGAUGGGUAUGGG ..................(((((((((((((((((((((((((....----....)))))))))))))))))))))))))..(((((((.((.((....)).)).))))))). ( -51.40) >DroSec_CAF1 19245 107 + 1 AAGGGGGAGGUCAUUGAAAUGUGACUAUAUGGCUGUAUAGCUAGCU------AUAUGGCUGUAUGGCUAUAUGGCUAUAUGGCCGUGCCGAUGGCAAUGGCGAUGGGUAUGGG ........((((((......))))))((((((((((((((((((((------....))))....))))))))))))))))..(((((((.((.((....)).)).))))))). ( -45.60) >DroSim_CAF1 13736 107 + 1 AAGGGGGAGGUCAUUGAAAUGUGGCUAUAUGGCUGUAUAGCUAGCU------AUAUGGCUGUAUGGCUAUAUGGCUAUAUGGCCGUGUCGAUGGCAAUGGCGAUGGGUAUGGG ..................(((((((((((((((((((((((((...------...)))))))))))))))))))))))))..((((..(.((.((....)).)).)..)))). ( -46.90) >DroEre_CAF1 17973 99 + 1 -AGGGGGAGGUCAUUGAAAUGUGGCUAUAUGGCUG------------UGGCUAUAUGGCUAUAUGGCGAUAUGGCUAUAUGGCCGUGCCGAUGCCGAUGCCAAUGGCGAUGG- -........(((((((..((.((((....((((..------------.((((((((((((((((....))))))))))))))))..))))..))))))..))))))).....- ( -42.80) >consensus AAGGGGGAGGUCAUUGAAAUGUGGCUAUAUGGCUGUAUAGCUAGCU______AUAUGGCUAUAUGGCUAUAUGGCUAUAUGGCCGUGCCGAUGGCAAUGGCGAUGGGUAUGGG ..................(((((((((((((((((((((((((............)))))))))))))))))))))))))..(((((((.((.((....)).)).))))))). (-36.14 = -39.07 + 2.94)

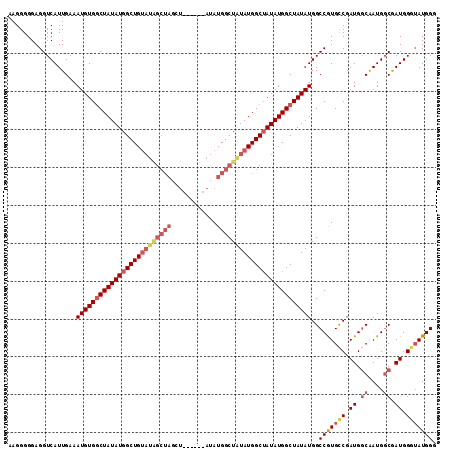
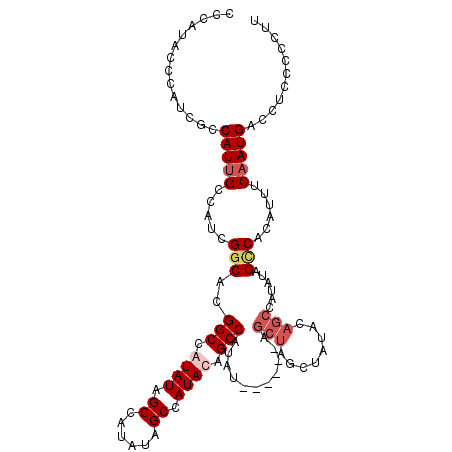
| Location | 15,950,859 – 15,950,968 |
|---|---|
| Length | 109 |
| Sequences | 4 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 85.80 |
| Mean single sequence MFE | -20.37 |
| Consensus MFE | -11.48 |
| Energy contribution | -13.10 |
| Covariance contribution | 1.62 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.85 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.630059 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15950859 109 - 22224390 CCCAUACCCAUCGCCAUUGCCAUCGGCAUGGCCAUAUAGCCAUAUAGCCAUAUAGCCAUAUA----CAGCUAGCCAUACAGCCAUAUAGCCACAUUUCAAUGACCUCCCCCUU ..............(((((.....(((.((((.((((.((......)).)))).))))((((----..(((........)))..)))))))......)))))........... ( -20.80) >DroSec_CAF1 19245 107 - 1 CCCAUACCCAUCGCCAUUGCCAUCGGCACGGCCAUAUAGCCAUAUAGCCAUACAGCCAUAU------AGCUAGCUAUACAGCCAUAUAGUCACAUUUCAAUGACCUCCCCCUU ............(((.........)))..(((..((((((....((((.(((......)))------.))))))))))..))).....((((........))))......... ( -20.90) >DroSim_CAF1 13736 107 - 1 CCCAUACCCAUCGCCAUUGCCAUCGACACGGCCAUAUAGCCAUAUAGCCAUACAGCCAUAU------AGCUAGCUAUACAGCCAUAUAGCCACAUUUCAAUGACCUCCCCCUU ..............(((((..........(((.((((.((..((((((.....(((.....------.))).))))))..)).)))).)))......)))))........... ( -17.89) >DroEre_CAF1 17973 99 - 1 -CCAUCGCCAUUGGCAUCGGCAUCGGCACGGCCAUAUAGCCAUAUCGCCAUAUAGCCAUAUAGCCA------------CAGCCAUAUAGCCACAUUUCAAUGACCUCCCCCU- -.......(((((((....))...(((..(((.((((.((......)).)))).)))((((.((..------------..)).)))).)))......)))))..........- ( -21.90) >consensus CCCAUACCCAUCGCCAUUGCCAUCGGCACGGCCAUAUAGCCAUAUAGCCAUACAGCCAUAU______AGCUAGCUAUACAGCCAUAUAGCCACAUUUCAAUGACCUCCCCCUU ..............(((((.....(((..(((.((((.((......)).)))).)))...........(((........)))......)))......)))))........... (-11.48 = -13.10 + 1.62)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:09:29 2006