
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 15,775,570 – 15,775,806 |
| Length | 236 |
| Max. P | 0.999341 |

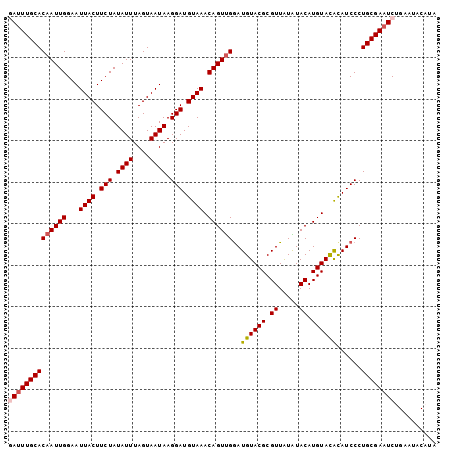
| Location | 15,775,570 – 15,775,673 |
|---|---|
| Length | 103 |
| Sequences | 5 |
| Columns | 103 |
| Reading direction | reverse |
| Mean pairwise identity | 95.05 |
| Mean single sequence MFE | -24.64 |
| Consensus MFE | -22.36 |
| Energy contribution | -22.76 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.33 |
| Structure conservation index | 0.91 |
| SVM decision value | 3.52 |
| SVM RNA-class probability | 0.999341 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15775570 103 - 22224390 GAUUUGCACAAUUGGAAUUACUUCUAUAUUUAGUAAUAAGGAUGUAAACAGUUGAAUGUACGCGUUAUAUACAUGUACGCAUCCCUGCGAAUCUGAAUACAUA ((((((((((((((...((((.(((.((((....)))).))).)))).))))))...(((((.((.....)).))))).......)))))))).......... ( -25.40) >DroSec_CAF1 6129 103 - 1 GAUUUGCACAAUUGGAAUUACUUCUAUAUUUAGUAAUAAGGAUGUAAACAGUUGGAUGUACGCGUUGUAUACAUGUACACAUCCCUGCGAAUCUGAAUACAUA ((((((((((((((...((((.(((.((((....)))).))).)))).))))))..((((((.((.....)).))))))......)))))))).......... ( -26.30) >DroSim_CAF1 4563 103 - 1 GAUUUGCACAAUUGGAAUUACUUCUAUAUUUAGUAAUAAGGAUGUAAACAGUUGGAUGUACGCGUUAUAUACAUGUACACAUCCCUGCGAAUCUGAAUACAUA ((((((((((((((...((((.(((.((((....)))).))).)))).))))))..((((((.((.....)).))))))......)))))))).......... ( -26.30) >DroEre_CAF1 6219 103 - 1 AAUUUGCACAAUUGGAAUUACUUCUAUAUUUAGUAAUAAGGAUGUAAACAGUUGGAUGUACGCGUUAUGUACAUGUAUAUAUCCCUGCGAAUCCGAAUACAUA .(((((((((((((...((((.(((.((((....)))).))).)))).))))))((((((..(((.......)))..))))))..)))))))........... ( -23.10) >DroYak_CAF1 5828 103 - 1 CAUUUGCACAAUUGGAAUUACUUCUAUAUUUAGUAAUAAGGAUGUAAACAGUCGGAUGUACGCGUUAUGUACAUGUAUAUAUCCCUGCGAUUCCGAAUACAUA ...........((((((((((.(((.((((....)))).))).))...(((..(((((((..(((.......)))..)))))))))).))))))))....... ( -22.10) >consensus GAUUUGCACAAUUGGAAUUACUUCUAUAUUUAGUAAUAAGGAUGUAAACAGUUGGAUGUACGCGUUAUAUACAUGUACACAUCCCUGCGAAUCUGAAUACAUA ((((((((((((((...((((.(((.((((....)))).))).)))).))))))..((((((.((.....)).))))))......)))))))).......... (-22.36 = -22.76 + 0.40)


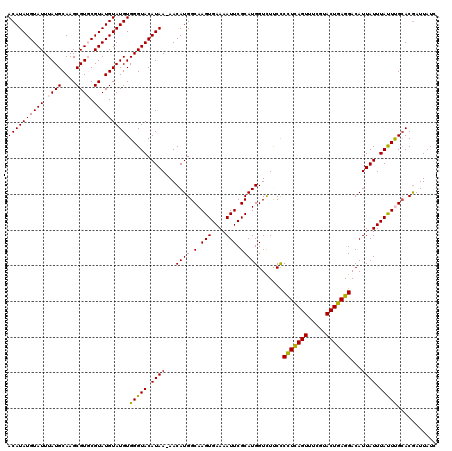
| Location | 15,775,673 – 15,775,766 |
|---|---|
| Length | 93 |
| Sequences | 5 |
| Columns | 94 |
| Reading direction | forward |
| Mean pairwise identity | 95.09 |
| Mean single sequence MFE | -26.94 |
| Consensus MFE | -24.22 |
| Energy contribution | -24.86 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.91 |
| Structure conservation index | 0.90 |
| SVM decision value | 2.42 |
| SVM RNA-class probability | 0.993663 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15775673 93 + 22224390 GCUAGUGGAGGUGUACAUAUGUAUUUAUGCAAGCGUGCGUAUGUAUGUGGGUACAUAA-AACAUGGCAAGUGAAAAUUCGCAUGGUCUUCUCCU ......((((((((((.(((((((.((((((....)))))).))))))).))))))..-.....(((..((((....))))...)))..)))). ( -28.30) >DroSec_CAF1 6232 93 + 1 UACAGUGGAGGUGUACAUAUGUAUUUAUGCAAGCGUGCGUAUGUAUGUGGGUACAUAA-AACAUGGCAAGUGAAAAUUCGCAUGGUCUCCCCCU ......((((((((((.(((((((.((((((....)))))).))))))).))))))..-..((((.(.(((....))).)))))..)))).... ( -27.80) >DroSim_CAF1 4666 93 + 1 GCCAGUGGAGGUGUACAUAUGUAUUUAUGCAAGCGUGCGUAUGUAUGUGGGUACAUAA-AACAUGGCAAGUGAAAAUUCGCAUGGUCUCCCCCU ((((((....((((((.(((((((.((((((....)))))).))))))).))))))..-.)).))))..((((....))))..((......)). ( -29.00) >DroEre_CAF1 6322 94 + 1 GCCAGUGGAGGUGUACAUAUGUAUUUAUGCAAGCGUUCGUAUGUAUGUGGGUACAUAAAAACAUGGCAAGUGAAAAUUCGCAUGGUUUUCCCUU ((((((....((((((.(((((((.((((........)))).))))))).))))))....)).))))(((.(((((((.....))))))).))) ( -24.80) >DroYak_CAF1 5931 94 + 1 GCCAGUGGAGGUGUACAUAUGUAUUUAUGCAAGCGUUCGUAUGUAUGUGGGUACAUAAAAACAUGGCAAGUGAAAAUUCGCAUGGUUUUCCCUU ((((((....((((((.(((((((.((((........)))).))))))).))))))....)).))))(((.(((((((.....))))))).))) ( -24.80) >consensus GCCAGUGGAGGUGUACAUAUGUAUUUAUGCAAGCGUGCGUAUGUAUGUGGGUACAUAA_AACAUGGCAAGUGAAAAUUCGCAUGGUCUUCCCCU ((((((....((((((.(((((((.((((((....)))))).))))))).))))))....)).))))..((((....))))............. (-24.22 = -24.86 + 0.64)



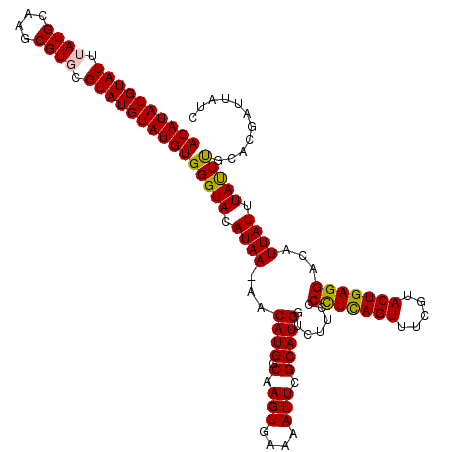
| Location | 15,775,687 – 15,775,806 |
|---|---|
| Length | 119 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.82 |
| Mean single sequence MFE | -29.93 |
| Consensus MFE | -27.80 |
| Energy contribution | -27.52 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.93 |
| SVM decision value | 1.88 |
| SVM RNA-class probability | 0.981066 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15775687 119 + 22224390 ACAUAUGUAUUUAUGCAAGCGUGCGUAUGUAUGUGGGUACAUAA-AACAUGGCAAGUGAAAAUUCGCAUGGUCUUCUCCUCAGUUUCACACUGAGGACAUUAUUUAUUUGGACGAUUAUC ...((((((((((((((.((....)).))))))..)))))))).-..((((.(.(((....))).)))))((((..((((((((.....))))))))............))))....... ( -29.44) >DroSec_CAF1 6246 119 + 1 ACAUAUGUAUUUAUGCAAGCGUGCGUAUGUAUGUGGGUACAUAA-AACAUGGCAAGUGAAAAUUCGCAUGGUCUCCCCCUUAGUUUCAUACUGAGGACAUUAUUUAUUUGCACGAUUAUC ...(((((.(((((((..((((((....))))))..)..)))))-))))))(((((((((.........((....))(((((((.....)))))))......)))))))))......... ( -29.30) >DroSim_CAF1 4680 119 + 1 ACAUAUGUAUUUAUGCAAGCGUGCGUAUGUAUGUGGGUACAUAA-AACAUGGCAAGUGAAAAUUCGCAUGGUCUCCCCCUUAGUUUCGUACUGAGGACAUUAUUUAUUUGCACGAUUAUC ...(((((.(((((((..((((((....))))))..)..)))))-))))))(((((((((.........((....))(((((((.....)))))))......)))))))))......... ( -29.30) >DroEre_CAF1 6336 120 + 1 ACAUAUGUAUUUAUGCAAGCGUUCGUAUGUAUGUGGGUACAUAAAAACAUGGCAAGUGAAAAUUCGCAUGGUUUUCCCUUCAGUUUCGUACUGAGGACAUUAUUUACUCGCACGAUUCUC (((((((.....(((....))).))))))).((((((((.((((((((...((.(((....))).))...))))...(((((((.....)))))))...)))).))))))))........ ( -30.80) >DroYak_CAF1 5945 120 + 1 ACAUAUGUAUUUAUGCAAGCGUUCGUAUGUAUGUGGGUACAUAAAAACAUGGCAAGUGAAAAUUCGCAUGGUUUUCCCUUCAGUUUCGUACUGAGGACAUUAUUUACUCGCACAAUUAUC (((((((.....(((....))).))))))).((((((((.((((((((...((.(((....))).))...))))...(((((((.....)))))))...)))).))))))))........ ( -30.80) >consensus ACAUAUGUAUUUAUGCAAGCGUGCGUAUGUAUGUGGGUACAUAA_AACAUGGCAAGUGAAAAUUCGCAUGGUCUUCCCCUCAGUUUCGUACUGAGGACAUUAUUUAUUUGCACGAUUAUC ...((((((((((((((.((....)).))))))..))))))))........(((((((((.........((....))(((((((.....)))))))......)))))))))......... (-27.80 = -27.52 + -0.28)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:06:47 2006