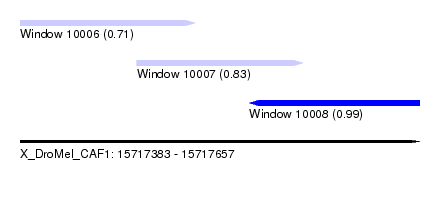
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 15,717,383 – 15,717,657 |
| Length | 274 |
| Max. P | 0.987427 |

| Location | 15,717,383 – 15,717,503 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.63 |
| Mean single sequence MFE | -38.38 |
| Consensus MFE | -32.70 |
| Energy contribution | -32.18 |
| Covariance contribution | -0.52 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.37 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.38 |
| SVM RNA-class probability | 0.714248 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15717383 120 + 22224390 CUGAUUGCGAGCCCAGUUUGCAGACACCCAGGUGAUUACUGUUUGCGUUUGCAUUUGCGGCGGCAUUAAGAUCACUUGAUAUUAGAUUAUUGAGCUGGUGCGUGGGCAGCGGUUUUGUUA ..((((((..(((((...(((((((.(.(((.......)))...).)))))))...(((.((((.((((((((...........)))).)))))))).))).))))).))))))...... ( -37.60) >DroSec_CAF1 866 120 + 1 CUGAUUGCGAGCCCAGUUUGCAGACACCCAGGUGAUUACUGUUUGCGUUUGCAUUCGCGGCGGCAUUAAGAUCACUUGAUAUUAGAUUAUUGAGCUGGUGCGUGGGCAGCGGCUUUGUUA ....((((..((((((..(((((((.(.(((.......)))...).)))))))..)(((.((((.((((((((...........)))).)))))))).))).))))).))))........ ( -37.30) >DroSim_CAF1 214 120 + 1 CUGAUUGCGAGCCCAGUUUGCAGACACCCAGGUGAUUACUGUUUGCGUUUGCAUUCGCGGCGGCAUUAAGAUCACUUGAUAUUAGAUUAUUGAGCUGGUGCGUGGGCAGCGGUUUUGUUA ..((((((..((((((..(((((((.(.(((.......)))...).)))))))..)(((.((((.((((((((...........)))).)))))))).))).))))).))))))...... ( -39.50) >DroEre_CAF1 890 114 + 1 CUGAUUGCGAGGCCAAUUUGCAGACACCCAGGUGAUUGCUGUUUGCGUUUGCA------GCGGCAUUAAGAUCACUUGAUAUUAGAUUAUUGAGCUGGUGCGAGGGCAGCGGUUUUGUCA ......((((((((.....((((.(((....))).)))).(((.((.((((((------.((((.((((((((...........)))).)))))))).)))))).))))))))))))).. ( -39.30) >DroYak_CAF1 246 114 + 1 CUGAUUGCGAACCCAGUUUGCAGACGCCCAGGUGAUUGCUGUUUGCGUUUGCA------GCGGCAUUAAGAUCACUUGAUAUUAGAUUAUUGAGCUGGUGCGAGGGCAGCGGUUUUGUCA .....(((((((...)))))))(((...(((((((((((((((.((....)))------)))))).....)))))))).............((((((.(((....))).)))))).))). ( -38.20) >consensus CUGAUUGCGAGCCCAGUUUGCAGACACCCAGGUGAUUACUGUUUGCGUUUGCAUU_GCGGCGGCAUUAAGAUCACUUGAUAUUAGAUUAUUGAGCUGGUGCGUGGGCAGCGGUUUUGUUA .....(((((((...)))))))((((..(((((((((......(((...(((.......))))))....))))))))).............((((((.(((....))).)))))))))). (-32.70 = -32.18 + -0.52)


| Location | 15,717,463 – 15,717,577 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.21 |
| Mean single sequence MFE | -35.06 |
| Consensus MFE | -30.28 |
| Energy contribution | -30.44 |
| Covariance contribution | 0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.44 |
| Structure conservation index | 0.86 |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.829576 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15717463 114 + 22224390 AUUAGAUUAUUGAGCUGGUGCGUGGGCAGCGGUUUUGUUACGUGCAAUUGGUCAAUAGUGU---UACUCGUGGCGGGGGAAUUGUGAGAUUUUUUGGUGUCGCUGC---UGCUGCCAGUA .....((((((((.(..(..(((((.(((.....))))))))..)....).))))))))..---......((((((.((....((((.(((....))).))))..)---).))))))... ( -35.70) >DroSec_CAF1 946 120 + 1 AUUAGAUUAUUGAGCUGGUGCGUGGGCAGCGGCUUUGUUACGUGCAAUUGGUCAAUAGUGUUAUUACUCGUGGCGGGGGAAUUGUGAGAUUUUUUGGUGUCGCUGCUGCUGCUGCCAGUA .....((((((((((((.(((....))).)))).((((.....))))....)))))))).....((((.(..((((.((....((((.(((....))).))))..)).))))..).)))) ( -39.30) >DroSim_CAF1 294 120 + 1 AUUAGAUUAUUGAGCUGGUGCGUGGGCAGCGGUUUUGUUACGUGCAAUUGGUCAAUAGUGUUAUUACUCGUGGCGGGGGAAUUGUGAGAUUUUUUGGUGUCGCUGCUGCUGCUGCCAGUA .....((((((((.(..(..(((((.(((.....))))))))..)....).)))))))).....((((.(..((((.((....((((.(((....))).))))..)).))))..).)))) ( -38.40) >DroEre_CAF1 964 111 + 1 AUUAGAUUAUUGAGCUGGUGCGAGGGCAGCGGUUUUGUCACAUGCAAUUGGUCAAUAGUGU---UACUUGUGGCGGGGGAAUUGUGAGAUUUUUUGGUGUCGCUG------CUGCCAGUA .............((((((((.((((((.(((...(.(((((.....((.((((..((...---..))..)))).)).....))))).)....))).)))).)))------).)))))). ( -30.80) >DroYak_CAF1 320 111 + 1 AUUAGAUUAUUGAGCUGGUGCGAGGGCAGCGGUUUUGUCACAUGCAAUUGGUCAAUAGUGU---UACUCGUGGCGGGGGAAUUGUGAGAUUUUUUGGUGUCGCUG------CUGCCAGUA .............((((((((.((((((.(((...(.(((((.....((.((((..((...---..))..)))).)).....))))).)....))).)))).)))------).)))))). ( -31.10) >consensus AUUAGAUUAUUGAGCUGGUGCGUGGGCAGCGGUUUUGUUACGUGCAAUUGGUCAAUAGUGU___UACUCGUGGCGGGGGAAUUGUGAGAUUUUUUGGUGUCGCUGC___UGCUGCCAGUA .....((((((((.(...(((((((((((.....))))).))))))...).))))))))...........((((((.......((((.(((....))).))))........))))))... (-30.28 = -30.44 + 0.16)


| Location | 15,717,540 – 15,717,657 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.29 |
| Mean single sequence MFE | -40.40 |
| Consensus MFE | -39.54 |
| Energy contribution | -39.74 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.53 |
| Structure conservation index | 0.98 |
| SVM decision value | 2.08 |
| SVM RNA-class probability | 0.987427 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15717540 117 - 22224390 AGCCUGCCAUCGCUGGGCGUUGGUGGCGCCGUAUCGCAUAGUCAUGACCACAUUGACGUUGGUGUGAUUCACGCCAGUUGUACUGGCAGCA---GCAGCGACACCAAAAAAUCUCACAAU .((((((....)).)))).((((((.(((.(((((((((.((((((....)).))))....)))))))....(((((.....)))))....---)).))).))))))............. ( -41.20) >DroSec_CAF1 1026 120 - 1 AGCCUGCCAUCGCUGGGCGUUGGUGGCGCCGUAUCGCAUAGUCAUGACCACAUUGACGUUGGUGUGAUCCACGCCAGUUGUACUGGCAGCAGCAGCAGCGACACCAAAAAAUCUCACAAU .((((((....)).)))).((((((.(((.(.(((((((.((((((....)).))))....))))))).)..(((((.....))))).((....)).))).))))))............. ( -41.50) >DroSim_CAF1 374 120 - 1 AGCCUGCCAUCGCUGGGCGUUGGUGGCGCCGUAUCGCAUAGUCAUGACCACAUUGACGUUGGUGUGAUCCACGCCAGUUGUACUGGCAGCAGCAGCAGCGACACCAAAAAAUCUCACAAU .((((((....)).)))).((((((.(((.(.(((((((.((((((....)).))))....))))))).)..(((((.....))))).((....)).))).))))))............. ( -41.50) >DroEre_CAF1 1041 114 - 1 AGCCUGCCACCGCUGGGCGUUGGUGGCGCCGUAUCGCAUAGUCAUGACCAUAUUGACGUUGGUGUGAUCCACGCCAGUUGUACUGGCAG------CAGCGACACCAAAAAAUCUCACAAU .((((((....)).)))).((((((.(((.(((((((((.((((.........))))....)))))))....(((((.....))))).)------).))).))))))............. ( -41.00) >DroYak_CAF1 397 114 - 1 AGCCUGCCAUCGCUGGGCGUUGGUGGCGCCGUAUCGCAUAGUCAUGACCAUAUUGACGUUGGUGUAAUUCACGCCAGUUGUACUGGCAG------CAGCGACACCAAAAAAUCUCACAAU .((((((....)).)))).((((((.(((.((...((((.((((.........))))....)))).......(((((.....))))).)------).))).))))))............. ( -36.80) >consensus AGCCUGCCAUCGCUGGGCGUUGGUGGCGCCGUAUCGCAUAGUCAUGACCACAUUGACGUUGGUGUGAUCCACGCCAGUUGUACUGGCAGCA___GCAGCGACACCAAAAAAUCUCACAAU .((((((....)).)))).((((((.(((.(.(((((((.((((.........))))....))))))).)..(((((.....)))))..........))).))))))............. (-39.54 = -39.74 + 0.20)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:05:38 2006