
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 15,678,817 – 15,679,014 |
| Length | 197 |
| Max. P | 0.677274 |

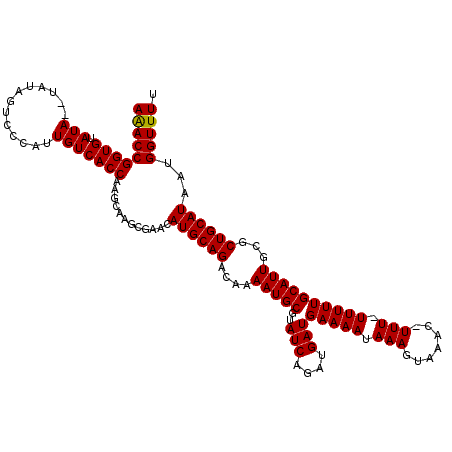
| Location | 15,678,817 – 15,678,934 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.73 |
| Mean single sequence MFE | -27.03 |
| Consensus MFE | -21.10 |
| Energy contribution | -20.94 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.78 |
| SVM decision value | 0.30 |
| SVM RNA-class probability | 0.677274 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15678817 117 - 22224390 AAACCGGUGAAUA--UAUAGUCCCAUUGUCACCAAGCAAGCGAACAUGCAGAUAAAAUGCGUAUCAGAUGAUGAAAAUAAAGUAAAC-UUUUUUUUUGCAUUGCGCUGCAUAAUGGUUUU ((((((((((...--.((......))..)))))..(((.(((...(((((.......)))))....((((.((((((.((((....)-))).)))))))))).)))))).....))))). ( -27.70) >DroSec_CAF1 6413 116 - 1 AAACCGGUGUAUA--UAUAGUCCCAUUGUCACCAAGCAAGCGAACAUGCAGACAAAAUGCGUAUCAGAUGAUGAAAAUAAAGUAAAC-UUU-UUUUUGCAUUGCGCUGCAUAAUGGUUUU (((((((((.(((--...........)))))))............((((((....(((((.((((....))))((((.((((....)-)))-.)))))))))...))))))...))))). ( -27.00) >DroSim_CAF1 3159 116 - 1 AAACCGGUGUAUA--UAUAGUACCAUUGUCACCAAGCAAGCGAACAUGCAGACAAAAUGCGUAUCAGAUGAUGAAAAUAAAGUAAAU-UUU-UUUUUGCAUUGCGCUGCAUAAUGGUUUU (((((((((.(((--...........)))))))..(((.(((...(((((((.(((((((.((((....))))........)))...-)))-).)))))))..)))))).....))))). ( -26.40) >DroEre_CAF1 5418 118 - 1 AGACCGGUGUAUAUAUAUAGUACCAUUGUCACCAAGCACACGAACAUGCAGACAAAAUGCGUAUCAGAGGAUGAAAAUAAAGUAAAC-UUU-UUUUUGCAUUGCUCUGCAUAAUGGUUUU (((((((((.(((.............)))))))............(((((((...(((((.((((....))))((((.((((....)-)))-.)))))))))..)))))))...))))). ( -27.52) >DroYak_CAF1 3177 117 - 1 AAACCGGUGUAUA--UAUAGUCCCAUUGUCACCAAGCACACGAACAUGCAGACAAAAUGCGUAUCAGAGGAUGAAAAUAAAGUAAACGUUU-UUUUUGCAUUGCUCUGCAUAAUGGUUUU (((((((((.(((--...........)))))))............(((((((...((((((....((((((((.............)))))-))).))))))..)))))))...))))). ( -26.52) >consensus AAACCGGUGUAUA__UAUAGUCCCAUUGUCACCAAGCAAGCGAACAUGCAGACAAAAUGCGUAUCAGAUGAUGAAAAUAAAGUAAAC_UUU_UUUUUGCAUUGCGCUGCAUAAUGGUUUU (((((((((.(((.............)))))))............((((((....(((((..(((....)))(((((.(((.......))).))))))))))...))))))...))))). (-21.10 = -20.94 + -0.16)


| Location | 15,678,896 – 15,679,014 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.75 |
| Mean single sequence MFE | -28.32 |
| Consensus MFE | -23.44 |
| Energy contribution | -24.24 |
| Covariance contribution | 0.80 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.29 |
| Structure conservation index | 0.83 |
| SVM decision value | -0.01 |
| SVM RNA-class probability | 0.527267 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15678896 118 - 22224390 UGAUCGUGUGUGGUAUGAUAAUGGAUACCUUAUCAUCGGAACGCACUUUGUUAUCUAGCAGAGCUGACUCACUUAUAUAAAAACCGGUGAAUA--UAUAGUCCCAUUGUCACCAAGCAAG ......(((.((((..((((((((((....(((((((((.........((((....))))(((....))).............)))))).)))--....)).)))))))))))).))).. ( -28.10) >DroSec_CAF1 6491 118 - 1 UGAUCGUGUGUGGUAUGAUAAUGGAUACCUUAUCAUCGGAACGCACUUUGUUAUCUAGCAGAGCCGACUCACUUAUAUAAAAACCGGUGUAUA--UAUAGUCCCAUUGUCACCAAGCAAG ......(((.((((..((((((((((....(((((((((.........((((....))))(((....))).............)))))).)))--....)).)))))))))))).))).. ( -28.10) >DroSim_CAF1 3237 118 - 1 UGAUCGUGUGUGGUAUGAUAAUGGAUACCUUAUCAUCGGAACGCACUUUGUUAUCUAGCAGUGCCGACUCACUUAUUUAAAAACCGGUGUAUA--UAUAGUACCAUUGUCACCAAGCAAG ......(((.((((..((((((((.(((..(((((((((...(((((..(((....)))))))).(.....)...........)))))).)))--....))))))))))))))).))).. ( -30.10) >DroEre_CAF1 5496 117 - 1 UGAUCGUGUGUGGUAUGAUA-UGGAUAACUUAUCAUCGGAAUACAC--UGUUAUCUAGCAGAGCCGACUCACUUAUAUAAAGACCGGUGUAUAUAUAUAGUACCAUUGUCACCAAGCACA .....((((.((((..((((-(((...((((((((((((......(--((((....)))))..........(((.....))).)))))).))).....))).))).)))))))).)))). ( -26.80) >DroYak_CAF1 3256 117 - 1 UGAUCGUGUGUGAUAUGAUA-UGGAUACCUUAUCAUCGGAAUACACCGUGUUAUCUAGCAGAGCCGACUCACUUAUAUAAAAACCGGUGUAUA--UAUAGUCCCAUUGUCACCAAGCACA .....(((((((((((((((-.((...)).)))))..(((((((((((((((....))))(((....)))..............)))))))).--.....)))...))))))...)))). ( -28.50) >consensus UGAUCGUGUGUGGUAUGAUAAUGGAUACCUUAUCAUCGGAACGCACUUUGUUAUCUAGCAGAGCCGACUCACUUAUAUAAAAACCGGUGUAUA__UAUAGUCCCAUUGUCACCAAGCAAG ......(((.(((..((((((((((((...(((((((((.........((((....))))(((....))).............)))))).)))..)))....)))))))))))).))).. (-23.44 = -24.24 + 0.80)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:04:39 2006