| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 15,639,206 – 15,639,317 |
| Length | 111 |
| Max. P | 0.919927 |

| Location | 15,639,206 – 15,639,317 |
|---|---|
| Length | 111 |
| Sequences | 4 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 93.42 |
| Mean single sequence MFE | -29.27 |
| Consensus MFE | -26.05 |
| Energy contribution | -25.67 |
| Covariance contribution | -0.38 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.00 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.13 |
| SVM RNA-class probability | 0.919927 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15639206 111 + 22224390 UGCUGUGCUGUGCAGUGCUGUAUGCGUUUAUUUGACAGCUUAUGAAUCACCCACAAAGAUUUAUUUGUGCUGAUUGCAGCACAACUGUUGCACCCCAU-UCAAAAAAAAAAC ((..((((...((((((((((.............)))))..(((((((.........))))))).(((((((....)))))))))))).))))..)).-............. ( -28.82) >DroSec_CAF1 3876 111 + 1 UGCUGUGCUGUGCAGUGCUGUAUGCGUUUAUUUGACAGCUUAUGAAUCACCCGCAAAGAUUUAUUUGUGCUGAUUGCAGCACAACUGUUGCACCCCAU-UCAAAAAAAAAAU ((..((((...((((((((((.............)))))..(((((((.........))))))).(((((((....)))))))))))).))))..)).-............. ( -28.82) >DroSim_CAF1 3868 111 + 1 UGCUGUGCUGUGCAGUGCUGUAUGCGUUUAUUUGACAGCUUAUGAAUCACCCGCAAAGAUUUAUUUGUGCUGAUUGCAGCACAACUGUUGCACCCCAU-UCAAAAAAAAAAC ((..((((...((((((((((.............)))))..(((((((.........))))))).(((((((....)))))))))))).))))..)).-............. ( -28.82) >DroEre_CAF1 3527 109 + 1 UGCUGUGCUGUGCUGAGCUGUGUGCAUUUAUUUGACAGCUUAUGAAUCACCCGCAAAGAUUCAUUUGCGCUGAUUGCAGCACAACUGUUGCACCCACUUUCAAAAAACA--- (((((((((((..((((((((...((......))))))))))..(((((..((((((......)))))).)))))))))))))......))).................--- ( -30.60) >consensus UGCUGUGCUGUGCAGUGCUGUAUGCGUUUAUUUGACAGCUUAUGAAUCACCCGCAAAGAUUUAUUUGUGCUGAUUGCAGCACAACUGUUGCACCCCAU_UCAAAAAAAAAAC (((((((((((.....(((((.............))))).....(((((..((((((......)))))).)))))))))))))......))).................... (-26.05 = -25.67 + -0.38)

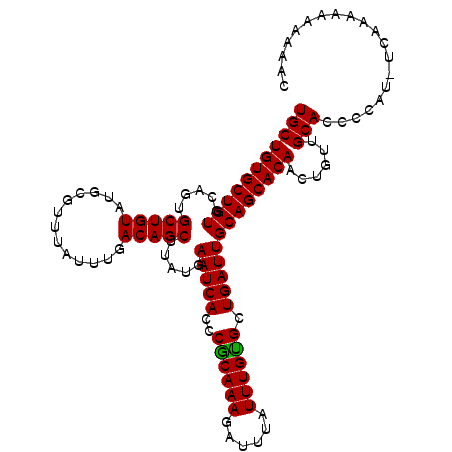

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:04:09 2006