
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 15,611,963 – 15,612,112 |
| Length | 149 |
| Max. P | 0.967607 |

| Location | 15,611,963 – 15,612,077 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 75.10 |
| Mean single sequence MFE | -47.75 |
| Consensus MFE | -25.75 |
| Energy contribution | -24.95 |
| Covariance contribution | -0.80 |
| Combinations/Pair | 1.45 |
| Mean z-score | -2.24 |
| Structure conservation index | 0.54 |
| SVM decision value | 1.61 |
| SVM RNA-class probability | 0.967607 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
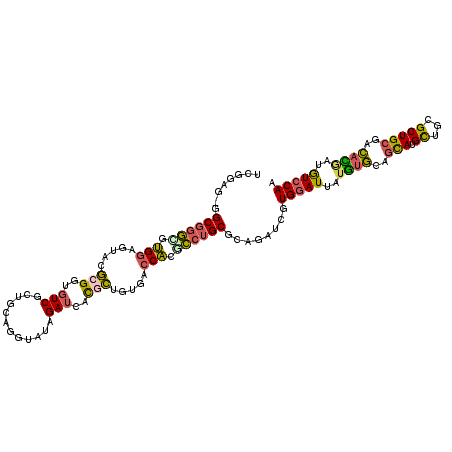
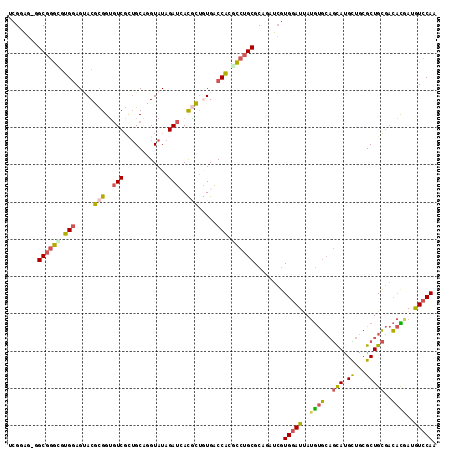
>X_DroMel_CAF1 15611963 114 - 22224390 ACGUAG-AGCGGGCGUGGAGUACGCGGUGUCGCUGCAGGUAUAGAUAAUGCUAUGACCACGCCUGCGCAAAUCGUGGAUUAUGUGCAGCAUGCUGCGCUGCGACACGAUAUCCAA .....(-.((((((((((.....((((.....)))).(((((.....)))))....)))))))))).)..((((((......((((((....)))))).....))))))...... ( -47.90) >DroVir_CAF1 19578 114 - 1 CCGGUU-GGCCACGUCGCAGUACUUGGGAUCCGAGCUGGUAUAGAGCACCGCCUGGCCGCUGAAGCACUGCGAUUGAAUCAAACUAUAUAUGUUGCGCUGGCUGCUCACAUCCAG .((((.-.((((.((((((((.(((.((..(((.((.(((.......))))).)))...)).))).)))))))))).....(((.......)))..))..))))........... ( -34.80) >DroSec_CAF1 412 114 - 1 UCGUAG-GGCGGGCGUGGAGUACGCGGUGUCGCUGCAUGUAUAGAUCACGCUGUGACCAAACCUGCGCAUGUCGUGGAUUAUGUGCAGCAUGCUGCGCUGCGACACGAUGUCCAA .....(-((((..((((..(((((((((((.((((((((((....((((((((((..........)))).).)))))..)))))))))))))))))).)))..)))).))))).. ( -51.00) >DroSim_CAF1 403 115 - 1 UCGUAGGGGCGGGCGUGGAGUACGCGGUGUCGCUGCAUGUAUAGAUCACGCUGUGACCAAACCUGCGCAUGUCGUGGAUUAUGUGCAGCAUGCUGCGCUGCGACACGAUGUCCAA ......(((((..((((..(((((((((((.((((((((((....((((((((((..........)))).).)))))..)))))))))))))))))).)))..)))).))))).. ( -50.80) >DroEre_CAF1 413 114 - 1 UCGGAG-CGCGGGCGUGGAGAACUCGGUGUCGUUGCAGGUAUAGAUCACAGUGUGGCCACGCCUGCGCAGAUCGUGGAUGAUGUGCAGCAUGUUGCGCUGCGACAUAAGGUCCAG .....(-(((((((((((...((.(.(((((............)).))).).))..))))))))))))......(((((.(((((((((.......))))).))))...))))). ( -50.20) >DroYak_CAF1 412 114 - 1 UCGGAG-CGCGGGUGUGGAGAACUCGGUGUCGUUGCAGGUAUAGAUGACGGUGUGGCCACGCCUGCGCAGAUCGUGGAUUAUGUGCAGCAUGCUGCGCUGCGACACAAUGUCCAG ..((((-(((((((((((....(.((.(((((((.........))))))).)).).)))))))))))).....(((......((((((....)))))).....)))....))).. ( -51.80) >consensus UCGGAG_GGCGGGCGUGGAGUACGCGGUGUCGCUGCAGGUAUAGAUCACGCUGUGACCACGCCUGCGCAGAUCGUGGAUUAUGUGCAGCAUGCUGCGCUGCGACACGAUGUCCAA ........((((((.(((.....(((..(((............)))..))).....))).))))))........(((((..((((..(((.((...)))))..))))..))))). (-25.75 = -24.95 + -0.80)

| Location | 15,612,000 – 15,612,112 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 73.45 |
| Mean single sequence MFE | -45.30 |
| Consensus MFE | -18.25 |
| Energy contribution | -18.57 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.46 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.40 |
| SVM decision value | 0.59 |
| SVM RNA-class probability | 0.791945 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
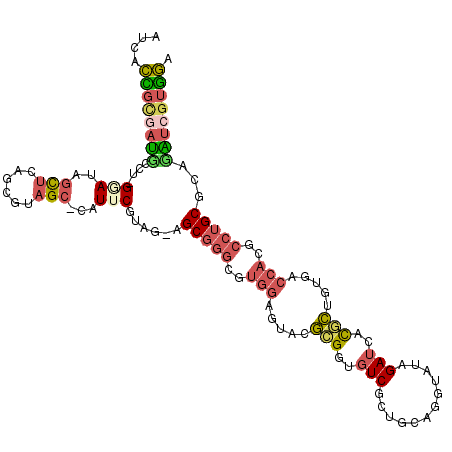
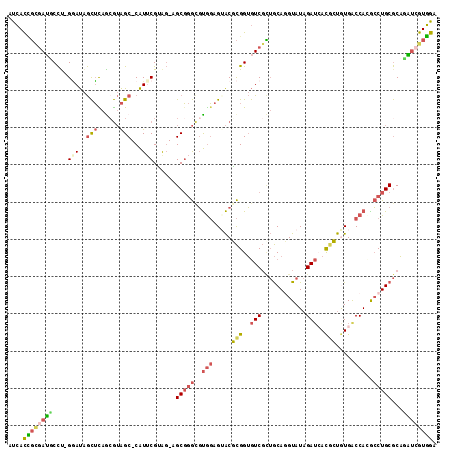
>X_DroMel_CAF1 15612000 112 - 22224390 AUCACCGCGAUUCCU-GGAUAGCUCAGCGUAGC-CAUACGUAG-AGCGGGCGUGGAGUACGCGGUGUCGCUGCAGGUAUAGAUAAUGCUAUGACCACGCCUGCGCAAAUCGUGGA ....((((((((..(-(....((((.(((((..-..))))).)-)))((((((((.....((((.....)))).(((((.....)))))....)))))))).))..)))))))). ( -48.40) >DroGri_CAF1 15410 112 - 1 AUGAUGCAGAGGCUC-GCACGAUGUAGCAAGGC-CGUCCGAGC-AGCCAAGUCACAGUGCUUGGGAUCCGAACAGGUAUAGAGCACAGCAUGACCAUGACUGCACUGCGAAUUAA ....(((((.((((.-((........))..)))-)......((-((.((.((((..((((((.....((.....))....))))))....))))..)).)))).)))))...... ( -34.00) >DroSec_CAF1 449 112 - 1 AUCACCGCGAUGCCU-GGAUAGCUCAGCGUAGC-CAUUCGUAG-GGCGGGCGUGGAGUACGCGGUGUCGCUGCAUGUAUAGAUCACGCUGUGACCAAACCUGCGCAUGUCGUGGA ....(((((((....-.....((((.((((.((-(........-)))..)))).)))).(((((((((((.((.((.......)).)).)))))...))).)))...))))))). ( -41.10) >DroSim_CAF1 440 115 - 1 AUCACCGCAAUGCCCGGGAUAGCUCAGCGUAGCCCAUUCGUAGGGGCGGGCGUGGAGUACGCGGUGUCGCUGCAUGUAUAGAUCACGCUGUGACCAAACCUGCGCAUGUCGUGGA ....((((.((((.((((...((((.((((.((((........))))..)))).)))).((((((((..(((......)))...))))))))......)))).))))...)))). ( -45.80) >DroEre_CAF1 450 112 - 1 AUCACUGUGAUUCCU-GAAUAGCUCAAGGUAGC-CAUUCGGAG-CGCGGGCGUGGAGAACUCGGUGUCGUUGCAGGUAUAGAUCACAGUGUGGCCACGCCUGCGCAGAUCGUGGA ....(..(((((.((-((((.(((......)))-.)))))).(-(((((((((((...((.(.(((((............)).))).).))..)))))))))))).)))))..). ( -47.70) >DroYak_CAF1 449 112 - 1 AUCACCGCGAUUCCU-GGAUAGCUUAACGUAGC-CAUUCGGAG-CGCGGGUGUGGAGAACUCGGUGUCGUUGCAGGUAUAGAUGACGGUGUGGCCACGCCUGCGCAGAUCGUGGA ....((((((((.((-((((.(((......)))-.)))))).(-(((((((((((....(.((.(((((((.........))))))).)).).)))))))))))).)))))))). ( -54.80) >consensus AUCACCGCGAUGCCU_GGAUAGCUCAGCGUAGC_CAUUCGUAG_AGCGGGCGUGGAGUACGCGGUGUCGCUGCAGGUAUAGAUCACGCUGUGACCACGCCUGCGCAGAUCGUGGA ....((((((((....(((..(((......)))...)))......(((((..(((.....(((..(((............)))..))).....)))..)))))...)))))))). (-18.25 = -18.57 + 0.31)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:03:40 2006