
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 15,605,952 – 15,606,109 |
| Length | 157 |
| Max. P | 0.941056 |

| Location | 15,605,952 – 15,606,072 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.42 |
| Mean single sequence MFE | -38.34 |
| Consensus MFE | -32.34 |
| Energy contribution | -33.34 |
| Covariance contribution | 1.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.84 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.31 |
| SVM RNA-class probability | 0.941056 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15605952 120 - 22224390 CCACGUUGUGCAGCGUGUGGCUGGAUAUUUCACUUAUCCAGCAUUUAUUGCUCGCAUCUAUGCUAAGUUGGCUAAUGCACAAUUGCGAUCGUCCGCCGACGGAGAAAACAAGCCAAGUAC ...(((.((((((((....((((((((.......))))))))......)))).))))..)))...(.((((((..((((....)))).((.((((....)))))).....)))))).).. ( -37.10) >DroSec_CAF1 4029 120 - 1 CCACAUUGUGCAGUGUGUGGCUGGAUAGUUCACUUAUCCAGCAUUUAUUGCUCGCAUCUAUGCUAAGUUGGCUAAUGCACAAUUGCGAUCGUCCGCCGACGGAGAAAACAAGCCAAGUAC .......(.((((((....(((((((((.....)))))))))...)))))).)(((....)))..(.((((((..((((....)))).((.((((....)))))).....)))))).).. ( -37.20) >DroSim_CAF1 3993 120 - 1 CCACAUUGUGCAGUGUGUGGCUGGAUAGUUCACUUAUCCAGCAUUUAUUGCUCGCAUCUAUGCUAAGUUGGCUAAUGCACAAUUGCGAUCGUCCGCCGACGGAGAAAACAAGCCAAGUAC .......(.((((((....(((((((((.....)))))))))...)))))).)(((....)))..(.((((((..((((....)))).((.((((....)))))).....)))))).).. ( -37.20) >DroEre_CAF1 4030 120 - 1 CUGCAUUGUGCAGUGUGUGGGUGGAUAGUUCACUUAUCCAGCAUUUAUUGCACGCAUCUAUGCUGAGUUGGCUAAUGCACAAUUGCGAUCGUCCGCCGACGGAGAAGACAAGCCAAGUAC ..(((((((((((((....(.(((((((.....))))))).)...))))))))).....))))..(.((((((..((((....)))).((.((((....)))))).....)))))).).. ( -37.60) >DroYak_CAF1 4042 120 - 1 CCACAUUGUGCAGUGUGUGGCUGGCUAGUUCACUUAUCCAGCAUUUAUUGCACGCAUCUAUGCUCAGUUGGCUAAUGCACAAUUGCGAUCGUCCGCCGACUGAGAAGACAAGCCAAGUGC .(((...((((((((....(((((..((....))...)))))...))))))))((.(((...((((((((((....(((....)))(......))))))))))).)))...))...))). ( -42.60) >consensus CCACAUUGUGCAGUGUGUGGCUGGAUAGUUCACUUAUCCAGCAUUUAUUGCUCGCAUCUAUGCUAAGUUGGCUAAUGCACAAUUGCGAUCGUCCGCCGACGGAGAAAACAAGCCAAGUAC .......(.((((((....(((((((((.....)))))))))...)))))).)(((....)))..(.((((((..((((....)))).((.((((....)))))).....)))))).).. (-32.34 = -33.34 + 1.00)

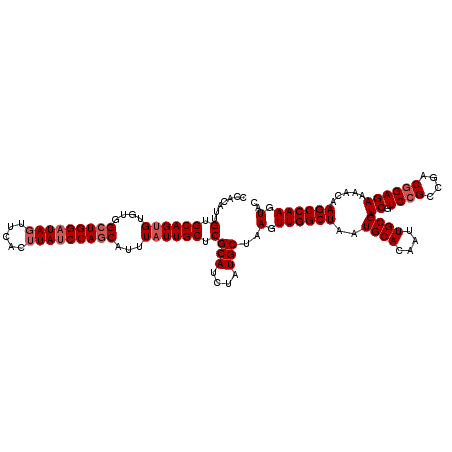

| Location | 15,605,992 – 15,606,109 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.52 |
| Mean single sequence MFE | -33.46 |
| Consensus MFE | -24.40 |
| Energy contribution | -24.76 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.16 |
| Mean z-score | -2.02 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.36 |
| SVM RNA-class probability | 0.706463 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15605992 117 + 22224390 GUGCAUUAGCCAACUUAGCAUAGAUGCGAGCAAUAAAUGCUGGAUAAGUGAAAUAUCCAGCCACACGCUGCACAACGUGGCCGCAUCCAAA---ACGAGUACCAUUGAAGCGCGACUAAU ((((.((((...((((......((((((.((.......((((((((.......))))))))..((((........))))))))))))....---..))))....)))).))))....... ( -35.20) >DroSec_CAF1 4069 117 + 1 GUGCAUUAGCCAACUUAGCAUAGAUGCGAGCAAUAAAUGCUGGAUAAGUGAACUAUCCAGCCACACACUGCACAAUGUGGUCGCAUCCAGA---ACGGGUACCAUUGAAGCGCGACUGAU ((((.((((...((((......(((((((.........((((((((.(....)))))))))..((((........)))).)))))))....---..))))....)))).))))....... ( -33.10) >DroSim_CAF1 4033 117 + 1 GUGCAUUAGCCAACUUAGCAUAGAUGCGAGCAAUAAAUGCUGGAUAAGUGAACUAUCCAGCCACACACUGCACAAUGUGGUCGCAUCCAGA---ACGGGUACCAUUGAAGCGCGACUGAU ((((.((((...((((......(((((((.........((((((((.(....)))))))))..((((........)))).)))))))....---..))))....)))).))))....... ( -33.10) >DroEre_CAF1 4070 117 + 1 GUGCAUUAGCCAACUCAGCAUAGAUGCGUGCAAUAAAUGCUGGAUAAGUGAACUAUCCACCCACACACUGCACAAUGCAGCAUCAUCUACA---ACGAGUACUAUUGAAGCGCGAUUAAU ((((.((((...((((....((((((.((((.........((((((.(....))))))).........(((.....)))))))))))))..---..))))....)))).))))....... ( -29.80) >DroYak_CAF1 4082 120 + 1 GUGCAUUAGCCAACUGAGCAUAGAUGCGUGCAAUAAAUGCUGGAUAAGUGAACUAGCCAGCCACACACUGCACAAUGUGGCAUCAUCUAGAGGUACUAGUACCAUUGAGGCGCGACUAAU ((((.(((((((.....(((....)))(((((......(((((...((....))..))))).......)))))....))))..........((((....))))..))).))))....... ( -36.12) >consensus GUGCAUUAGCCAACUUAGCAUAGAUGCGAGCAAUAAAUGCUGGAUAAGUGAACUAUCCAGCCACACACUGCACAAUGUGGCCGCAUCCAGA___ACGAGUACCAUUGAAGCGCGACUAAU ((((.((((...((((......((((((.((.......((((((((.......))))))))..((((........)))))))))))).........))))....)))).))))....... (-24.40 = -24.76 + 0.36)

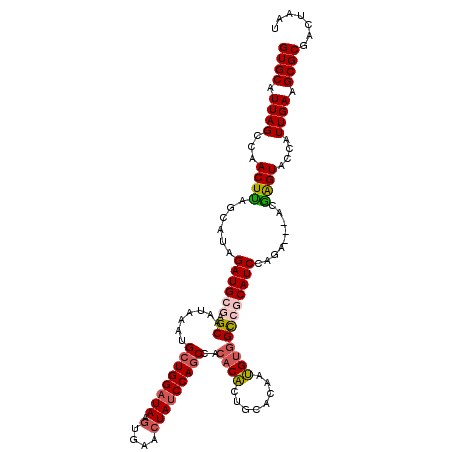
| Location | 15,605,992 – 15,606,109 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.52 |
| Mean single sequence MFE | -37.90 |
| Consensus MFE | -29.38 |
| Energy contribution | -30.50 |
| Covariance contribution | 1.12 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.24 |
| Structure conservation index | 0.78 |
| SVM decision value | 1.31 |
| SVM RNA-class probability | 0.940006 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15605992 117 - 22224390 AUUAGUCGCGCUUCAAUGGUACUCGU---UUUGGAUGCGGCCACGUUGUGCAGCGUGUGGCUGGAUAUUUCACUUAUCCAGCAUUUAUUGCUCGCAUCUAUGCUAAGUUGGCUAAUGCAC ((((((((.(((....(((((.....---..((((((((((((((((....))))))..((((((((.......)))))))).......)).)))))))))))))))))))))))).... ( -41.21) >DroSec_CAF1 4069 117 - 1 AUCAGUCGCGCUUCAAUGGUACCCGU---UCUGGAUGCGACCACAUUGUGCAGUGUGUGGCUGGAUAGUUCACUUAUCCAGCAUUUAUUGCUCGCAUCUAUGCUAAGUUGGCUAAUGCAC ....((((((((..(((((...))))---)..)).))))))......(.((((((....(((((((((.....)))))))))...)))))).)((((....(((.....)))..)))).. ( -38.90) >DroSim_CAF1 4033 117 - 1 AUCAGUCGCGCUUCAAUGGUACCCGU---UCUGGAUGCGACCACAUUGUGCAGUGUGUGGCUGGAUAGUUCACUUAUCCAGCAUUUAUUGCUCGCAUCUAUGCUAAGUUGGCUAAUGCAC ....((((((((..(((((...))))---)..)).))))))......(.((((((....(((((((((.....)))))))))...)))))).)((((....(((.....)))..)))).. ( -38.90) >DroEre_CAF1 4070 117 - 1 AUUAAUCGCGCUUCAAUAGUACUCGU---UGUAGAUGAUGCUGCAUUGUGCAGUGUGUGGGUGGAUAGUUCACUUAUCCAGCAUUUAUUGCACGCAUCUAUGCUGAGUUGGCUAAUGCAC .........((.....(((((((((.---.(((((((.((.((((..((((..((..((((((((...))))))))..))))))....)))))))))))))..)))))..))))..)).. ( -34.20) >DroYak_CAF1 4082 120 - 1 AUUAGUCGCGCCUCAAUGGUACUAGUACCUCUAGAUGAUGCCACAUUGUGCAGUGUGUGGCUGGCUAGUUCACUUAUCCAGCAUUUAUUGCACGCAUCUAUGCUCAGUUGGCUAAUGCAC ((((((((((((.....))).((((.....))))..(((((......((((((((....(((((..((....))...)))))...)))))))))))))........).)))))))).... ( -36.30) >consensus AUUAGUCGCGCUUCAAUGGUACUCGU___UCUGGAUGCGGCCACAUUGUGCAGUGUGUGGCUGGAUAGUUCACUUAUCCAGCAUUUAUUGCUCGCAUCUAUGCUAAGUUGGCUAAUGCAC ((((((((((((.....)))...........((((((((((((((((....)))))).)(((((((((.....))))))))).........)))))))))......).)))))))).... (-29.38 = -30.50 + 1.12)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:03:30 2006