
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 1,666,515 – 1,666,687 |
| Length | 172 |
| Max. P | 0.999857 |

| Location | 1,666,515 – 1,666,612 |
|---|---|
| Length | 97 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 87.78 |
| Mean single sequence MFE | -46.30 |
| Consensus MFE | -41.74 |
| Energy contribution | -41.42 |
| Covariance contribution | -0.32 |
| Combinations/Pair | 1.06 |
| Mean z-score | -3.16 |
| Structure conservation index | 0.90 |
| SVM decision value | 4.22 |
| SVM RNA-class probability | 0.999840 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1666515 97 + 22224390 -----------------------CCACCGUCAGCAUCCAUGCCUGCAGUCUACAGCUUGCAGUCUGCGGUGGCGCACACCUACCGCAGGAUGCAGGUGGUGUGGUCAGUGUCAGCCUGUU -----------------------.((((..(.((((((....((((((.(....).))))))..((((((((.(....))))))))))))))).)..)))).(((........))).... ( -39.10) >DroSec_CAF1 827 97 + 1 -----------------------CCACCAUCAGCAUCCAUGCCUGCAGUCUACAGCUUGCAGUCUGCGGUGGCGCACACCUACCGCAGGAUGCAGGUGGUGUGGUCAGUGUCAGCCUGUU -----------------------.(((((((.((((((....((((((.(....).))))))..((((((((.(....))))))))))))))).))))))).(((........))).... ( -41.70) >DroSim_CAF1 1707 97 + 1 -----------------------CCACCAUCAGCAUCCAUGCCUGCAGUCUACAGCUUGCAGUCUGCGGUGGCGCACACCUACCGCAGGAUGCAGGUGGUGUGGUCAGUGUCAGCCUGUU -----------------------.(((((((.((((((....((((((.(....).))))))..((((((((.(....))))))))))))))).))))))).(((........))).... ( -41.70) >DroEre_CAF1 628 103 + 1 -----------------CACCAGCCACCAUCAGCAUCCAAGCCUGCAGUCUACAGCUUGCAGUCUGCGGUGGCGCACACCCACCGCAGGAUGCAGGUGGUGUGGUCAGCGUCAGCCUGUU -----------------.((((..(((((((.((((((.((.((((((.(....).)))))).))(((((((.(....)))))))).)))))).)))))))))))((((....).))).. ( -47.30) >DroYak_CAF1 1811 120 + 1 AUGUAACAGGCAGAAGGCACCAGGCACCAUCAGCAUCCAUGACUGCAGUGUACAGCUUGCAGUCUGCGGUGGCGCACACCUACCGCAGGAUGCAGGUGGUGUGGUCAGUGUCAGCCUGUU ....(((((((....(((((...((((((((.((((((..(((((((((.....)).)))))))((((((((.(....))))))))))))))).)))))))).....))))).))))))) ( -61.70) >consensus _______________________CCACCAUCAGCAUCCAUGCCUGCAGUCUACAGCUUGCAGUCUGCGGUGGCGCACACCUACCGCAGGAUGCAGGUGGUGUGGUCAGUGUCAGCCUGUU ........................(((((((.((((((....(((((((.....)).)))))..((((((((.(....))))))))))))))).))))))).(((........))).... (-41.74 = -41.42 + -0.32)

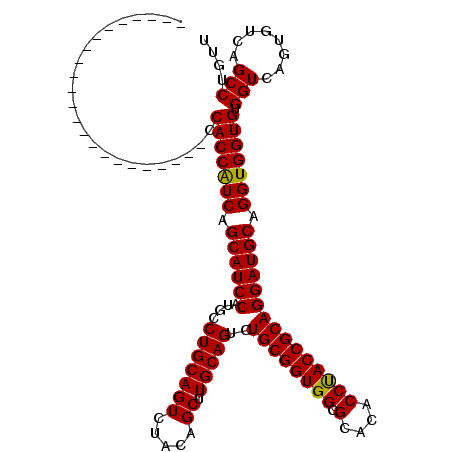
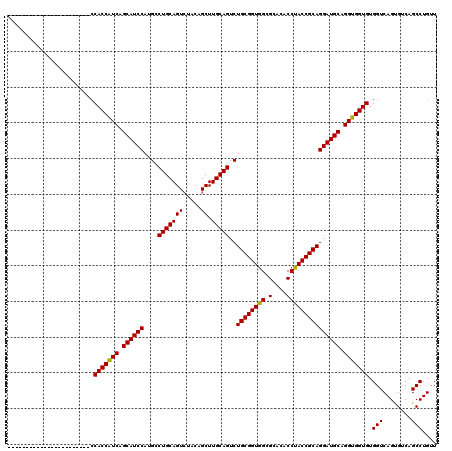
| Location | 1,666,515 – 1,666,612 |
|---|---|
| Length | 97 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.78 |
| Mean single sequence MFE | -46.62 |
| Consensus MFE | -39.46 |
| Energy contribution | -39.86 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.20 |
| Structure conservation index | 0.85 |
| SVM decision value | 3.57 |
| SVM RNA-class probability | 0.999402 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1666515 97 - 22224390 AACAGGCUGACACUGACCACACCACCUGCAUCCUGCGGUAGGUGUGCGCCACCGCAGACUGCAAGCUGUAGACUGCAGGCAUGGAUGCUGACGGUGG----------------------- ..(((.......)))......(((((.((((((((((((.(((....))))))((((.((((.....)))).))))..))).))))))....)))))----------------------- ( -42.10) >DroSec_CAF1 827 97 - 1 AACAGGCUGACACUGACCACACCACCUGCAUCCUGCGGUAGGUGUGCGCCACCGCAGACUGCAAGCUGUAGACUGCAGGCAUGGAUGCUGAUGGUGG----------------------- ..(((.......)))....(((((.(.((((((((((((.(((....))))))((((.((((.....)))).))))..))).)))))).).))))).----------------------- ( -43.30) >DroSim_CAF1 1707 97 - 1 AACAGGCUGACACUGACCACACCACCUGCAUCCUGCGGUAGGUGUGCGCCACCGCAGACUGCAAGCUGUAGACUGCAGGCAUGGAUGCUGAUGGUGG----------------------- ..(((.......)))....(((((.(.((((((((((((.(((....))))))((((.((((.....)))).))))..))).)))))).).))))).----------------------- ( -43.30) >DroEre_CAF1 628 103 - 1 AACAGGCUGACGCUGACCACACCACCUGCAUCCUGCGGUGGGUGUGCGCCACCGCAGACUGCAAGCUGUAGACUGCAGGCUUGGAUGCUGAUGGUGGCUGGUG----------------- ....(((....))).((((..(((((.(((((((((((((((....).)))))))))....((((((((.....))).))))))))))....))))).)))).----------------- ( -47.40) >DroYak_CAF1 1811 120 - 1 AACAGGCUGACACUGACCACACCACCUGCAUCCUGCGGUAGGUGUGCGCCACCGCAGACUGCAAGCUGUACACUGCAGUCAUGGAUGCUGAUGGUGCCUGGUGCCUUCUGCCUGUUACAU (((((((.((.....(((((((((.(.((((((((((((.(((....)))))))))(((((((..........)))))))..)))))).).)))))..))))....)).))))))).... ( -57.00) >consensus AACAGGCUGACACUGACCACACCACCUGCAUCCUGCGGUAGGUGUGCGCCACCGCAGACUGCAAGCUGUAGACUGCAGGCAUGGAUGCUGAUGGUGG_______________________ ..(((.......)))....(((((.(.((((((((((((.(((....)))))))))..(((((..(....)..)))))....)))))).).)))))........................ (-39.46 = -39.86 + 0.40)

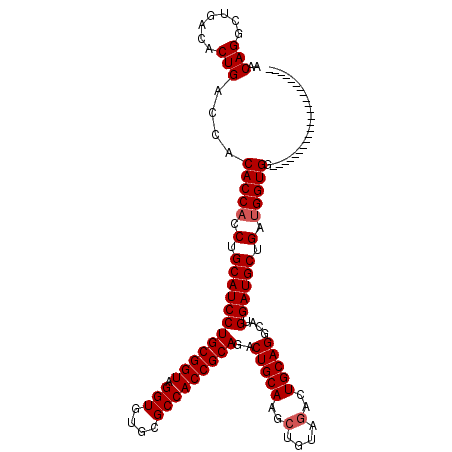
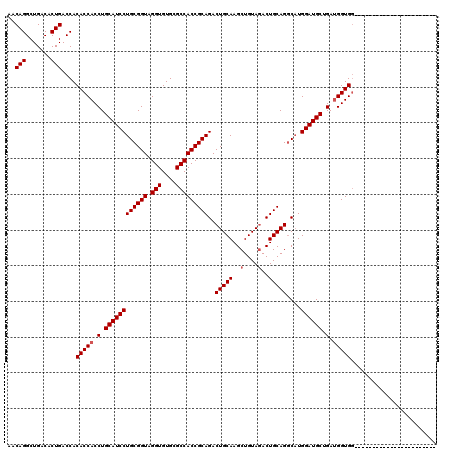
| Location | 1,666,532 – 1,666,652 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.33 |
| Mean single sequence MFE | -57.16 |
| Consensus MFE | -54.80 |
| Energy contribution | -55.04 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.02 |
| Mean z-score | -3.84 |
| Structure conservation index | 0.96 |
| SVM decision value | 4.27 |
| SVM RNA-class probability | 0.999857 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1666532 120 + 22224390 GCCUGCAGUCUACAGCUUGCAGUCUGCGGUGGCGCACACCUACCGCAGGAUGCAGGUGGUGUGGUCAGUGUCAGCCUGUUGCUGCGCGUUAUCAAAUGCAGGAGUUGUAUGCAACAGGCA (((((..((.((((((((((((((((((.((((.((((((.(((((.....)).))))))))))))).)).)))......)))))(((((....)))))..)))))))).))..))))). ( -56.40) >DroSec_CAF1 844 120 + 1 GCCUGCAGUCUACAGCUUGCAGUCUGCGGUGGCGCACACCUACCGCAGGAUGCAGGUGGUGUGGUCAGUGUCAGCCUGUUGCUGCGCGUUAUCAAAUGCAGGAGUUGUAUGCAACAGGCA (((((..((.((((((((((((((((((.((((.((((((.(((((.....)).))))))))))))).)).)))......)))))(((((....)))))..)))))))).))..))))). ( -56.40) >DroSim_CAF1 1724 120 + 1 GCCUGCAGUCUACAGCUUGCAGUCUGCGGUGGCGCACACCUACCGCAGGAUGCAGGUGGUGUGGUCAGUGUCAGCCUGUUGCUGCGCGUUAUCAAAUGCAGGAGUUGUAUGCAACAGGCA (((((..((.((((((((((((((((((.((((.((((((.(((((.....)).))))))))))))).)).)))......)))))(((((....)))))..)))))))).))..))))). ( -56.40) >DroEre_CAF1 651 120 + 1 GCCUGCAGUCUACAGCUUGCAGUCUGCGGUGGCGCACACCCACCGCAGGAUGCAGGUGGUGUGGUCAGCGUCAGCCUGUUGCUGCGCGUUAUCAAAUGCAGGAGUUGUAUGCAACAGGCA .(((((((.((((((((((((..(((((((((.(....))))))))))..)))))))..))))).).(((.((((.....))))))).........)))))).((((....))))..... ( -59.10) >DroYak_CAF1 1851 120 + 1 GACUGCAGUGUACAGCUUGCAGUCUGCGGUGGCGCACACCUACCGCAGGAUGCAGGUGGUGUGGUCAGUGUCAGCCUGUUGCUGCGCGUUAUCAAAUGCAGCAGUUGUAUGCAACAGGCA (((((((((.....)).))))))).(((.((((.((((((.(((((.....)).))))))))))))).)))..(((((((((((((((((....))))).))).......))))))))). ( -57.51) >consensus GCCUGCAGUCUACAGCUUGCAGUCUGCGGUGGCGCACACCUACCGCAGGAUGCAGGUGGUGUGGUCAGUGUCAGCCUGUUGCUGCGCGUUAUCAAAUGCAGGAGUUGUAUGCAACAGGCA (((((..(..((((((((((((((((((.((((.((((((.(((((.....)).))))))))))))).)).)))......)))))(((((....)))))..))))))))..)..))))). (-54.80 = -55.04 + 0.24)

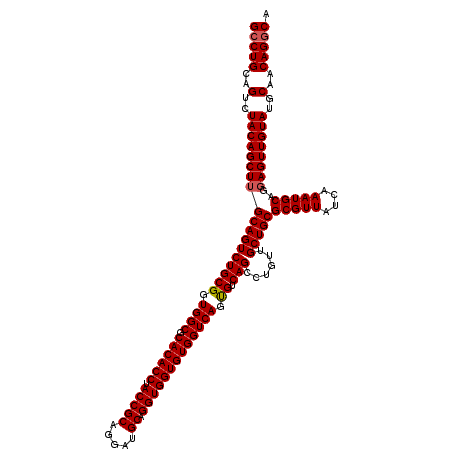
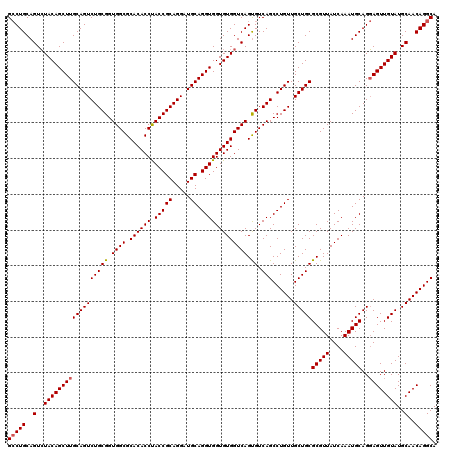
| Location | 1,666,532 – 1,666,652 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.33 |
| Mean single sequence MFE | -45.30 |
| Consensus MFE | -42.92 |
| Energy contribution | -43.32 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.57 |
| Structure conservation index | 0.95 |
| SVM decision value | 3.63 |
| SVM RNA-class probability | 0.999467 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1666532 120 - 22224390 UGCCUGUUGCAUACAACUCCUGCAUUUGAUAACGCGCAGCAACAGGCUGACACUGACCACACCACCUGCAUCCUGCGGUAGGUGUGCGCCACCGCAGACUGCAAGCUGUAGACUGCAGGC .(((((((((..........(((............)))))))))))).................((((((..(((((((.(((....)))))))))).((((.....))))..)))))). ( -45.10) >DroSec_CAF1 844 120 - 1 UGCCUGUUGCAUACAACUCCUGCAUUUGAUAACGCGCAGCAACAGGCUGACACUGACCACACCACCUGCAUCCUGCGGUAGGUGUGCGCCACCGCAGACUGCAAGCUGUAGACUGCAGGC .(((((((((..........(((............)))))))))))).................((((((..(((((((.(((....)))))))))).((((.....))))..)))))). ( -45.10) >DroSim_CAF1 1724 120 - 1 UGCCUGUUGCAUACAACUCCUGCAUUUGAUAACGCGCAGCAACAGGCUGACACUGACCACACCACCUGCAUCCUGCGGUAGGUGUGCGCCACCGCAGACUGCAAGCUGUAGACUGCAGGC .(((((((((..........(((............)))))))))))).................((((((..(((((((.(((....)))))))))).((((.....))))..)))))). ( -45.10) >DroEre_CAF1 651 120 - 1 UGCCUGUUGCAUACAACUCCUGCAUUUGAUAACGCGCAGCAACAGGCUGACGCUGACCACACCACCUGCAUCCUGCGGUGGGUGUGCGCCACCGCAGACUGCAAGCUGUAGACUGCAGGC .((((((.(..((((.((...............(((((((.....)))).))).............((((..((((((((((....).)))))))))..)))))).))))..).)))))) ( -47.40) >DroYak_CAF1 1851 120 - 1 UGCCUGUUGCAUACAACUGCUGCAUUUGAUAACGCGCAGCAACAGGCUGACACUGACCACACCACCUGCAUCCUGCGGUAGGUGUGCGCCACCGCAGACUGCAAGCUGUACACUGCAGUC .(((((((((.......(((.((..........))))))))))))))...................((((..(((((((.(((....))))))))))..)))).(((((.....))))). ( -43.81) >consensus UGCCUGUUGCAUACAACUCCUGCAUUUGAUAACGCGCAGCAACAGGCUGACACUGACCACACCACCUGCAUCCUGCGGUAGGUGUGCGCCACCGCAGACUGCAAGCUGUAGACUGCAGGC .(((((((((..........(((............)))))))))))).................((((((..(((((((.(((....)))))))))).((((.....))))..)))))). (-42.92 = -43.32 + 0.40)

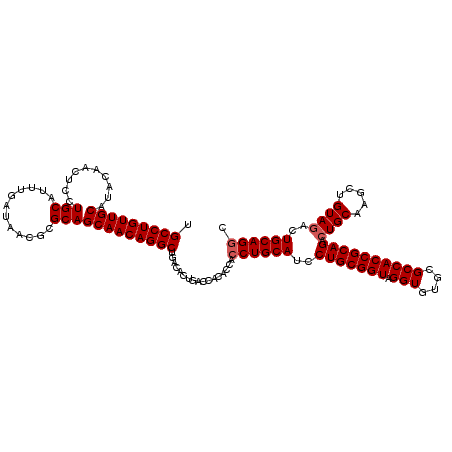
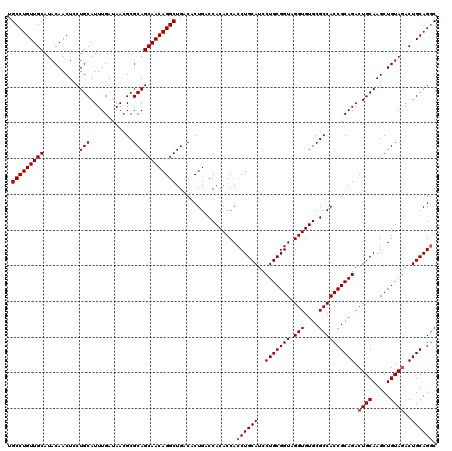
| Location | 1,666,572 – 1,666,687 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.21 |
| Mean single sequence MFE | -46.30 |
| Consensus MFE | -36.93 |
| Energy contribution | -37.33 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.16 |
| Structure conservation index | 0.80 |
| SVM decision value | 1.11 |
| SVM RNA-class probability | 0.916775 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1666572 115 + 22224390 UACCGCAGGAUGCAGGUGGUGUGGUCAGUGUCAGCCUGUUGCUGCGCGUUAUCAAAUGCAGGAGUUGUAUGCAACAGGCAGGAGGCCCACCGAC---CAAUACAUGCGCAU--CUCAUGC ....(((((((((..(((...(((((.(((...((((.(((((..(((((....)))))....((((....)))).))))).)))).))).)))---))...)))..))))--))..))) ( -46.60) >DroSec_CAF1 884 115 + 1 UACCGCAGGAUGCAGGUGGUGUGGUCAGUGUCAGCCUGUUGCUGCGCGUUAUCAAAUGCAGGAGUUGUAUGCAACAGGCAGGAGGCCCACCGAC---CAAUACAUGCGCAU--CUCAUGC ....(((((((((..(((...(((((.(((...((((.(((((..(((((....)))))....((((....)))).))))).)))).))).)))---))...)))..))))--))..))) ( -46.60) >DroSim_CAF1 1764 115 + 1 UACCGCAGGAUGCAGGUGGUGUGGUCAGUGUCAGCCUGUUGCUGCGCGUUAUCAAAUGCAGGAGUUGUAUGCAACAGGCAGGAGGCCCACCGAC---CAAUACAUGCGCAU--CUCAUGC ....(((((((((..(((...(((((.(((...((((.(((((..(((((....)))))....((((....)))).))))).)))).))).)))---))...)))..))))--))..))) ( -46.60) >DroEre_CAF1 691 115 + 1 CACCGCAGGAUGCAGGUGGUGUGGUCAGCGUCAGCCUGUUGCUGCGCGUUAUCAAAUGCAGGAGUUGUAUGCAACAGGCAGGAGGCCCACCGAC---CCAUACAUGCGCAC--CUCACGC ....(.(((.((((.((((..((((..((.((.(((((((((((((((((....))))).......))).)))))))))..)).))..))))..---))))...))))..)--))).... ( -43.11) >DroYak_CAF1 1891 120 + 1 UACCGCAGGAUGCAGGUGGUGUGGUCAGUGUCAGCCUGUUGCUGCGCGUUAUCAAAUGCAGCAGUUGUAUGCAACAGGCAGGAGGCCCACCGACCACCAAUACACACGCAUGUGUCAUGC ....(((.((((((((((((((((((.(((...((((.((((((((((((....))))).)).((((....)))).))))).)))).))).))))))....)).))).).)))))).))) ( -48.60) >consensus UACCGCAGGAUGCAGGUGGUGUGGUCAGUGUCAGCCUGUUGCUGCGCGUUAUCAAAUGCAGGAGUUGUAUGCAACAGGCAGGAGGCCCACCGAC___CAAUACAUGCGCAU__CUCAUGC .(((((.....)).)))((((.((((....((.(((((((((((((((((....))))).......))).))))))))).)).))))))))................((((.....)))) (-36.93 = -37.33 + 0.40)

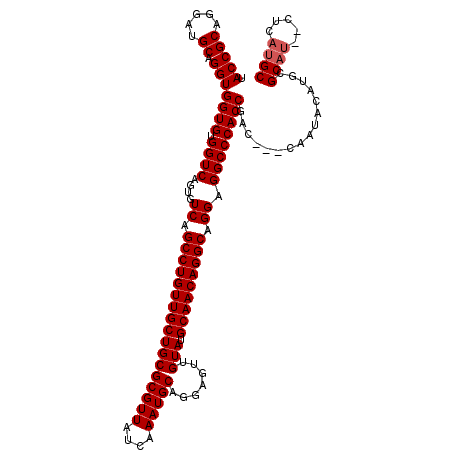
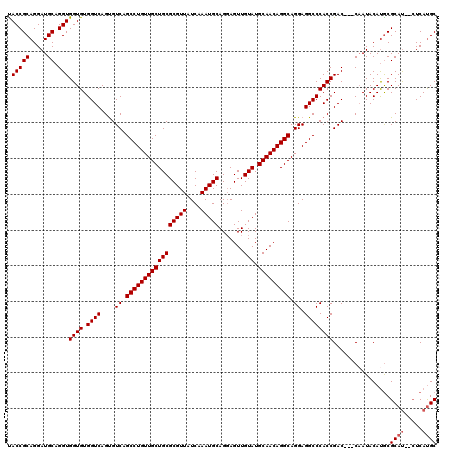
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:49:02 2006