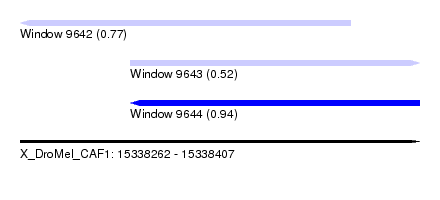
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 15,338,262 – 15,338,407 |
| Length | 145 |
| Max. P | 0.944375 |

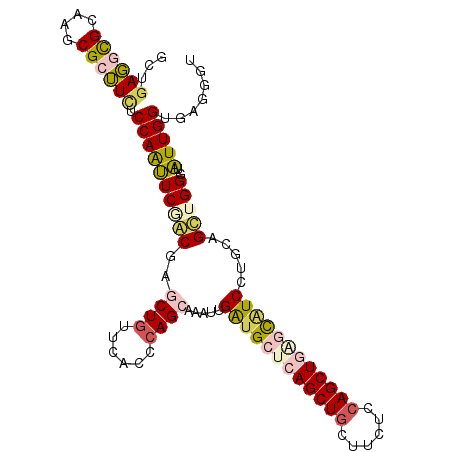
| Location | 15,338,262 – 15,338,382 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.22 |
| Mean single sequence MFE | -50.50 |
| Consensus MFE | -28.01 |
| Energy contribution | -29.68 |
| Covariance contribution | 1.68 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.23 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.52 |
| SVM RNA-class probability | 0.768976 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15338262 120 - 22224390 UCGACGAGCUGUUCACCCAGCAUAUUGAUGCUCAGCUGCUUUUCCAGCUGAGCAUCCUGCAGUUGGGUAUUGGCGAGGGUCAGCUCGGAGAUUUGUGAUUCUAGGGAGACGAGACGAUCG ((..((((((....(((((((.....((((((((((((......)))))))))))).....)))))))...(((....)))))))))..)).....((((((..(....).)))..))). ( -50.90) >DroPse_CAF1 26344 120 - 1 UCGGCCAGCUGAUCGCCCAGAAGGCUGGAGGAGAGCUCCUUCUCCAGCUGGGCGUCCUGCAGCUGGCUGUUGGCCAGGCUGAGCUCCGCGAUGCGCGUCUCGAGGGCCACGAGCUUGUCG .((((((((((..((((((....((((((((((.....)))))))))))))))).....))))))))))..(((.(((((..((((((.(((....))).)).))))....)))))))). ( -64.20) >DroSec_CAF1 42869 120 - 1 UCUUCGAGCUGUUCACUCAGCAUAUUGAUGCAGAGCUGCUUCUCCAGCUGAGUAUCCUCCAGCUGGGUAUUGGUGAGGGUCAGCUCAGCGAUUUGUGAUUCUAGAGAGACGAGGCGAUCG .....((((((..(.((((.((....((.(((....))).))((((((((((....)).))))))))...)).)))))..))))))..(((((..((.(((....))).))....))))) ( -39.50) >DroEre_CAF1 18594 120 - 1 UCGACGAGCUGUUCACCGAGCAAAUUGAUGCUCAGCUGCUUCUCCAGCUGAGCAUCCUGCAGUUGGGUAUUGGUAAGGGACAGCUCCGAGAUUUGUGAUUCCAGGGAGACGAGACGAUCG (((..((((((((((((.(((.....((((((((((((......)))))))))))).....))).))).........))))))))))))((((..((.(((....))).))....)))). ( -45.80) >DroYak_CAF1 19094 120 - 1 UCAACAAGCUGUUCACCCAGCAAGUUGAUGCUCAGCUGCUUCUCCAGCUGAGCAUCCUGCAGUUGGGUAUUGGUGAGUGUCAGCUCCGAGAUUUGUGAUUCUAGGGAGACGAGACGAUCG .......((((...(((((((..((.((((((((((((......))))))))))))..)).)))))))..))))((.((((.(((((.(((........)))..)))).)..)))).)). ( -47.60) >DroAna_CAF1 41588 120 - 1 UCCGCCAGCUGCUCGCCCAGGAGGUUGAUGCUCAGCUGCUUCUCCAGCUGGCCGUCCUGGAGCUGGCUGUUGGUCAACGACAGCUCGGAGAUUUGGGCCUCCAGGGAGACGAGGCGAUCG ..((((...((.((.(((.((((((.((((..((((((......))))))..))))(..((.((((((((((.....))))))).)))...))..))))))).))).)))).)))).... ( -55.00) >consensus UCGACGAGCUGUUCACCCAGCAAAUUGAUGCUCAGCUGCUUCUCCAGCUGAGCAUCCUGCAGCUGGGUAUUGGUGAGGGUCAGCUCCGAGAUUUGUGAUUCUAGGGAGACGAGACGAUCG ......(((((((((((((((.....((((((((((((......)))))))))))).....)))))))........))).)))))....((((..((.(((....))).))....)))). (-28.01 = -29.68 + 1.68)

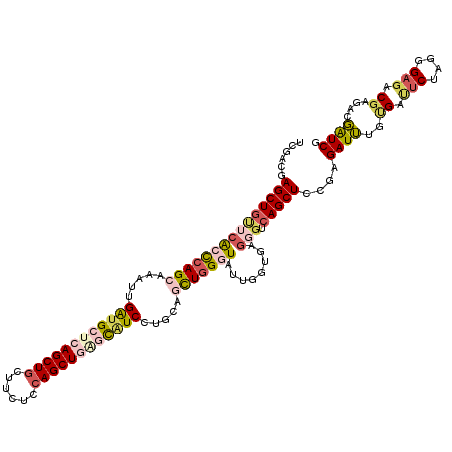
| Location | 15,338,302 – 15,338,407 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 80.38 |
| Mean single sequence MFE | -40.82 |
| Consensus MFE | -19.33 |
| Energy contribution | -20.25 |
| Covariance contribution | 0.92 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.29 |
| Structure conservation index | 0.47 |
| SVM decision value | -0.03 |
| SVM RNA-class probability | 0.520594 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15338302 105 + 22224390 ACCCUCGCCAAUACCCAACUGCAGGAUGCUCAGCUGGAAAAGCAGCUGAGCAUCAAUAUGCUGGGUGAACAGCUCGUCGAAUUGGAGAAGCGCUUGCGCCUCAGC .......((((((((((...(((.((((((((((((......))))))))))))....))))))))((.....)).....))))).((.(((....))).))... ( -42.00) >DroPse_CAF1 26384 105 + 1 AGCCUGGCCAACAGCCAGCUGCAGGACGCCCAGCUGGAGAAGGAGCUCUCCUCCAGCCUUCUGGGCGAUCAGCUGGCCGAGCUGGAGACCCGCCUGCGCAUCAGC .(((((((.....)))))..(((((.(((((((((((((.((.....)).)))))))....)))))).((((((.....)))))).......)))))))...... ( -49.00) >DroSec_CAF1 42909 105 + 1 ACCCUCACCAAUACCCAGCUGGAGGAUACUCAGCUGGAGAAGCAGCUCUGCAUCAAUAUGCUGAGUGAACAGCUCGAAGAAUUGGAAAAGCGCUUGCGCCUCAGC .......(((((..(((((((.((....))))))))).((.((.((((.((((....)))).)))).....)))).....)))))....(((....)))...... ( -32.50) >DroEre_CAF1 18634 105 + 1 UCCCUUACCAAUACCCAACUGCAGGAUGCUCAGCUGGAGAAGCAGCUGAGCAUCAAUUUGCUCGGUGAACAGCUCGUCGAAUUGGAGAAGCGCCUGCGACUCAGC .......(((((........(((.((((((((((((......))))))))))))....)))(((..((.....))..)))))))).((..((....))..))... ( -37.80) >DroYak_CAF1 19134 105 + 1 ACACUCACCAAUACCCAACUGCAGGAUGCUCAGCUGGAGAAGCAGCUGAGCAUCAACUUGCUGGGUGAACAGCUUGUUGAAUUGGAGAAGCGCUUGCGCCUCAGC .......((((((((((...(((.((((((((((((......))))))))))))....))))))))...(((....))).))))).((.(((....))).))... ( -42.40) >DroAna_CAF1 41628 105 + 1 UCGUUGACCAACAGCCAGCUCCAGGACGGCCAGCUGGAGAAGCAGCUGAGCAUCAACCUCCUGGGCGAGCAGCUGGCGGAGCUGGAGAAGCGCCUCCACCUGAGC ..(((....))).(((((((((((((..((((((((......)))))).)).......))))))(....))))))))..((.(((((......))))).)).... ( -41.20) >consensus ACCCUCACCAAUACCCAACUGCAGGAUGCUCAGCUGGAGAAGCAGCUGAGCAUCAACAUGCUGGGUGAACAGCUCGUCGAAUUGGAGAAGCGCCUGCGCCUCAGC ..................(((.(((((((.((((((......)))))).)))))....(((.(((((.....(..((...))..).....)))))))).))))). (-19.33 = -20.25 + 0.92)

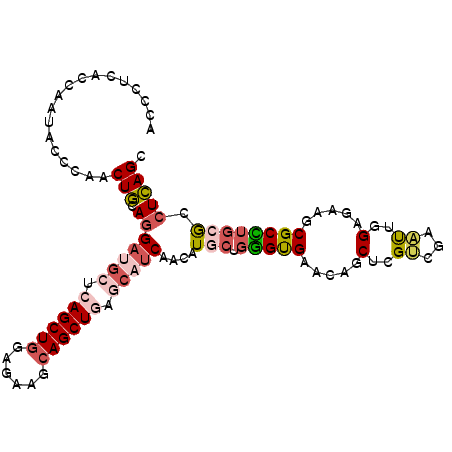

| Location | 15,338,302 – 15,338,407 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 80.38 |
| Mean single sequence MFE | -45.60 |
| Consensus MFE | -31.13 |
| Energy contribution | -31.88 |
| Covariance contribution | 0.76 |
| Combinations/Pair | 1.34 |
| Mean z-score | -2.70 |
| Structure conservation index | 0.68 |
| SVM decision value | 1.34 |
| SVM RNA-class probability | 0.944375 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15338302 105 - 22224390 GCUGAGGCGCAAGCGCUUCUCCAAUUCGACGAGCUGUUCACCCAGCAUAUUGAUGCUCAGCUGCUUUUCCAGCUGAGCAUCCUGCAGUUGGGUAUUGGCGAGGGU ((..(.((....))((((.((......)).)))).....(((((((.....((((((((((((......)))))))))))).....))))))).)..))...... ( -46.00) >DroPse_CAF1 26384 105 - 1 GCUGAUGCGCAGGCGGGUCUCCAGCUCGGCCAGCUGAUCGCCCAGAAGGCUGGAGGAGAGCUCCUUCUCCAGCUGGGCGUCCUGCAGCUGGCUGUUGGCCAGGCU ((((((.((....)).))).(((((..(((((((((..((((((....((((((((((.....)))))))))))))))).....))))))))))))))...))). ( -58.80) >DroSec_CAF1 42909 105 - 1 GCUGAGGCGCAAGCGCUUUUCCAAUUCUUCGAGCUGUUCACUCAGCAUAUUGAUGCAGAGCUGCUUCUCCAGCUGAGUAUCCUCCAGCUGGGUAUUGGUGAGGGU ((((((((....))((((............))))......)))))).(((..((((((((....))))(((((((((....)).)))))))))))..)))..... ( -37.20) >DroEre_CAF1 18634 105 - 1 GCUGAGUCGCAGGCGCUUCUCCAAUUCGACGAGCUGUUCACCGAGCAAAUUGAUGCUCAGCUGCUUCUCCAGCUGAGCAUCCUGCAGUUGGGUAUUGGUAAGGGA (((........))).(((..((((((((((..(((((((...)))))....((((((((((((......))))))))))))..)).)))))..)))))..))).. ( -38.60) >DroYak_CAF1 19134 105 - 1 GCUGAGGCGCAAGCGCUUCUCCAAUUCAACAAGCUGUUCACCCAGCAAGUUGAUGCUCAGCUGCUUCUCCAGCUGAGCAUCCUGCAGUUGGGUAUUGGUGAGUGU (((((((((....)))))).((((((((((..((((......))))..((.((((((((((((......))))))))))))..)).)))))..)))))..))).. ( -46.00) >DroAna_CAF1 41628 105 - 1 GCUCAGGUGGAGGCGCUUCUCCAGCUCCGCCAGCUGCUCGCCCAGGAGGUUGAUGCUCAGCUGCUUCUCCAGCUGGCCGUCCUGGAGCUGGCUGUUGGUCAACGA ....((.((((((....)))))).))..((((((.(((.((((((((.(.....((.((((((......))))))))).)))))).)).))).))))))...... ( -47.00) >consensus GCUGAGGCGCAAGCGCUUCUCCAAUUCGACGAGCUGUUCACCCAGCAAAUUGAUGCUCAGCUGCUUCUCCAGCUGAGCAUCCUGCAGCUGGGUAUUGGUGAGGGU ...((((((....)))))).((((((((((..((((......)))).....((((((((((((......)))))))))))).....)))))..)))))....... (-31.13 = -31.88 + 0.76)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:00:09 2006