
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 15,170,215 – 15,170,361 |
| Length | 146 |
| Max. P | 0.948827 |

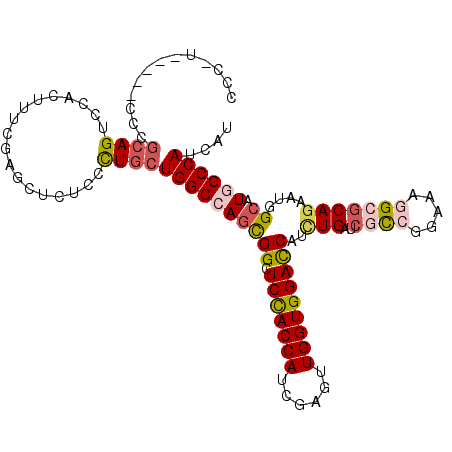
| Location | 15,170,215 – 15,170,321 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 82.18 |
| Mean single sequence MFE | -40.36 |
| Consensus MFE | -26.72 |
| Energy contribution | -27.55 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.10 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.41 |
| SVM RNA-class probability | 0.726457 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
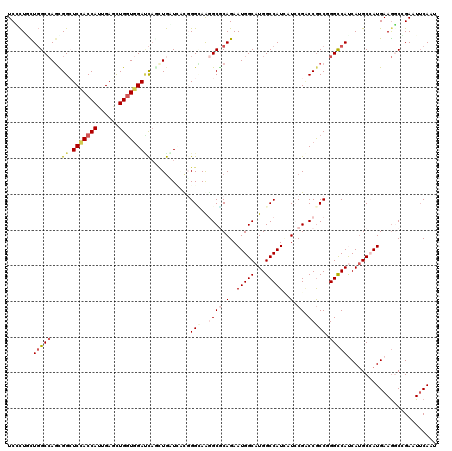
>X_DroMel_CAF1 15170215 106 + 22224390 CCC-A-----CCCGCAGUCCACAUUCGAGCUCUCCCUGCUGGCCAGCGGCUCCACCAUCGAAUUGGUGGACCAUCUGGUCGCCGGAAAGGCGCAGAAUGGCAUGGCCAUCAU ...-.-----...((((..................))))((((((((((.(((((((......))))))))).((((..((((.....))))))))...)).)))))).... ( -41.07) >DroSec_CAF1 751 106 + 1 CCC-A-----CCCGCAGUCCACUUUCGAGCUCUCCCUGCUGGCCAGCGGCUCUACCAUCGAGUUGGUGGACCAUCUGGUCACCGGAAAGGCGCAGAAUGGCAUGGCCAUCAU ...-.-----..(((..(((.......(((.......)))((((((.((.(((((((......)))))))))..))))))...)))...)))..((.((((...)))))).. ( -37.20) >DroEre_CAF1 780 107 + 1 UCC-U----CCUCGCAGUCCACAUUCGAGCUCUCCUUGCUGGCGAGUGGCUCCACCAUCGAGUUGGUGGACCACCUGGUCGCCGGGAAGGCGCAGAAUGGCAUGGCCAUAAU ...-.----....((......(((((..((...((((.(((((((((((.(((((((......)))))))))))....))))))).)))).)).))))).....))...... ( -45.10) >DroWil_CAF1 477 106 + 1 UCC-U-----AUUCCAGUCCACUUUUGAGCUGUCACUUUUGGCAAGUGGAUCCACCAUUGAGUUGGUGGAUCAAUUGAUCAUGGGCAAAGCGCAGAAUGGCAUGGCCAUUAU ...-.-----...(((..(((.(((((.(((....((..((((((...(((((((((......)))))))))..))).)))..))...))).))))))))..)))....... ( -35.60) >DroYak_CAF1 752 107 + 1 CCC-U----UCACGCAGUCCACCUUCGAGCUUUCCUUGCUGGCCAGCGGCUCCACCAUCGAGCUGGUGGACCAUCUGAUCGCCGGAAAGGCGCACAAUGGCAUGGCCAUCAU ...-.----....((.(((((((.....(((..((.....))..)))(((((.......)))))))))))).........(((.....)))))...(((((...)))))... ( -36.00) >DroAna_CAF1 633 112 + 1 UCCCUUCUCCCCCGCAGUCCACUUUUGAGCUAUCUCUGCUGGCCACUGGCUCCACCAUCGAGCUGGUGGACCAGCUGAUUGCUGGGAGCGCCCAGAAUGGCAUGGCCAUCAU ..((..(((((..((((((.......(((....))).(((((((((..((((.......))))..)))).))))).)))))).))))).(((......)))..))....... ( -47.20) >consensus CCC_U_____CCCGCAGUCCACUUUCGAGCUCUCCCUGCUGGCCAGCGGCUCCACCAUCGAGUUGGUGGACCAUCUGAUCGCCGGAAAGGCGCAGAAUGGCAUGGCCAUCAU .............((((..................))))((((((((((.(((((((......)))))))))..(((..((((.....)))))))....)).)))))).... (-26.72 = -27.55 + 0.84)


| Location | 15,170,241 – 15,170,361 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.56 |
| Mean single sequence MFE | -48.50 |
| Consensus MFE | -23.96 |
| Energy contribution | -25.02 |
| Covariance contribution | 1.06 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.53 |
| Structure conservation index | 0.49 |
| SVM decision value | 0.75 |
| SVM RNA-class probability | 0.841234 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15170241 120 + 22224390 UCCCUGCUGGCCAGCGGCUCCACCAUCGAAUUGGUGGACCAUCUGGUCGCCGGAAAGGCGCAGAAUGGCAUGGCCAUCAUCCGGCCACCGGGUCACCAUGCGAUGAAGGCCGAGUACAAC ...((((.((((((.((.(((((((......)))))))))..))))))(((.....)))))))....((.(((((.((((((((((....)))).....).))))).))))).))..... ( -53.90) >DroVir_CAF1 1345 120 + 1 UCGCUCUUGGCCAGCGGCUCGACCAUAGAGCUGGUGGAGCAGCUGCUGCUGGGUAAGGCGCAGAAUGGCAUGGCCAUAAUACGACCGCCGGGCCAUCAUGCGAUGAAGUCGGAAUUCAAU ..((.((((.((((((((((.((((......)))).))......)))))))).))))))(((..(((((.((((............)))).)))))..)))..((((.......)))).. ( -47.40) >DroPse_CAF1 962 120 + 1 UCCUUGCUUGCCAGCGGAUCCACCAUUGAUCUGGUGGAUUCCAUAAUUACGGGCAAGGCGCAGAAUGGCAUGGCCAUCAUCCGACCGCCAGGCCAUCAUGCCAUGAAGGCCGAAUUCAAU .(((((((((.....((((((((((......))))))))))........))))))))).(((..(((((.((((..((....))..)))).)))))..)))..((((.......)))).. ( -46.82) >DroWil_CAF1 503 120 + 1 UCACUUUUGGCAAGUGGAUCCACCAUUGAGUUGGUGGAUCAAUUGAUCAUGGGCAAAGCGCAGAAUGGCAUGGCCAUUAUACGUCCGCCUGGCCAUCAUGCCAUGAAAGCGGAAUUCAAU ((.(((((((((....(((((((((......)))))))))...((((..(((((...(((...((((((...))))))...)))..)))))...)))))))))..)))).))........ ( -42.70) >DroMoj_CAF1 737 120 + 1 UCGCUAUUGGCCAGUGGCUCGACCAUUGAGCUGAUCGAUCAGCUGCUGCUGGGUAAGGCACAGAAUGGCAUGGCCAUAAUCCGACCGCCUGGCCAUCAUGCCAUGAAGUCGGAGUUCAAU ..(((.(((.((((..((((((...))))(((((....))))).))..)))).))))))...(((((((...)))....((((((..(.((((......)))).)..))))))))))... ( -45.40) >DroAna_CAF1 665 120 + 1 UCUCUGCUGGCCACUGGCUCCACCAUCGAGCUGGUGGACCAGCUGAUUGCUGGGAGCGCCCAGAAUGGCAUGGCCAUCAUCCGGCCGCCCGGCCACCACGCCAUGAAGGCCGAGUUCAAU ..((.(((((((((..((((.......))))..)))).))))).))...(((((....)))))...(((..((((.......)))))))(((((..((.....))..)))))........ ( -54.80) >consensus UCCCUGCUGGCCAGCGGCUCCACCAUUGAGCUGGUGGAUCAGCUGAUCACGGGCAAGGCGCAGAAUGGCAUGGCCAUCAUCCGACCGCCGGGCCAUCAUGCCAUGAAGGCCGAAUUCAAU .......(((((...((.(((((((......)))))))))...........(((..((((.(..(((((...)))))..).)).))))).)))))......................... (-23.96 = -25.02 + 1.06)


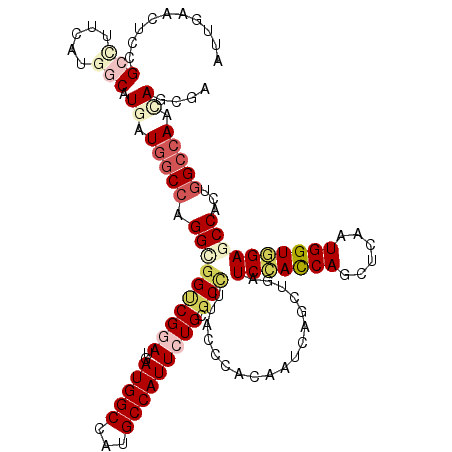
| Location | 15,170,241 – 15,170,361 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.56 |
| Mean single sequence MFE | -50.27 |
| Consensus MFE | -27.77 |
| Energy contribution | -29.55 |
| Covariance contribution | 1.78 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.79 |
| Structure conservation index | 0.55 |
| SVM decision value | 1.39 |
| SVM RNA-class probability | 0.948827 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15170241 120 - 22224390 GUUGUACUCGGCCUUCAUCGCAUGGUGACCCGGUGGCCGGAUGAUGGCCAUGCCAUUCUGCGCCUUUCCGGCGACCAGAUGGUCCACCAAUUCGAUGGUGGAGCCGCUGGCCAGCAGGGA ((((..(.((((((((((((..(((((..((((.(((((((..(((((...))))))))).)))...)))).((((....)))))))))...)))))).)).))))..)..))))..... ( -52.70) >DroVir_CAF1 1345 120 - 1 AUUGAAUUCCGACUUCAUCGCAUGAUGGCCCGGCGGUCGUAUUAUGGCCAUGCCAUUCUGCGCCUUACCCAGCAGCAGCUGCUCCACCAGCUCUAUGGUCGAGCCGCUGGCCAAGAGCGA ..((((.......))))((((....(((((((((((.((((..(((((...)))))..))))))....(((..((.(((((......))))))).)))....))))..)))))...)))) ( -43.20) >DroPse_CAF1 962 120 - 1 AUUGAAUUCGGCCUUCAUGGCAUGAUGGCCUGGCGGUCGGAUGAUGGCCAUGCCAUUCUGCGCCUUGCCCGUAAUUAUGGAAUCCACCAGAUCAAUGGUGGAUCCGCUGGCAAGCAAGGA ...........((((....(((.((((((.((((.(((....))).)))).)))))).)))((.(((((.((......(((.(((((((......)))))))))))).))))))))))). ( -52.30) >DroWil_CAF1 503 120 - 1 AUUGAAUUCCGCUUUCAUGGCAUGAUGGCCAGGCGGACGUAUAAUGGCCAUGCCAUUCUGCGCUUUGCCCAUGAUCAAUUGAUCCACCAACUCAAUGGUGGAUCCACUUGCCAAAAGUGA .........((((((..(((((((((.(...((((((((((.((((((...)))))).)))).))))))..).))))...(((((((((......)))))))))....))))))))))). ( -47.80) >DroMoj_CAF1 737 120 - 1 AUUGAACUCCGACUUCAUGGCAUGAUGGCCAGGCGGUCGGAUUAUGGCCAUGCCAUUCUGUGCCUUACCCAGCAGCAGCUGAUCGAUCAGCUCAAUGGUCGAGCCACUGGCCAAUAGCGA .......(((((((.(.((((......)))).).)))))))...((((((.........(((.(((..(((...(.((((((....)))))))..)))..))).)))))))))....... ( -42.40) >DroAna_CAF1 665 120 - 1 AUUGAACUCGGCCUUCAUGGCGUGGUGGCCGGGCGGCCGGAUGAUGGCCAUGCCAUUCUGGGCGCUCCCAGCAAUCAGCUGGUCCACCAGCUCGAUGGUGGAGCCAGUGGCCAGCAGAGA ......(((.(((.....))).((.((((((((((.(((((..(((((...)))))))))).)))))..........((((((((((((......))))))).)))))))))).))))). ( -63.20) >consensus AUUGAACUCCGCCUUCAUGGCAUGAUGGCCAGGCGGUCGGAUGAUGGCCAUGCCAUUCUGCGCCUUACCCACAAUCAGCUGAUCCACCAGCUCAAUGGUGGAGCCACUGGCCAACAGCGA ..........(((.....))).((.(((((.((((((((((..(((((...))))))))).)))..................(((((((......))))))))))...))))).)).... (-27.77 = -29.55 + 1.78)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:58:58 2006