
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 15,152,464 – 15,152,605 |
| Length | 141 |
| Max. P | 0.805614 |

| Location | 15,152,464 – 15,152,567 |
|---|---|
| Length | 103 |
| Sequences | 4 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 91.89 |
| Mean single sequence MFE | -22.70 |
| Consensus MFE | -18.33 |
| Energy contribution | -18.32 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.63 |
| SVM RNA-class probability | 0.805614 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
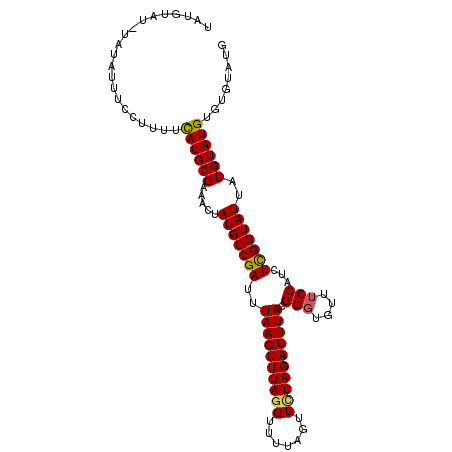
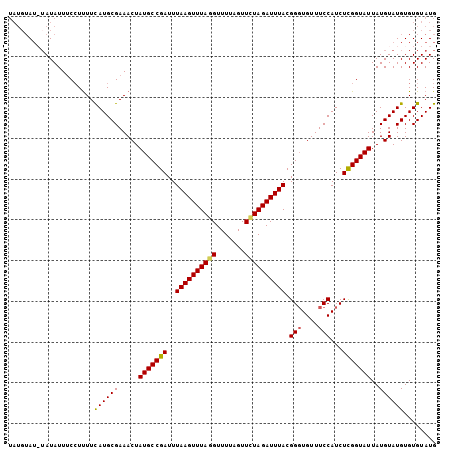
>X_DroMel_CAF1 15152464 103 - 22224390 UAUGUAU--AUAUUACCUUUUCAUGCGAAACUAUGCCGAUUUAAGUUUAGGUUUUAGUUUUAGAUUUACGGUUGUUUCCAUCUCGGUAUUAUGUAUGUGUGUAUG (((((((--(((.((((.....(((.(((((...((((...((((((((((.......)))))))))))))).))))))))...)))).))))))))))...... ( -26.60) >DroSec_CAF1 3406 105 - 1 UAUGUAUAUAUAUUUCCUUUUCAUGCGAAACUAUGCCGAUUUAAGUUUAGGUUUUAGUUCUAGAUUUACGGGUGUUUCCAUCUCGGUAUUAUGUAUGUGUGUAUG ((..(((((((((.....((((....))))..(((((((..((((((((((.......)))))))))).(((....)))...))))))).)))))))))..)).. ( -25.10) >DroSim_CAF1 3419 105 - 1 UAUGUAUAUAUAUUUCCUUUUCAUGCGAAACUAUGCCGAUUUAAGUUUAGGUUUUAGUUCUAGAUUUACGGGUGUUUCCAUCUCGGUAUUAUGUAUGUGUGUAUG ((..(((((((((.....((((....))))..(((((((..((((((((((.......)))))))))).(((....)))...))))))).)))))))))..)).. ( -25.10) >DroYak_CAF1 3328 98 - 1 -----A--UAUAUUUCAUCUUUAUGCGAAACUAUGCCGAUUUAAGUUUACGUUUUAGUUAUAGAUUUACGGGUGUUUCCAUCUUGGUAUUAUGUAUGUGUGUAUG -----(--(((((...(((...(((.(((((....(((...((((((((...........)))))))))))..))))))))...)))...))))))......... ( -14.00) >consensus UAUGUAU_UAUAUUUCCUUUUCAUGCGAAACUAUGCCGAUUUAAGUUUAGGUUUUAGUUCUAGAUUUACGGGUGUUUCCAUCUCGGUAUUAUGUAUGUGUGUAUG .....................((((((.....(((((((..((((((((((.......)))))))))).(((....)))...)))))))..))))))........ (-18.33 = -18.32 + -0.00)

| Location | 15,152,495 – 15,152,605 |
|---|---|
| Length | 110 |
| Sequences | 4 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 88.87 |
| Mean single sequence MFE | -24.07 |
| Consensus MFE | -17.66 |
| Energy contribution | -19.22 |
| Covariance contribution | 1.56 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.02 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.56 |
| SVM RNA-class probability | 0.782132 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
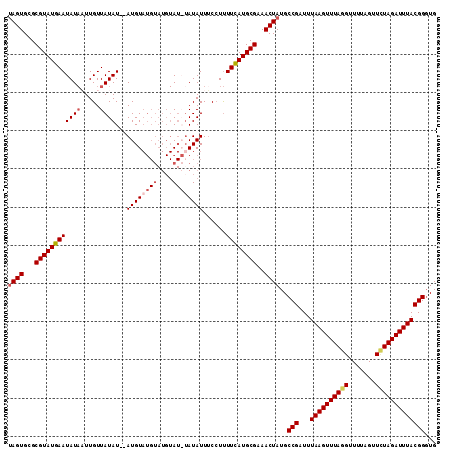
>X_DroMel_CAF1 15152495 110 - 22224390 UAGUGCGCGUAUGAAUAUAUUUGUUAUAUUUAUGUAUGUAUGUAU--AUAUUACCUUUUCAUGCGAAACUAUGCCGAUUUAAGUUUAGGUUUUAGUUUUAGAUUUACGGUUG ((((...((((((((((((.....))))..(((((((....))))--))).......))))))))..)))).((((...((((((((((.......)))))))))))))).. ( -23.50) >DroSec_CAF1 3437 110 - 1 UAGUGCGCGUAUGAAUAUAAUUGUUAUAU--AUGUAUGUAUGUAUAUAUAUUUCCUUUUCAUGCGAAACUAUGCCGAUUUAAGUUUAGGUUUUAGUUCUAGAUUUACGGGUG ((((...((((((((.......(..((((--((((((....))))))))))..)...))))))))..))))..(((...((((((((((.......)))))))))))))... ( -28.30) >DroSim_CAF1 3450 110 - 1 UAGUGCGCGUAUGAAUAUAAUUGUUAUAU--AUGUAUGUAUGUAUAUAUAUUUCCUUUUCAUGCGAAACUAUGCCGAUUUAAGUUUAGGUUUUAGUUCUAGAUUUACGGGUG ((((...((((((((.......(..((((--((((((....))))))))))..)...))))))))..))))..(((...((((((((((.......)))))))))))))... ( -28.30) >DroYak_CAF1 3359 98 - 1 AAGUGCGCGUAUGAAUAU--UUGUUAUAU--AUGU--------A--UAUAUUUCAUCUUUAUGCGAAACUAUGCCGAUUUAAGUUUACGUUUUAGUUAUAGAUUUACGGGUG .(((...((((((((...--.((..((((--(...--------.--)))))..))..))))))))..)))...(((...((((((((...........)))))))))))... ( -16.20) >consensus UAGUGCGCGUAUGAAUAUAAUUGUUAUAU__AUGUAUGUAUGUAU_UAUAUUUCCUUUUCAUGCGAAACUAUGCCGAUUUAAGUUUAGGUUUUAGUUCUAGAUUUACGGGUG ((((...((((((((((((.....))))...((((((((....))))))))......))))))))..))))..(((...((((((((((.......)))))))))))))... (-17.66 = -19.22 + 1.56)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:58:47 2006