
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 15,135,850 – 15,136,046 |
| Length | 196 |
| Max. P | 0.999792 |

| Location | 15,135,850 – 15,135,970 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.83 |
| Mean single sequence MFE | -31.54 |
| Consensus MFE | -29.34 |
| Energy contribution | -29.74 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.34 |
| Structure conservation index | 0.93 |
| SVM decision value | 0.52 |
| SVM RNA-class probability | 0.767448 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15135850 120 + 22224390 GAUAAAUCGAAUCUUAAUUGGAGGCGAAUUUAGCAUGCCUGAUUGGAUCGCAUAUAGCCGAGAUAGCUUGACGCCAUCUUAGGCAGCUAUGCUAAUUAAAUCGAUUUGACUUAGAUUAGC ..((((((((...((((((((.(((.......((...((.....))...)).....((((((((.((.....)).))))).))).)))...)))))))).))))))))............ ( -31.60) >DroSec_CAF1 47303 120 + 1 GAUAAAUCGAAUCUUAAUUGGAGGCGAAUCUUGCAUGCCUGAUUGGAUCGCAUAUAGCCGAGAUAGCUUGACGCCAUCUUAGGCAGCUAUGCUAAUUAAAUCGAUUUGACUUAGAUUAGC ..((((((((...((((((((.(((......(((...((.....))...)))....)))...((((((....(((......))))))))).)))))))).))))))))............ ( -32.30) >DroSim_CAF1 37998 120 + 1 GAUAAAUCGAAUCUUAAUUGGAGGCGAAUUUAGCAUGCCUGAUUGGAUCGCAUAUAGCCGAGAUAGCUUGACGCCAUCUUAGGCAGCUAUGCUAAUUAAAUCGAUUUGACUUAGAUUAGC ..((((((((...((((((((.(((.......((...((.....))...)).....((((((((.((.....)).))))).))).)))...)))))))).))))))))............ ( -31.60) >DroEre_CAF1 54155 120 + 1 GAUAAAUCGAAUCUUAAUUGGAGGCGAAUUUAGCAUGCCUGAUUGGAUCGCAUAUUGCCGAGAUAGCUUGACGCCAUCUUAGCCAGCUAUGCUAAUUAAAUCGAUUUGGCUUAGAUUAGC ..((((((((...((((((((.(((.......((...((.....))...)).....((..((((.((.....)).))))..))..)))...)))))))).))))))))(((......))) ( -30.10) >DroYak_CAF1 54887 120 + 1 GAUAAAUCGAAUCUUAAUUGGAGGCGAAUUUAGCAUGCCUGAUUGGAUCGCAUAUAGCCGAGAUAGCUUGACGCCAUCUUAGGCAGCUAUGCUAAUCAAAUCGAUUUGGCUUAGAUUAGC ......(((..(((.....)))..)))...((((.(((((((.(((...((.....))((((....))))...)))..))))))))))).(((((((((..(.....)..)).))))))) ( -32.10) >consensus GAUAAAUCGAAUCUUAAUUGGAGGCGAAUUUAGCAUGCCUGAUUGGAUCGCAUAUAGCCGAGAUAGCUUGACGCCAUCUUAGGCAGCUAUGCUAAUUAAAUCGAUUUGACUUAGAUUAGC ..((((((((...((((((((.(((.......((...((.....))...)).....((((((((.((.....)).))))).))).)))...)))))))).))))))))............ (-29.34 = -29.74 + 0.40)



| Location | 15,135,850 – 15,135,970 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.83 |
| Mean single sequence MFE | -34.26 |
| Consensus MFE | -31.58 |
| Energy contribution | -31.38 |
| Covariance contribution | -0.20 |
| Combinations/Pair | 1.06 |
| Mean z-score | -3.39 |
| Structure conservation index | 0.92 |
| SVM decision value | 4.09 |
| SVM RNA-class probability | 0.999792 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15135850 120 - 22224390 GCUAAUCUAAGUCAAAUCGAUUUAAUUAGCAUAGCUGCCUAAGAUGGCGUCAAGCUAUCUCGGCUAUAUGCGAUCCAAUCAGGCAUGCUAAAUUCGCCUCCAAUUAAGAUUCGAUUUAUC .......((((((.((((((((..(((.(((((...(((..(((((((.....))))))).)))..))))))))..))))((((...........))))........)))).)))))).. ( -33.90) >DroSec_CAF1 47303 120 - 1 GCUAAUCUAAGUCAAAUCGAUUUAAUUAGCAUAGCUGCCUAAGAUGGCGUCAAGCUAUCUCGGCUAUAUGCGAUCCAAUCAGGCAUGCAAGAUUCGCCUCCAAUUAAGAUUCGAUUUAUC .......((((((.((((((((..(((.(((((...(((..(((((((.....))))))).)))..))))))))..))))((((...........))))........)))).)))))).. ( -33.90) >DroSim_CAF1 37998 120 - 1 GCUAAUCUAAGUCAAAUCGAUUUAAUUAGCAUAGCUGCCUAAGAUGGCGUCAAGCUAUCUCGGCUAUAUGCGAUCCAAUCAGGCAUGCUAAAUUCGCCUCCAAUUAAGAUUCGAUUUAUC .......((((((.((((((((..(((.(((((...(((..(((((((.....))))))).)))..))))))))..))))((((...........))))........)))).)))))).. ( -33.90) >DroEre_CAF1 54155 120 - 1 GCUAAUCUAAGCCAAAUCGAUUUAAUUAGCAUAGCUGGCUAAGAUGGCGUCAAGCUAUCUCGGCAAUAUGCGAUCCAAUCAGGCAUGCUAAAUUCGCCUCCAAUUAAGAUUCGAUUUAUC (((......))).((((((((((((((.(((((....((..(((((((.....)))))))..))..))))).........((((...........))))..)))))))..)))))))... ( -34.90) >DroYak_CAF1 54887 120 - 1 GCUAAUCUAAGCCAAAUCGAUUUGAUUAGCAUAGCUGCCUAAGAUGGCGUCAAGCUAUCUCGGCUAUAUGCGAUCCAAUCAGGCAUGCUAAAUUCGCCUCCAAUUAAGAUUCGAUUUAUC (((......))).(((((((((.((((.(((((...(((..(((((((.....))))))).)))..))))))))).))))((((...........)))).............)))))... ( -34.70) >consensus GCUAAUCUAAGUCAAAUCGAUUUAAUUAGCAUAGCUGCCUAAGAUGGCGUCAAGCUAUCUCGGCUAUAUGCGAUCCAAUCAGGCAUGCUAAAUUCGCCUCCAAUUAAGAUUCGAUUUAUC (((......))).((((((((((((((.(((((...(((..(((((((.....))))))).)))..))))).........((((...........))))..)))))))..)))))))... (-31.58 = -31.38 + -0.20)

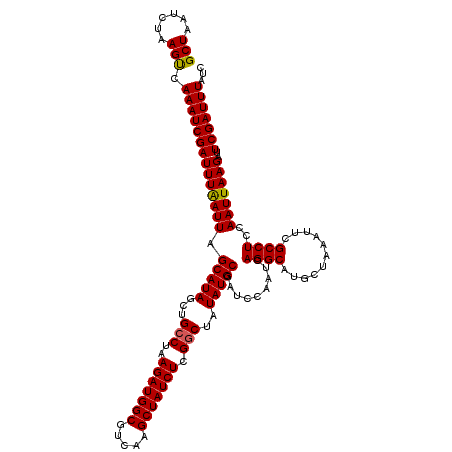

| Location | 15,135,890 – 15,136,010 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.67 |
| Mean single sequence MFE | -35.08 |
| Consensus MFE | -31.42 |
| Energy contribution | -31.42 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.24 |
| Structure conservation index | 0.90 |
| SVM decision value | 0.26 |
| SVM RNA-class probability | 0.658281 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15135890 120 + 22224390 GAUUGGAUCGCAUAUAGCCGAGAUAGCUUGACGCCAUCUUAGGCAGCUAUGCUAAUUAAAUCGAUUUGACUUAGAUUAGCCACGCUUGCCGCGGUUCACAUGGCGUAUGUGCAAUAAACG .........(((((((((((((((.((.....)).))))).((((((...(((((((.((((.....)).)).)))))))...)).))))...........))).)))))))........ ( -34.80) >DroSec_CAF1 47343 120 + 1 GAUUGGAUCGCAUAUAGCCGAGAUAGCUUGACGCCAUCUUAGGCAGCUAUGCUAAUUAAAUCGAUUUGACUUAGAUUAGCCACGCCAGCAGCGGUUCACAUGGCGUAUGUGCAAUAAACG .........(((((((((((((((.((.....)).))))).))).((((((((((((.((((.....)).)).)))))))..(((.....)))......))))).)))))))........ ( -33.10) >DroSim_CAF1 38038 120 + 1 GAUUGGAUCGCAUAUAGCCGAGAUAGCUUGACGCCAUCUUAGGCAGCUAUGCUAAUUAAAUCGAUUUGACUUAGAUUAGCCACGCCAGCCGCGGUUCACAUGGCGUAUGUGCAAUAAACG .........(((((((((((((((.((.....)).))))).))).(((((((((((((((....))))).))))...((((.((.....)).))))..)))))).)))))))........ ( -33.10) >DroEre_CAF1 54195 119 + 1 GAUUGGAUCGCAUAUUGCCGAGAUAGCUUGACGCCAUCUUAGCCAGCUAUGCUAAUUAAAUCGAUUUGGCUUAGAUUAGCCACGC-UGCUGCGGUUCACAUGGCGUAUGUGCAAUAGCCU ...(((((((((....((..((((.((.....)).))))..))((((...(((((((((..(.....)..)).)))))))...))-)).)))))))))...(((.(((.....)))))). ( -35.50) >DroYak_CAF1 54927 120 + 1 GAUUGGAUCGCAUAUAGCCGAGAUAGCUUGACGCCAUCUUAGGCAGCUAUGCUAAUCAAAUCGAUUUGGCUUAGAUUAGCCACGCUUGCUGCGGUUCACAUGGCGUAUGUGCAAUAAACG .........(((((((((((((((.((.....)).))))).))).((((((..((((.........(((((......))))).((.....))))))..)))))).)))))))........ ( -38.90) >consensus GAUUGGAUCGCAUAUAGCCGAGAUAGCUUGACGCCAUCUUAGGCAGCUAUGCUAAUUAAAUCGAUUUGACUUAGAUUAGCCACGCCUGCCGCGGUUCACAUGGCGUAUGUGCAAUAAACG .........(((((((((((((((.((.....)).))))).))).((((((((((((.((((.....)).)).)))))))..(((.....)))......))))).)))))))........ (-31.42 = -31.42 + -0.00)


| Location | 15,135,890 – 15,136,010 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.67 |
| Mean single sequence MFE | -40.28 |
| Consensus MFE | -34.90 |
| Energy contribution | -35.38 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.05 |
| Mean z-score | -3.28 |
| Structure conservation index | 0.87 |
| SVM decision value | 3.74 |
| SVM RNA-class probability | 0.999574 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15135890 120 - 22224390 CGUUUAUUGCACAUACGCCAUGUGAACCGCGGCAAGCGUGGCUAAUCUAAGUCAAAUCGAUUUAAUUAGCAUAGCUGCCUAAGAUGGCGUCAAGCUAUCUCGGCUAUAUGCGAUCCAAUC .....((((((.(((.(((...........((((.((...((((((.((((((.....))))))))))))...))))))..(((((((.....))))))).)))))).))))))...... ( -41.40) >DroSec_CAF1 47343 120 - 1 CGUUUAUUGCACAUACGCCAUGUGAACCGCUGCUGGCGUGGCUAAUCUAAGUCAAAUCGAUUUAAUUAGCAUAGCUGCCUAAGAUGGCGUCAAGCUAUCUCGGCUAUAUGCGAUCCAAUC .....((((((..(((((((.((........)))))))))((((((.((((((.....))))))))))))(((((((....(((((((.....)))))))))))))).))))))...... ( -41.90) >DroSim_CAF1 38038 120 - 1 CGUUUAUUGCACAUACGCCAUGUGAACCGCGGCUGGCGUGGCUAAUCUAAGUCAAAUCGAUUUAAUUAGCAUAGCUGCCUAAGAUGGCGUCAAGCUAUCUCGGCUAUAUGCGAUCCAAUC .....((((((..(((((((((((...))))..)))))))((((((.((((((.....))))))))))))(((((((....(((((((.....)))))))))))))).))))))...... ( -41.80) >DroEre_CAF1 54195 119 - 1 AGGCUAUUGCACAUACGCCAUGUGAACCGCAGCA-GCGUGGCUAAUCUAAGCCAAAUCGAUUUAAUUAGCAUAGCUGGCUAAGAUGGCGUCAAGCUAUCUCGGCAAUAUGCGAUCCAAUC .((......(((((.....)))))...((((.((-((...((((((.(((..(.....)..)))))))))...))))((..(((((((.....)))))))..))....))))..)).... ( -37.70) >DroYak_CAF1 54927 120 - 1 CGUUUAUUGCACAUACGCCAUGUGAACCGCAGCAAGCGUGGCUAAUCUAAGCCAAAUCGAUUUGAUUAGCAUAGCUGCCUAAGAUGGCGUCAAGCUAUCUCGGCUAUAUGCGAUCCAAUC .((((((.((......))...))))))(((.....)))(((((......)))))....((((.((((.(((((...(((..(((((((.....))))))).)))..))))))))).)))) ( -38.60) >consensus CGUUUAUUGCACAUACGCCAUGUGAACCGCAGCAAGCGUGGCUAAUCUAAGUCAAAUCGAUUUAAUUAGCAUAGCUGCCUAAGAUGGCGUCAAGCUAUCUCGGCUAUAUGCGAUCCAAUC .....((((((..(((((..((((...))))....)))))((((((.((((((.....))))))))))))(((((((....(((((((.....)))))))))))))).))))))...... (-34.90 = -35.38 + 0.48)

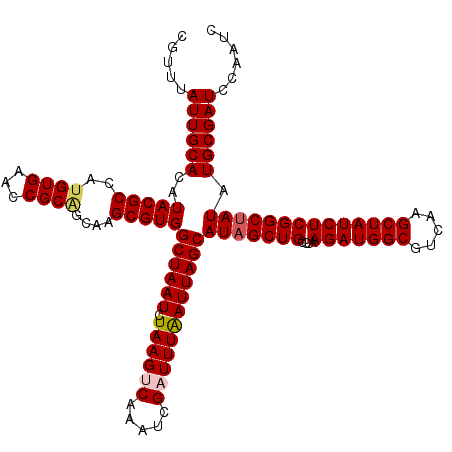

| Location | 15,135,930 – 15,136,046 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.90 |
| Mean single sequence MFE | -41.85 |
| Consensus MFE | -35.43 |
| Energy contribution | -36.03 |
| Covariance contribution | 0.60 |
| Combinations/Pair | 1.06 |
| Mean z-score | -3.06 |
| Structure conservation index | 0.85 |
| SVM decision value | 3.64 |
| SVM RNA-class probability | 0.999482 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15135930 116 + 22224390 AGGCAGCUAUGCUAAUUAAAUCGAUUUGACUUAGAUUAGCCACGCUUGCCGCGGUUCACAUGGCGUAUGUGCAAUAAACGCGGCUCACCAUAACGCAUACGCCGUGUGGCCAGGAA---- .((((((...(((((((.((((.....)).)).)))))))...)).))))..(((.((((((((((((((((.......))((....)).....))))))))))))))))).....---- ( -45.30) >DroSec_CAF1 47383 116 + 1 AGGCAGCUAUGCUAAUUAAAUCGAUUUGACUUAGAUUAGCCACGCCAGCAGCGGUUCACAUGGCGUAUGUGCAAUAAACGCGGCUCAACAUAACGCAUACGCCGUGUGGCCAGGCA---- .(((.((((..(((((((((....))))).))))..))))...))).((...(((.((((((((((((((((.......))(......).....)))))))))))))))))..)).---- ( -42.30) >DroSim_CAF1 38078 116 + 1 AGGCAGCUAUGCUAAUUAAAUCGAUUUGACUUAGAUUAGCCACGCCAGCCGCGGUUCACAUGGCGUAUGUGCAAUAAACGCGGCUCAACAUAACGCAUACGCCGUGUGGCCAGGCA---- .(((.((((..(((((((((....))))).))))..))))...))).(((..(((.((((((((((((((((.......))(......).....))))))))))))))))).))).---- ( -46.20) >DroEre_CAF1 54235 119 + 1 AGCCAGCUAUGCUAAUUAAAUCGAUUUGGCUUAGAUUAGCCACGC-UGCUGCGGUUCACAUGGCGUAUGUGCAAUAGCCUCGAUACAACAUAACGCAUACGCCUAGUGGCCAGGAACGCA .((((((...((.((((.....))))(((((......))))).))-.)))).(((.(((..((((((((((......................))))))))))..))))))......)). ( -35.25) >DroYak_CAF1 54967 116 + 1 AGGCAGCUAUGCUAAUCAAAUCGAUUUGGCUUAGAUUAGCCACGCUUGCUGCGGUUCACAUGGCGUAUGUGCAAUAAACGAGGCACACCAUAACGCAUACGCCCAGUGGCCAGGAA---- ..(((((...((.((((.....))))(((((......))))).))..)))))(((.(((..(((((((((((.......).((....))....))))))))))..)))))).....---- ( -40.20) >consensus AGGCAGCUAUGCUAAUUAAAUCGAUUUGACUUAGAUUAGCCACGCCUGCCGCGGUUCACAUGGCGUAUGUGCAAUAAACGCGGCUCAACAUAACGCAUACGCCGUGUGGCCAGGAA____ .(((.((((..(((((((((....))))).))))..))))..(((.....)))...(((((((((((((((......................))))))))))))))))))......... (-35.43 = -36.03 + 0.60)

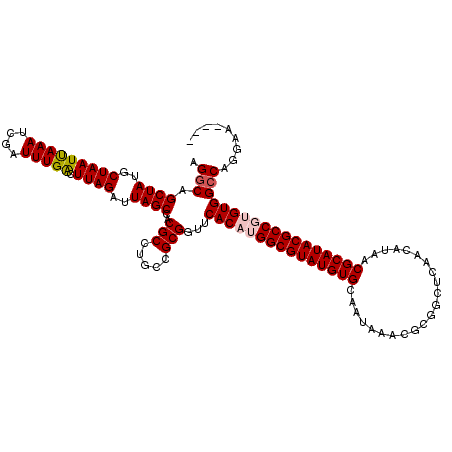
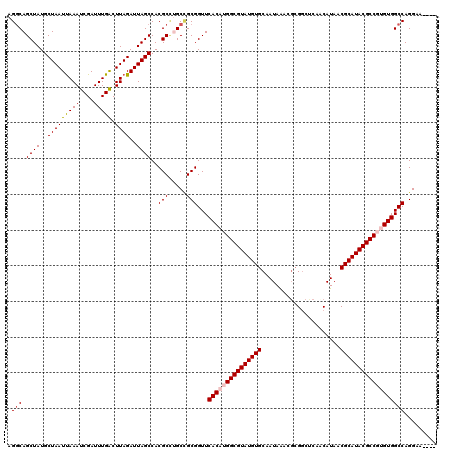
| Location | 15,135,930 – 15,136,046 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.90 |
| Mean single sequence MFE | -40.48 |
| Consensus MFE | -33.08 |
| Energy contribution | -33.96 |
| Covariance contribution | 0.88 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.24 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.94 |
| SVM RNA-class probability | 0.983354 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
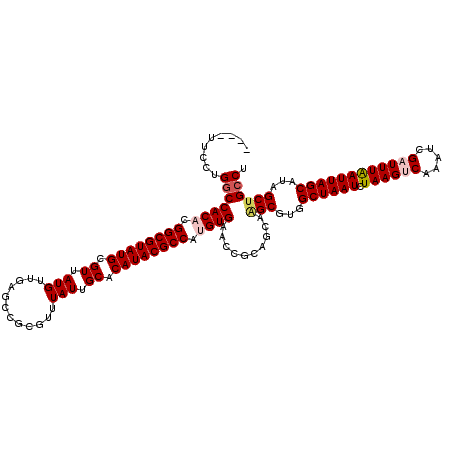
>X_DroMel_CAF1 15135930 116 - 22224390 ----UUCCUGGCCACACGGCGUAUGCGUUAUGGUGAGCCGCGUUUAUUGCACAUACGCCAUGUGAACCGCGGCAAGCGUGGCUAAUCUAAGUCAAAUCGAUUUAAUUAGCAUAGCUGCCU ----.....((.((((.((((((((.((...(((((((...))))))))).)))))))).))))..))..((((.((...((((((.((((((.....))))))))))))...)))))). ( -44.70) >DroSec_CAF1 47383 116 - 1 ----UGCCUGGCCACACGGCGUAUGCGUUAUGUUGAGCCGCGUUUAUUGCACAUACGCCAUGUGAACCGCUGCUGGCGUGGCUAAUCUAAGUCAAAUCGAUUUAAUUAGCAUAGCUGCCU ----.((.(((.((((.((((((((.((.((((......)))).....)).)))))))).))))..)))..)).(((((.((((((.((((((.....))))))))))))...)).))). ( -40.20) >DroSim_CAF1 38078 116 - 1 ----UGCCUGGCCACACGGCGUAUGCGUUAUGUUGAGCCGCGUUUAUUGCACAUACGCCAUGUGAACCGCGGCUGGCGUGGCUAAUCUAAGUCAAAUCGAUUUAAUUAGCAUAGCUGCCU ----.((((((.((((.((((((((.((.((((......)))).....)).)))))))).))))..))).))).(((((.((((((.((((((.....))))))))))))...)).))). ( -44.80) >DroEre_CAF1 54235 119 - 1 UGCGUUCCUGGCCACUAGGCGUAUGCGUUAUGUUGUAUCGAGGCUAUUGCACAUACGCCAUGUGAACCGCAGCA-GCGUGGCUAAUCUAAGCCAAAUCGAUUUAAUUAGCAUAGCUGGCU ((((.....(..(((..((((((((.((...(((.......)))....)).))))))))..)))..))))).((-((...((((((.(((..(.....)..)))))))))...))))... ( -35.40) >DroYak_CAF1 54967 116 - 1 ----UUCCUGGCCACUGGGCGUAUGCGUUAUGGUGUGCCUCGUUUAUUGCACAUACGCCAUGUGAACCGCAGCAAGCGUGGCUAAUCUAAGCCAAAUCGAUUUGAUUAGCAUAGCUGCCU ----.....((.(((..((((((((.((.((((..........)))).)).))))))))..)))..))(((((..((.(((((......)))))((((.....)))).))...))))).. ( -37.30) >consensus ____UUCCUGGCCACACGGCGUAUGCGUUAUGUUGAGCCGCGUUUAUUGCACAUACGCCAUGUGAACCGCAGCAAGCGUGGCUAAUCUAAGUCAAAUCGAUUUAAUUAGCAUAGCUGCCU .........(((((((.((((((((.((.(((............))).)).)))))))).))))..........(((...((((((.((((((.....))))))))))))...)))))). (-33.08 = -33.96 + 0.88)

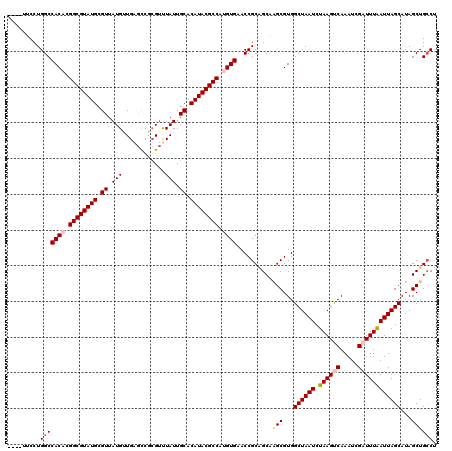
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:58:35 2006