| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 1,637,901 – 1,638,061 |
| Length | 160 |
| Max. P | 0.994822 |

| Location | 1,637,901 – 1,638,021 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.44 |
| Mean single sequence MFE | -30.22 |
| Consensus MFE | -26.32 |
| Energy contribution | -26.48 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.52 |
| Structure conservation index | 0.87 |
| SVM decision value | 0.37 |
| SVM RNA-class probability | 0.707984 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
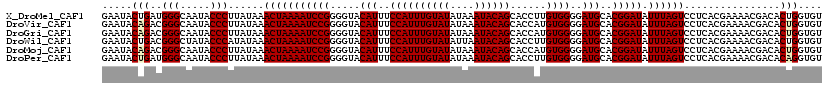
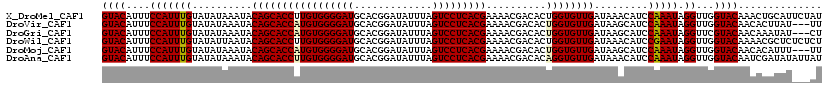
>X_DroMel_CAF1 1637901 120 + 22224390 GAAUACUGAUGGGCAAUACCCUUAUAAACUAAAAUCCGGGGUACAUUUCCAUUUGUAUAUAAAUACAGCACCUUGUGGGGAUGCACGGAUAUUUAGUCCUCACGAAAACGACACUGGUGU ..((((.((((((...((((((...............))))))....)))))).)))).........(((((((((((((((.............))))))))))..........))))) ( -30.78) >DroVir_CAF1 5178 120 + 1 GAAUACAGACGGGCAAUACCCUUAUAAACUAAAAUCCGGGGUACAUUUCCAUUUGUAUAUAAAUACAGCACCAUGUGGGGAUGCACGGAUAUUUAGUCCUCACGAAAACGACACUGGUGU ..(((((((.(((...((((((...............))))))....))).))))))).........(((((((((((((((.............)))))))))..........)))))) ( -30.58) >DroGri_CAF1 4053 120 + 1 GAAUACAGACGGGCAAUACCCUUAUAAACUAAAAUCCGGGGUACAUUUCCAUUUGUAUAUAAAUACAGCACCAUGUGGGGAUGCACGGAUAUUUAGUCCUCACGAAAACGACACUGGUGU ..(((((((.(((...((((((...............))))))....))).))))))).........(((((((((((((((.............)))))))))..........)))))) ( -30.58) >DroWil_CAF1 20465 120 + 1 GAAUACUGACGGGCUAUACCCAUAUAAACUAAAAUCCGGGGUACAUUUCCAUUUGUAUAUUAAUACAGCACCUUGUGGGGAUGCACGGAUAUUUAGUCCUCACGAAAACGACACUGGUGU ..(((((...(((.....)))......(((((((((((.....(((..((((((((((....))))))......))))..)))..))))).))))))..................))))) ( -27.70) >DroMoj_CAF1 5033 120 + 1 GAAUACAGACGGGCAAUACCCUUAUAAACUAAAAUCCGGGGUACAUUUCCAUUUGUAUAUAAAUACAGCACCAUGUGGGGAUGCACGGAUAUUUAGUCCUCACGAAAACGACACUGGUGU ..(((((((.(((...((((((...............))))))....))).))))))).........(((((((((((((((.............)))))))))..........)))))) ( -30.58) >DroPer_CAF1 6482 120 + 1 GAAUACUGAUGGGCAAUACCCUUAUAAACUAAAAUCCGGGGUACAUUUCCAUUUGUAUAUAAAUACAGCACCUUGUGGGGAUGCACGGAUAUUUAGUCCUCACGAAAACGACACAGGUGU ..((((.((((((...((((((...............))))))....)))))).)))).........(((((((((((((((.............)))))))))..........)))))) ( -31.08) >consensus GAAUACAGACGGGCAAUACCCUUAUAAACUAAAAUCCGGGGUACAUUUCCAUUUGUAUAUAAAUACAGCACCAUGUGGGGAUGCACGGAUAUUUAGUCCUCACGAAAACGACACUGGUGU .....(((..(((.....)))......(((((((((((.....(((..((((((((((....))))))......))))..)))..))))).))))))................))).... (-26.32 = -26.48 + 0.17)
| Location | 1,637,941 – 1,638,061 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.96 |
| Mean single sequence MFE | -29.86 |
| Consensus MFE | -27.87 |
| Energy contribution | -27.90 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.93 |
| SVM decision value | 2.51 |
| SVM RNA-class probability | 0.994822 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
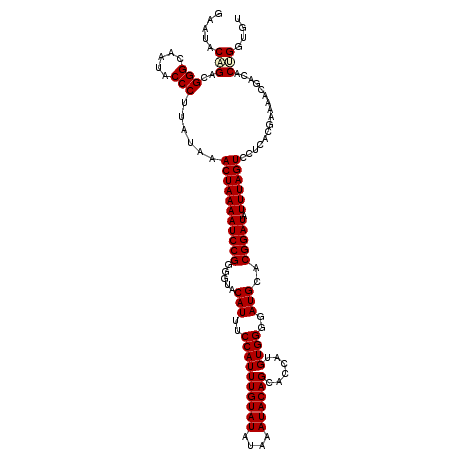
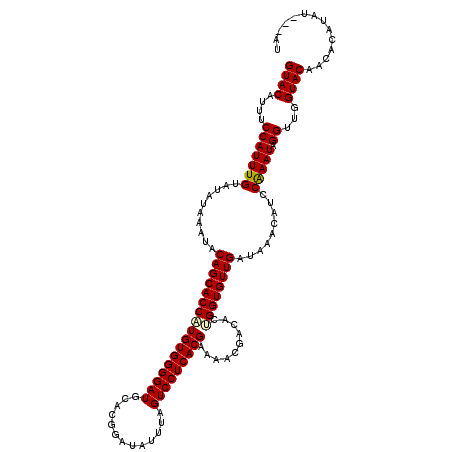
>X_DroMel_CAF1 1637941 120 + 22224390 GUACAUUUCCAUUUGUAUAUAAAUACAGCACCUUGUGGGGAUGCACGGAUAUUUAGUCCUCACGAAAACGACACUGGUGUUGAUAAACAUCCAAAUAGGUUGGUACAAACUGCAUUCUAU ((((....(((((((..........(((((((((((((((((.............))))))))))..........))))))).........))))).))...)))).............. ( -28.53) >DroVir_CAF1 5218 117 + 1 GUACAUUUCCAUUUGUAUAUAAAUACAGCACCAUGUGGGGAUGCACGGAUAUUUAGUCCUCACGAAAACGACACUGGUGUUGAUAAGCAUCCAAAUAGGUUGGUACAACACUUAU---UU ((((........((((((....)))))).(((.(((..((((((..((((.....)))).........(((((....)))))....))))))..))))))..)))).........---.. ( -30.60) >DroGri_CAF1 4093 117 + 1 GUACAUUUCCAUUUGUAUAUAAAUACAGCACCAUGUGGGGAUGCACGGAUAUUUAGUCCUCACGAAAACGACACUGGUGUUGAUAAGCAUCCAAAUAGGUUCGUACAACAAAUAU---CU ((((.........(((((....)))))(.(((.(((..((((((..((((.....)))).........(((((....)))))....))))))..)))))).))))).........---.. ( -31.50) >DroWil_CAF1 20505 120 + 1 GUACAUUUCCAUUUGUAUAUUAAUACAGCACCUUGUGGGGAUGCACGGAUAUUUAGUCCUCACGAAAACGACACUGGUGUUGAUAAACAUCCGAAUAGGUUGGUACAAAACGCUCUCUCU ((((....(((((((.((.((.((.(((((((((((((((((.............))))))))))..........))))))))).)).)).))))).))...)))).............. ( -29.12) >DroMoj_CAF1 5073 117 + 1 GUACAUUUCCAUUUGUAUAUAAAUACAGCACCAUGUGGGGAUGCACGGAUAUUUAGUCCUCACGAAAACGACACUGGUGUUGAUAAGCAUCCAAAUAGGUUGGUACAACACAUUU---UU ((((........((((((....)))))).(((.(((..((((((..((((.....)))).........(((((....)))))....))))))..))))))..)))).........---.. ( -30.60) >DroAna_CAF1 2475 120 + 1 GUACAUUUCCAUUUGUAUAUAAAUACAGCACCUUGUGGGGAUGCACGGAUAUUUAGUCCUCACGAAAACGACACAGGUGUUGAUAAACAUCCAAAUAGGUUGGUACAAUCGAUAUAUUAU ((((....(((((((..........(((((((((((((((((.............)))))))))..........)))))))).........))))).))...)))).............. ( -28.83) >consensus GUACAUUUCCAUUUGUAUAUAAAUACAGCACCAUGUGGGGAUGCACGGAUAUUUAGUCCUCACGAAAACGACACUGGUGUUGAUAAACAUCCAAAUAGGUUGGUACAACACAUAU___AU ((((....(((((((..........(((((((((((((((((.............)))))))))..........)))))))).........))))).))...)))).............. (-27.87 = -27.90 + 0.03)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:48:24 2006