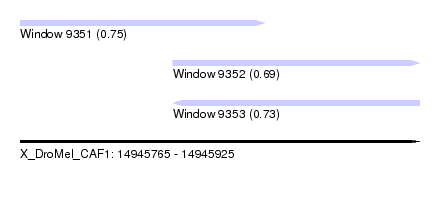
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 14,945,765 – 14,945,925 |
| Length | 160 |
| Max. P | 0.746466 |

| Location | 14,945,765 – 14,945,863 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 74.87 |
| Mean single sequence MFE | -39.35 |
| Consensus MFE | -22.83 |
| Energy contribution | -23.58 |
| Covariance contribution | 0.75 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.58 |
| Structure conservation index | 0.58 |
| SVM decision value | 0.46 |
| SVM RNA-class probability | 0.746466 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14945765 98 + 22224390 ----GAUGUCAUCAUC-----AUCCGC--CUCCA-----AUC---AUCCCGGGCAUUCGCAGUCGCAGCAGCGCGCCAGUCACGGCAGCGUCGCCG---CUGUGCUGCAAUCAAUGCCGG ----((((....))))-----......--.....-----...---...((((.((((.((((.((((((.((((((..((....)).))).))).)---)))))))))....)))))))) ( -38.20) >DroVir_CAF1 2501 114 + 1 CGCAGCUGUCCCAAUCCCAAACCCAAUCCCAUUCGCAAUCCC---AGCAGCAGCAAUCGCAGUCACAGCAGCGCGCCAGCUACGGCAGCGUCGCCG---CUGCGCUGCAGUCAUUGCCUG .((.(((((.................................---.))))).))....(((((.((.((((((((((......)))((((....))---))))))))).)).)))))... ( -37.28) >DroSec_CAF1 2150 98 + 1 ----GAUGUCAUCAUC-----GUCCGC--CUCCA-----AUC---AUCCCGGGCAUUCGCAGUCGCAGCAGCGCGCCAGUCACGGCAGCGUGGCCG---CUGUGCUGCAGUCAAUGCCGG ----((((....))))-----......--.....-----...---...((((.((((.((((.((((((.(((((((......))).))))....)---)))))))))....)))))))) ( -37.70) >DroEre_CAF1 2123 98 + 1 ----GAUGUCAUCAUC-----GUCCGC--CUCCA-----AUU---AUACGGGGCAUUCGCAGUCGCAGCAGCGCGCCAGCCACGGCAGCGUCGCCG---CUGUGCUGCAGUCAAUGCCGG ----((((....))))-----..((((--(.((.-----...---....)))))....((((.((((((.((((((..((....)).))).))).)---)))))))))..........)) ( -41.50) >DroYak_CAF1 4437 98 + 1 ----GAUGUCAUCAUC-----GUCCGC--CUCCA-----AUC---AUCACGGGCAUUCACAGUCGCAGCAGCGCGCUAGUCACGGCAGCGUCGCCG---CUGUGCUGCAGUCAAUGCCGG ----((((....))))-----......--.....-----...---....(.((((((..(((.((((((.(((((((.((....)))))).))).)---)))))))).....)))))).) ( -36.60) >DroAna_CAF1 2070 98 + 1 ----C------GCCUC-----CUCCGG--CUCCC-----CCUUCGGGCACGCCCAUCCGCAGGCGCAGCAGCGCGCCAGCCAUGGCAGCGUCGCUGCCUCUGUGCUCCAGUCCAUGCCGG ----.------.....-----..((((--(....-----.((..(((((((((........)))(((((.((((((((....)))).)))).)))))....)))))).)).....))))) ( -44.80) >consensus ____GAUGUCAUCAUC_____GUCCGC__CUCCA_____AUC___AUCACGGGCAUUCGCAGUCGCAGCAGCGCGCCAGCCACGGCAGCGUCGCCG___CUGUGCUGCAGUCAAUGCCGG ...................................................((((((.((((.((((((.(((((((......))).)))).))......))))))))....)))))).. (-22.83 = -23.58 + 0.75)

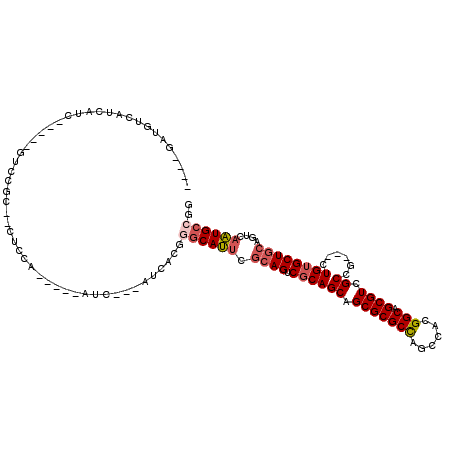
| Location | 14,945,826 – 14,945,925 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | forward |
| Mean pairwise identity | 78.92 |
| Mean single sequence MFE | -42.87 |
| Consensus MFE | -30.33 |
| Energy contribution | -30.93 |
| Covariance contribution | 0.60 |
| Combinations/Pair | 1.22 |
| Mean z-score | -1.27 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.33 |
| SVM RNA-class probability | 0.691707 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14945826 99 + 22224390 CACGGCAGCGUCGCCG---CUGUGCUGCAAUCAAUGCCGGAGGCUGAGCCCGGUUACGGAUCCGGCUCCGGUUCAAUAUCCGGCUCCAGUUCCGGCUCCGGU ..((((((((.((...---.)))))))).......(((((((.(((((((.((.((..((.(((....))).))..)).)))))).))))))))))..)).. ( -42.70) >DroSec_CAF1 2211 99 + 1 CACGGCAGCGUGGCCG---CUGUGCUGCAGUCAAUGCCGGAGGCUGAGCCCGGUUACGGAUCCGGCUCCGGUUCGAUAUCCGGGUCCAGUUCCGGCUCCGGU ..((((((((..(...---)..)))))).......(((((((.(((..(((((.((((((.(((....))))))).)).)))))..))))))))))..)).. ( -51.80) >DroEre_CAF1 2184 99 + 1 CACGGCAGCGUCGCCG---CUGUGCUGCAGUCAAUGCCGGAGGCUGAGCCCGGUUACGGAUCCGGCUCCGGUUCGAUAUCCGGCUCCAGUUCCGGCUCCGGU ..((((((((.((...---.)))))))).......(((((((.(((((((.((.((((((.(((....))))))).)).)))))).))))))))))..)).. ( -44.10) >DroWil_CAF1 10458 75 + 1 UACGGCAGCGUGUCCG---CUGCGCUGCCAACAAUGCCGGAGG-UGAGU---GUUCCGGGU--------------------GGAUCUAAUGCCGGUGCUGGU ...((((((((.....---..))))))))..((..(((((...-..((.---..(((....--------------------))).))....)))))..)).. ( -30.20) >DroYak_CAF1 4498 99 + 1 CACGGCAGCGUCGCCG---CUGUGCUGCAGUCAAUGCCGGAGGCUGAGCCCGGUUACGGAUCCGGCUCCGGUUCGAUAUCCGGCUCCAGUUCCGGCUCCGGU ..((((((((.((...---.)))))))).......(((((((.(((((((.((.((((((.(((....))))))).)).)))))).))))))))))..)).. ( -44.10) >DroAna_CAF1 2128 96 + 1 CAUGGCAGCGUCGCUGCCUCUGUGCUCCAGUCCAUGCCGGAGACGGAGCCCGGCUACGGAUCUGGAUCCGGCU------CCGGUUCGAGUUCAGGCUCCGGC ...((((((...))))))((((.((((.((.((..((((((..((((..(((....))).))))..)))))).------..)))).)))).))))....... ( -44.30) >consensus CACGGCAGCGUCGCCG___CUGUGCUGCAGUCAAUGCCGGAGGCUGAGCCCGGUUACGGAUCCGGCUCCGGUUCGAUAUCCGGCUCCAGUUCCGGCUCCGGU ....((((((..(......)..)))))).......(((((((.(.((((((((........))))..((((........)))).....)))).).))))))) (-30.33 = -30.93 + 0.60)

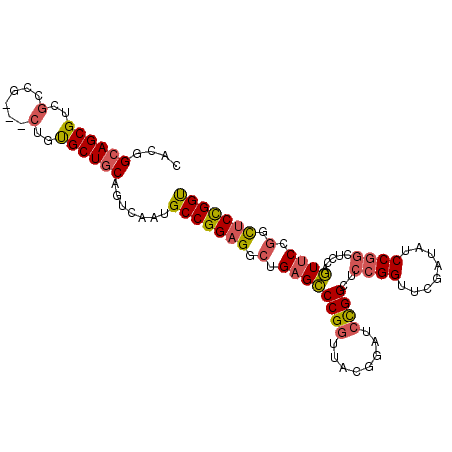

| Location | 14,945,826 – 14,945,925 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | reverse |
| Mean pairwise identity | 78.92 |
| Mean single sequence MFE | -42.22 |
| Consensus MFE | -24.87 |
| Energy contribution | -27.21 |
| Covariance contribution | 2.34 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.42 |
| SVM RNA-class probability | 0.729126 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14945826 99 - 22224390 ACCGGAGCCGGAACUGGAGCCGGAUAUUGAACCGGAGCCGGAUCCGUAACCGGGCUCAGCCUCCGGCAUUGAUUGCAGCACAG---CGGCGACGCUGCCGUG ..(((.((((((....(((((((.(((.((.(((....))).)).))).)).)))))....)))))).............(((---((....)))))))).. ( -45.20) >DroSec_CAF1 2211 99 - 1 ACCGGAGCCGGAACUGGACCCGGAUAUCGAACCGGAGCCGGAUCCGUAACCGGGCUCAGCCUCCGGCAUUGACUGCAGCACAG---CGGCCACGCUGCCGUG ..(((.((((((.(((..(((((.(((.((.(((....))).)).))).)))))..)))..)))))).............(((---((....)))))))).. ( -48.00) >DroEre_CAF1 2184 99 - 1 ACCGGAGCCGGAACUGGAGCCGGAUAUCGAACCGGAGCCGGAUCCGUAACCGGGCUCAGCCUCCGGCAUUGACUGCAGCACAG---CGGCGACGCUGCCGUG ..(((.((((((....(((((((.(((.((.(((....))).)).))).)).)))))....)))))).............(((---((....)))))))).. ( -45.20) >DroWil_CAF1 10458 75 - 1 ACCAGCACCGGCAUUAGAUCC--------------------ACCCGGAAC---ACUCA-CCUCCGGCAUUGUUGGCAGCGCAG---CGGACACGCUGCCGUA .((((((((((....((.(((--------------------....)))..---.))..-...))))...))))))..((((((---((....))))).))). ( -25.30) >DroYak_CAF1 4498 99 - 1 ACCGGAGCCGGAACUGGAGCCGGAUAUCGAACCGGAGCCGGAUCCGUAACCGGGCUCAGCCUCCGGCAUUGACUGCAGCACAG---CGGCGACGCUGCCGUG ..(((.((((((....(((((((.(((.((.(((....))).)).))).)).)))))....)))))).............(((---((....)))))))).. ( -45.20) >DroAna_CAF1 2128 96 - 1 GCCGGAGCCUGAACUCGAACCGG------AGCCGGAUCCAGAUCCGUAGCCGGGCUCCGUCUCCGGCAUGGACUGGAGCACAGAGGCAGCGACGCUGCCAUG .((((..((.(........).))------..)))).(((((.(((((.((((((.......)))))))))))))))).......((((((...))))))... ( -44.40) >consensus ACCGGAGCCGGAACUGGAGCCGGAUAUCGAACCGGAGCCGGAUCCGUAACCGGGCUCAGCCUCCGGCAUUGACUGCAGCACAG___CGGCGACGCUGCCGUG .((((...((((.((((..((((........))))..)))).))))...)))).....(((...)))....((.(((((..............))))).)). (-24.87 = -27.21 + 2.34)


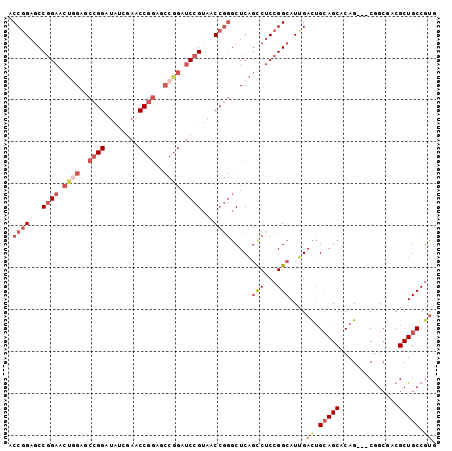
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:55:49 2006