
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 14,830,565 – 14,830,681 |
| Length | 116 |
| Max. P | 0.815105 |

| Location | 14,830,565 – 14,830,681 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.67 |
| Mean single sequence MFE | -42.55 |
| Consensus MFE | -36.90 |
| Energy contribution | -37.02 |
| Covariance contribution | 0.11 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.69 |
| Structure conservation index | 0.87 |
| SVM decision value | 0.66 |
| SVM RNA-class probability | 0.815105 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
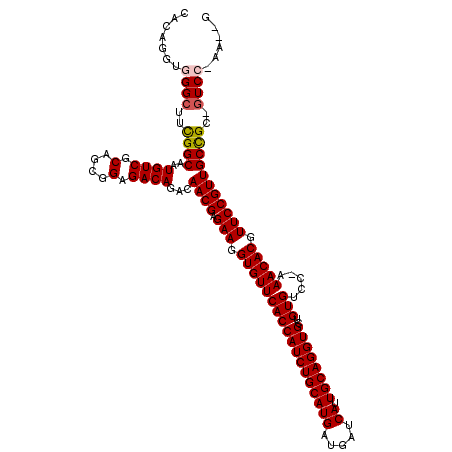
>X_DroMel_CAF1 14830565 116 + 22224390 UGCAGGUGGGCUUUGGCAAUGUCGCCGCGGAGACAGACAACGAGAAGGUGUUCACCAUCUGCAUGAUGAUCAUUGCAGGUGCUGUGUCCUUAACACGUUCCGUUGCUGC-GUCC-AA--G .((.(((((((....))....)))))))(((..(((.(((((.(((.((((((((((((((((((.....)).))))))))..))).....))))).))))))))))).-.)))-..--. ( -44.60) >DroVir_CAF1 15575 115 + 1 AACAGGUGGGCUUCGGCAAUGUCGCAGCGGAGACAGACAACGAGAAGGUGUUCACCAUCUGCAUGAUGAUCAUUGCAGGUGCUGUGUCC-AAACACGUUCCGUUGCCGC-GUCC-AA--A ......(((((..((((..((((........))))...((((.(((.((((((((((((((((((.....)).))))))))..)))...-.))))).))))))))))).-))))-).--. ( -43.00) >DroGri_CAF1 8030 116 + 1 CACAGGUGGGCUUCGGCAAUGUCGCAGCGGAGACAGACAACGAGAAGGUGUUCACCAUCUGCAUGAUGAUCAUUGCAGGUGCUGUGUCC-AAACACGUUCCGUUGCCGC-GUCCAAA--G ......(((((..((((..((((........))))...((((.(((.((((((((((((((((((.....)).))))))))..)))...-.))))).))))))))))).-)))))..--. ( -43.00) >DroWil_CAF1 14076 118 + 1 CACAGGUGGGCUUUGGCAAUGUCGCCGCGGAGACAGACAACGAGAAGGUGUUCACCAUCUGCAUGAUGAUCAUUGCAGGUGCUGUGUCC-AAACACGUUCCGUUGCCGUUGUCA-GAAUG ..((....(((..((((..((((.(....).))))...((((.(((.((((((((((((((((((.....)).))))))))..)))...-.))))).)))))))))))..))).-...)) ( -40.80) >DroMoj_CAF1 13071 115 + 1 CACAGGUGGGAUUCGGCAAUGUCGCAGCGGAGACAGACAACGAGAAGGUGUUCACCAUCUGCAUGAUGAUCAUUGCAGGUGCUGUGUCC-AAACACGUUCCGUUGCCGC-GUCA-AA--G .........(((.((((..((((........))))...((((.(((.((((((((((((((((((.....)).))))))))..)))...-.))))).))))))))))).-))).-..--. ( -39.70) >DroAna_CAF1 9480 116 + 1 UCCAGGUGGGCUUUGGCAAUGUCGCCGCGGAGACAGACAACGAGAAGGUGUUCACCAUCUGCAUGAUGAUCAUUGCAGGUGCUGUGUCCUUAACACGUUCCGUUGCCGC-GUCC-AA--U ......(((((...(((......)))((((.......(((((.(((.((((((((((((((((((.....)).))))))))..))).....))))).))))))))))))-))))-).--. ( -44.21) >consensus CACAGGUGGGCUUCGGCAAUGUCGCAGCGGAGACAGACAACGAGAAGGUGUUCACCAUCUGCAUGAUGAUCAUUGCAGGUGCUGUGUCC_AAACACGUUCCGUUGCCGC_GUCC_AA__G .......((((..((((..((((.(....).))))...((((.(((.((((((((((((((((((.....)).))))))))..))).....))))).)))))))))))..))))...... (-36.90 = -37.02 + 0.11)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:55:06 2006