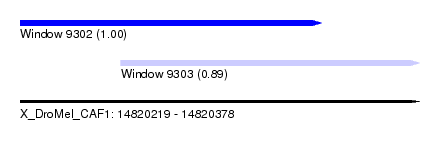
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 14,820,219 – 14,820,378 |
| Length | 159 |
| Max. P | 0.998690 |

| Location | 14,820,219 – 14,820,339 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.83 |
| Mean single sequence MFE | -40.33 |
| Consensus MFE | -38.14 |
| Energy contribution | -38.25 |
| Covariance contribution | 0.11 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.95 |
| SVM decision value | 3.19 |
| SVM RNA-class probability | 0.998690 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
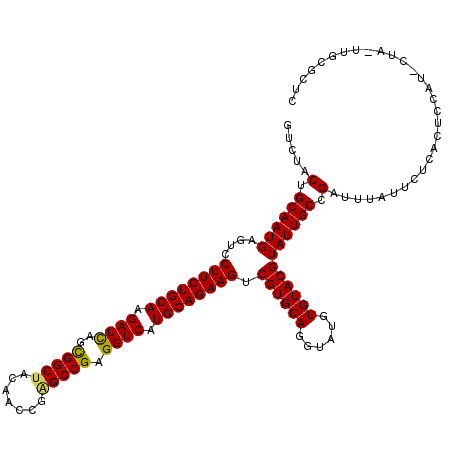
>X_DroMel_CAF1 14820219 120 + 22224390 GUUUCUUUUGGCCAACGCACAAAUCGUCGAUUUCCCGAUCGUCUACUGCAAUGAGUCCUUCUGCAAGAUCAGCGGCUAUAACCGUGCCGAGGUCAUGCAGAAGUCGUGCAGGUAUGUGUA ................(((((....(.(((((....))))).)..(((((.(((...((((((((.((((..((((.........)))).)))).))))))))))))))))...))))). ( -40.40) >DroVir_CAF1 13 120 + 1 GUUUCUAUUGGCCAACGCACAAAUCGUCGAUUUUCCGAUCGUCUACUGCAAUGAGUCCUUCUGCAAGAUUAGCGGCUACAACCGAGCCGAGGUCAUGCAGAAGUCGUGCAGGUAUGUGUA ................(((((....(.(((((....))))).)..(((((.(((...((((((((.((((..(((((.......))))).)))).))))))))))))))))...))))). ( -40.30) >DroPse_CAF1 11565 120 + 1 GUUUCUUUUGGCCAACGCACAAAUCGUCGAUUUUCCGAUCGUCUACUGCAAUGAGUCCUUCUGCAAGAUCAGCGGCUACAACCGGGCCGAGGUCAUGCAGAAGUCGUGCAGGUAUGUGUA ................(((((....(.(((((....))))).)..(((((.(((...((((((((.((((..(((((.......))))).)))).))))))))))))))))...))))). ( -41.60) >DroEre_CAF1 14 120 + 1 GUUUCUUUUGGCUAACGCACAAAUCGUCGAUUUCCCGAUCGUCUACUGCAAUGAGUCCUUCUGCAAGAUCAGCGGCUAUAACCGUGCCGAGGUCAUGCAGAAGUCGUGCAGGUAUGUGUA ................(((((....(.(((((....))))).)..(((((.(((...((((((((.((((..((((.........)))).)))).))))))))))))))))...))))). ( -40.40) >DroWil_CAF1 14 120 + 1 GUUUCUAUUGGCAAAUGCACAAAUCGUUGACUUUCCCAUUGUGUACUGCAAUGAGUCCUUCUGCAAGAUCAGUGGCUACAAUCGAGCCGAGGUCAUGCAGAAGUCGUGCAGGUAUGUGUA ..........((...(((((........((((....((((((.....))))))))))((((((((.((((..(((((.......))))).)))).))))))))..))))).))....... ( -39.00) >DroMoj_CAF1 14 120 + 1 GUUUCUAUUGGCCAACGCACAAAUCGUCGAUUUUCCGAUCGUCUACUGCAAUGAGUCCUUCUGCAAGAUUAGCGGCUACAACCGAGCCGAGGUCAUGCAGAAGUCGUGCAGGUAUGUGUA ................(((((....(.(((((....))))).)..(((((.(((...((((((((.((((..(((((.......))))).)))).))))))))))))))))...))))). ( -40.30) >consensus GUUUCUAUUGGCCAACGCACAAAUCGUCGAUUUUCCGAUCGUCUACUGCAAUGAGUCCUUCUGCAAGAUCAGCGGCUACAACCGAGCCGAGGUCAUGCAGAAGUCGUGCAGGUAUGUGUA ................(((((....(.(((((....))))).)..(((((.(((...((((((((.((((..(((((.......))))).)))).))))))))))))))))...))))). (-38.14 = -38.25 + 0.11)



| Location | 14,820,259 – 14,820,378 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.75 |
| Mean single sequence MFE | -38.57 |
| Consensus MFE | -34.56 |
| Energy contribution | -34.20 |
| Covariance contribution | -0.36 |
| Combinations/Pair | 1.10 |
| Mean z-score | -1.75 |
| Structure conservation index | 0.90 |
| SVM decision value | 0.97 |
| SVM RNA-class probability | 0.892409 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14820259 119 + 22224390 GUCUACUGCAAUGAGUCCUUCUGCAAGAUCAGCGGCUAUAACCGUGCCGAGGUCAUGCAGAAGUCGUGCAGGUAUGUGUACGUAUUGCCGAUUUAUACUCACUUCAA-CUACUUUCGCUC ...........(((((.((((((((.((((..((((.........)))).)))).))))))))(((.((((.((((....))))))))))).....)))))......-............ ( -35.80) >DroVir_CAF1 53 118 + 1 GUCUACUGCAAUGAGUCCUUCUGCAAGAUUAGCGGCUACAACCGAGCCGAGGUCAUGCAGAAGUCGUGCAGGUAUGUGUACGUAUUGCCGAUUUAUAUUCACUCGCU-CUG-UUGCGCUC .......(((((((((.((((((((.((((..(((((.......))))).)))).))))))))(((.((((.((((....))))))))))).............)))-).)-)))).... ( -40.80) >DroPse_CAF1 11605 115 + 1 GUCUACUGCAAUGAGUCCUUCUGCAAGAUCAGCGGCUACAACCGGGCCGAGGUCAUGCAGAAGUCGUGCAGGUAUGUGUACGUAUUGCCGAUUUAUUCUCACUCCGA-A----CUCGCUC ............((((.((((((((.((((..(((((.......))))).)))).))))))))(((.((((.((((....)))))))))))................-)----))).... ( -36.40) >DroWil_CAF1 54 120 + 1 GUGUACUGCAAUGAGUCCUUCUGCAAGAUCAGUGGCUACAAUCGAGCCGAGGUCAUGCAGAAGUCGUGCAGGUAUGUGUACGUAUUGCCGAUUUAUUUUCACUCCGCUCAAAUUGCUGUU ....((.(((((((((.((((((((.((((..(((((.......))))).)))).))))))))(((.((((.((((....)))))))))))..............)))))..)))).)). ( -40.00) >DroMoj_CAF1 54 118 + 1 GUCUACUGCAAUGAGUCCUUCUGCAAGAUUAGCGGCUACAACCGAGCCGAGGUCAUGCAGAAGUCGUGCAGGUAUGUGUACGUAUUGCCGAUUUAUUCUCACUCGCU-CUG-UUGCGCUC .......(((((((((.((((((((.((((..(((((.......))))).)))).))))))))(((.((((.((((....))))))))))).............)))-).)-)))).... ( -40.80) >DroAna_CAF1 54 116 + 1 GUCUACUGCAAUGAGUCCUUCUGCAAGAUCAGCGGCUACAACCGGGCCGAGGUCAUGCAGAAGUCGUGCAGGUAUGUGUACGUAUUGCCGAUUUAUUCUC---UCAU-CCUCUUUCGAGU ..........(((((..((((((((.((((..(((((.......))))).)))).))))))))(((.((((.((((....)))))))))))........)---))))-.(((....))). ( -37.60) >consensus GUCUACUGCAAUGAGUCCUUCUGCAAGAUCAGCGGCUACAACCGAGCCGAGGUCAUGCAGAAGUCGUGCAGGUAUGUGUACGUAUUGCCGAUUUAUUCUCACUCCAU_CUA_UUGCGCUC .....(.((((((....((((((((.((((..(((((.......))))).)))).)))))))).((((((......)))))))))))).).............................. (-34.56 = -34.20 + -0.36)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:55:04 2006