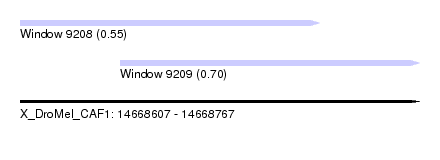
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 14,668,607 – 14,668,767 |
| Length | 160 |
| Max. P | 0.696614 |

| Location | 14,668,607 – 14,668,727 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.33 |
| Mean single sequence MFE | -42.37 |
| Consensus MFE | -30.15 |
| Energy contribution | -29.02 |
| Covariance contribution | -1.13 |
| Combinations/Pair | 1.45 |
| Mean z-score | -1.43 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.03 |
| SVM RNA-class probability | 0.546258 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14668607 120 + 22224390 GUGAUCUCUGCUGUGUGAUUAGCAGUCUGGCGCAGCUAUUGCUCGAUCCGCAUUUCCGUACUAUCGAUGGCUUCCAAUCGCUGGUGCAGAAGGAGUGGGUGGCCCUGGAGCAUCCGUUUC (.(((((((((((((((((.....)))..)))))))....(((..((((((.((((.((((((.((((((...)).)))).)))))).))))..)))))))))...)))).))))..... ( -40.20) >DroGri_CAF1 1017 120 + 1 GCGAUCUGUGCUGUGUCAUUAGCAGCCUCACGCAACUCCUUCUCGAUCCGCAUUUUCGCACCAUCGAUGGAUUUCAGUCACUGGUGCAGAAGGAUUGGGUUGCGUUGGAGCAUCCGUUUC (((.....(((((......)))))((((.((((((((((((((.((.........))((((((..(((.(....).)))..)))))))))))))....))))))).)).))...)))... ( -40.20) >DroWil_CAF1 1415 120 + 1 GGGACUUAUGCUGUGUCAUCAGCAGUCUGACUCAACUGCAAUUGGAUCCAUAUUUUCGCAGCAUCGAUGGCUUCCAGUCGCUCAUUCAAAAGGAAUGGAUUGCUCUCGAGCAUCCGUUUC (.(((..((((((((......(((((........)))))...((....))......))))))))...(((...)))))).)..........(((((((((.(((....)))))))))))) ( -34.10) >DroYak_CAF1 1442 120 + 1 GUGAUCUGUGCUGUGUGAUCAGCAGUCUAACGCAAUUGCUGCUCGAUCCGCAUUUCCGCACCAUCGAUGGCUUCCAGUCGCUGGUGCAGAAGGAAUGGGUGGCUUUGGAGCAUCCGUUUC ...........((((.((((((((((...........)))))).)))))))).(((.((((((.((((((...))).))).)))))).)))((((((((((.((....)))))))))))) ( -44.30) >DroMoj_CAF1 1038 120 + 1 GGGAUUUGUGCUGCGUGAUUAGCAGCUUGACUCAGCUGCUACUCGAUCCGCAUUUCCGCACCAUCGACGGCUUCCAGUCGCUGGUGCAAAAGGAAUGGGUGGCCUUGGAGCAUCCGUUCC .(((..((((.....(((.((((((((......)))))))).)))...))))..)))((((((.((((........)))).))))))....((((((((((.(....)..)))))))))) ( -55.20) >DroAna_CAF1 1403 120 + 1 GGGAUCUCUGCUGUGUGAUUAGUAGUCUGACCCAGCUCCUGCUUGAUCCUCACUUCCGGAGCAUCGAUGGCUUCCAGUCGCUGAUUCAAAAGGAAUGGGUGGCCCUGGAGCAUCCGUUCC (((.((.((((((......))))))...))))).(((((.(((((((.(((.......))).))))..))).....((((((.((((.....)))).))))))...)))))......... ( -40.20) >consensus GGGAUCUGUGCUGUGUGAUUAGCAGUCUGACGCAACUGCUGCUCGAUCCGCAUUUCCGCACCAUCGAUGGCUUCCAGUCGCUGGUGCAAAAGGAAUGGGUGGCCCUGGAGCAUCCGUUUC (((.....(((((......)))))......)))........................((((((.((((........)))).))))))....(((((((((.((......))))))))))) (-30.15 = -29.02 + -1.13)
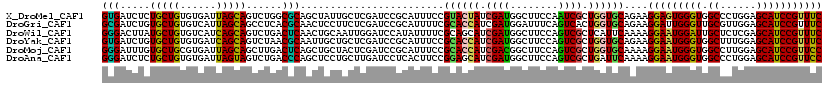

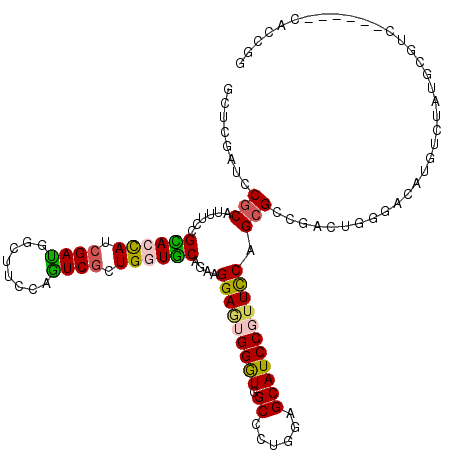
| Location | 14,668,647 – 14,668,767 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.68 |
| Mean single sequence MFE | -45.52 |
| Consensus MFE | -27.58 |
| Energy contribution | -27.37 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.34 |
| SVM RNA-class probability | 0.696614 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14668647 120 + 22224390 GCUCGAUCCGCAUUUCCGUACUAUCGAUGGCUUCCAAUCGCUGGUGCAGAAGGAGUGGGUGGCCCUGGAGCAUCCGUUUCAGCGACGCCUGGGUCAUGUCUAUCCUGCCCAGCCGGCCGG .....((((((.((((.((((((.((((((...)).)))).)))))).))))..))))))(((((((((((....)))))))....(.(((((.((.(.....).))))))).))))).. ( -45.70) >DroVir_CAF1 1077 114 + 1 GCUCGAUCCGCAUUUUCGCACCAUCGAUGGCUUUCAGUCGCUGGUGCAGAAGGAGUGGGUGGCCUUGGAGCAUCCCUUCCAGCGGCGACUCGGACAUGUGUAUGUAUC------AACAGG (..(((.((((.(((((((((((.((((........)))).)))))).))))).))))((.(((((((((......)))))).))).)))))..).............------...... ( -40.70) >DroPse_CAF1 1422 117 + 1 ACUCGAUCCGCACUUCCGUAGCAUCGAUGGCUUCCAGUCGCUGGUGCAGAAGGAGUGGAUCGCCCUCGAGCAUCCCUUCCAGCGUCGGCUCGGACAUGUCUACUCGGC---GACCAACGG ....(((((((.((((.(((.((.((((((...))).))).)).))).))))..)))))))(((....(((((....((((((....))).))).))).))....)))---......... ( -39.10) >DroGri_CAF1 1057 114 + 1 UCUCGAUCCGCAUUUUCGCACCAUCGAUGGAUUUCAGUCACUGGUGCAGAAGGAUUGGGUUGCGUUGGAGCAUCCGUUUCAGCGUCGAUUGGGUCAUGUCUAUGUGUC------UCGAGG .(((((..(((((....((((((..(((.(....).)))..))))))(((..((((.((((((((((((((....))))))))).))))).))))...))))))))..------))))). ( -47.10) >DroMoj_CAF1 1078 114 + 1 ACUCGAUCCGCAUUUCCGCACCAUCGACGGCUUCCAGUCGCUGGUGCAAAAGGAAUGGGUGGCCUUGGAGCAUCCGUUCCAGCGCCGCCUGGGUCAUGUUUAUGUGUC------CAACAG ....(((((.((((((.((((((.((((........)))).))))))....))))))((((((.(((((((....))))))).)))))).))))).......(((...------..))). ( -50.50) >DroAna_CAF1 1443 120 + 1 GCUUGAUCCUCACUUCCGGAGCAUCGAUGGCUUCCAGUCGCUGAUUCAAAAGGAAUGGGUGGCCCUGGAGCAUCCGUUCCAGCGCCGUCUGGGACAUGUCUAUCCCACCCAGAGCUCUGG ...............(((((((.((((((((.....((((((.((((.....)))).)))))).(((((((....))))))).))))))(((((........)))))....))))))))) ( -50.00) >consensus GCUCGAUCCGCAUUUCCGCACCAUCGAUGGCUUCCAGUCGCUGGUGCAGAAGGAGUGGGUGGCCCUGGAGCAUCCGUUCCAGCGCCGACUGGGACAUGUCUAUGCGUC______CACCGG ........(((......((((((.((((........)))).))))))....(((((((((.((......))))))))))).))).................................... (-27.58 = -27.37 + -0.22)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:53:40 2006