
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 14,528,020 – 14,528,111 |
| Length | 91 |
| Max. P | 0.611336 |

| Location | 14,528,020 – 14,528,111 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 103 |
| Reading direction | reverse |
| Mean pairwise identity | 81.48 |
| Mean single sequence MFE | -22.88 |
| Consensus MFE | -11.49 |
| Energy contribution | -11.38 |
| Covariance contribution | -0.11 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.52 |
| Structure conservation index | 0.50 |
| SVM decision value | 0.16 |
| SVM RNA-class probability | 0.611336 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
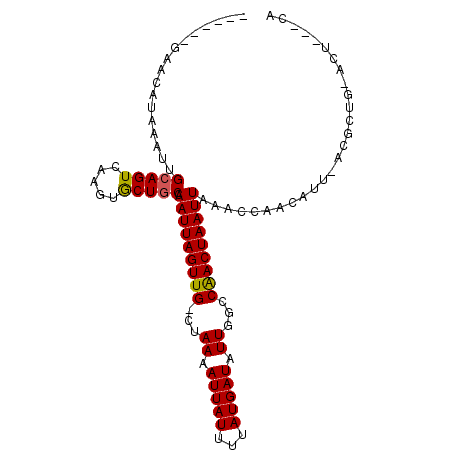
>X_DroMel_CAF1 14528020 91 - 22224390 -------ACUAUAAAUUGAAGUCAAGUGCUGCCAAUUAGUUG-CUAAAAUUAUUUUAUGAUAUUUUCCGACUAAUUAAAUGAACAUUUACGUUG-CGC---CA -------........(((....)))((((.((.(((((((((-..((((((((...))))).)))..)))))))))((((....))))..)).)-)))---.. ( -13.10) >DroVir_CAF1 2923 97 - 1 --UGUGGAACAUAAAUUGCAGUCAAGUGCUGCCAAUUAGUUGGCUAAAAUUAUUUUAUGAUAUUGGCCAACUAAUUAAACCAGCAUU-ACGCUGGACU---GA --...............(((((.....))))).((((((((((((((.(((((...))))).))))))))))))))...(((((...-..)))))...---.. ( -33.00) >DroGri_CAF1 2902 97 - 1 GCUAU-GCACAUAAAUUGCAGUCAAGUACUGCCAAUUAGUUGCUAAAAAUUAUUUUAUGAUAUUGGCCAACUAAUUAAACCAGUAUU-ACGCUG-ACU---GA .....-............(((((((((((((..(((((((((((((..(((((...))))).)))).)))))))))....)))))))-....))-)))---). ( -23.10) >DroMoj_CAF1 3619 96 - 1 --UGUGGCACAUAAAUUGCAGUCAAGUGCUGCCAAUUAGUUGGCUAAAAUUAUUUUAUGAUAUUGGCCAACUAAUUAAACCGGCAUU-ACGCUG-ACU---GA --................((((((.(((.((((((((((((((((((.(((((...))))).)))))))))))))).....)))).)-))..))-)))---). ( -35.40) >DroAna_CAF1 2951 89 - 1 -------ACCAUAAAUUGCAGUCAAGUGCUGCCAAUUAGUUG-CUAAAAUUAUUUUAUGAUAUUGUCCAACUAAUUAAAUGAACAUUAAGGGU---GU---CC -------(((.......(((((.....))))).(((((((((-.(((.(((((...))))).)))..)))))))))..............)))---..---.. ( -15.90) >DroPer_CAF1 5934 94 - 1 ------GACCAUAAAUUGCAGUCAAGUGCUGCCAAUUAGUUG-CUAAAAUUAUUUUAUGAUAUUGUCCAACUAAUUAAAUGAACAUUAAGG-GG-CGCAUUCG ------(((((.....))..))).(((((.((((((((((((-.(((.(((((...))))).)))..))))))))).(((....)))....-))-)))))).. ( -16.80) >consensus ______GAACAUAAAUUGCAGUCAAGUGCUGCCAAUUAGUUG_CUAAAAUUAUUUUAUGAUAUUGGCCAACUAAUUAAACCAACAUU_ACGCUG_ACU___CA .................(((((.....))))).(((((((((...((.(((((...))))).))...)))))))))........................... (-11.49 = -11.38 + -0.11)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:52:41 2006