
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 14,426,818 – 14,427,122 |
| Length | 304 |
| Max. P | 0.999380 |

| Location | 14,426,818 – 14,426,921 |
|---|---|
| Length | 103 |
| Sequences | 4 |
| Columns | 103 |
| Reading direction | forward |
| Mean pairwise identity | 85.67 |
| Mean single sequence MFE | -21.81 |
| Consensus MFE | -17.50 |
| Energy contribution | -18.38 |
| Covariance contribution | 0.88 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.59 |
| SVM RNA-class probability | 0.791064 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14426818 103 + 22224390 GCCCACGAGGCGUCGAAAUUUCAUCAGCCCAACGACCGCGUGUACGUACAUAACACUCUAUGUAUGUACAUAUGUACAUACACAUUCAUAGUUUCAAAGUUCU ......(((((((((.................))))((..((((((((((((......))))))))))))..))................)))))........ ( -23.33) >DroSec_CAF1 6224 97 + 1 GCCCACGAGGCGUCGAAAUUUCAUCAGCCCAACGAGCGCGUGUACGUACAUAACACUCUAUGUAUGUACAUAUGUAC------AUUCAUAGUUUCACAGUUCU (((.....)))((.((((((...((........))(((..((((((((((((......))))))))))))..)))..------......))))))))...... ( -24.60) >DroSim_CAF1 6236 97 + 1 GCCCACGAGGCGUCGAAAUUUCAUCAGCCCAGCGAGCGCGUGUACGUACAUAACACUCUAUGUAUGUACAUAUGUAC------AUUCAUAGUUUCACAGUUCU (((.....)))((.((((((......((...))(.(((..((((((((((((......))))))))))))..))).)------......))))))))...... ( -25.70) >DroYak_CAF1 6307 81 + 1 GCCCACGAGGCGUCGAAAUUUCAUCAGCCCAACCACCGCGUGU----ACAUAACACUCUAUGU------------AC------AUUCAUAAUUUUAAAGUUCU ......(.((((..((....))..).)))).......(.((((----(((((......)))))------------))------)).)................ ( -13.60) >consensus GCCCACGAGGCGUCGAAAUUUCAUCAGCCCAACGACCGCGUGUACGUACAUAACACUCUAUGUAUGUACAUAUGUAC______AUUCAUAGUUUCAAAGUUCU (((.....)))...((((((......((.........))(((((((((((((......)))))))))))))..................))))))........ (-17.50 = -18.38 + 0.88)



| Location | 14,426,853 – 14,426,948 |
|---|---|
| Length | 95 |
| Sequences | 3 |
| Columns | 95 |
| Reading direction | forward |
| Mean pairwise identity | 94.27 |
| Mean single sequence MFE | -20.60 |
| Consensus MFE | -19.70 |
| Energy contribution | -20.03 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.86 |
| Structure conservation index | 0.96 |
| SVM decision value | 2.02 |
| SVM RNA-class probability | 0.985869 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14426853 95 + 22224390 CCGCGUGUACGUACAUAACACUCUAUGUAUGUACAUAUGUACAUACACAUUCAUAGUUUCAAAGUUCUCCCACUAUUUUUCGGCUUGAUUCUGCC ..(((((((((((((((......)))))))))))))..............(((.((((..(((((.........)))))..)))))))....)). ( -19.60) >DroSec_CAF1 6259 89 + 1 GCGCGUGUACGUACAUAACACUCUAUGUAUGUACAUAUGUAC------AUUCAUAGUUUCACAGUUCUCCCACUAUUUUUCGGCUUGAUUCUGCC (((..((((((((((((......))))))))))))..)))..------.................................(((........))) ( -21.10) >DroSim_CAF1 6271 89 + 1 GCGCGUGUACGUACAUAACACUCUAUGUAUGUACAUAUGUAC------AUUCAUAGUUUCACAGUUCUCCCACUAUUUUUCGGCUUGAUUCUGCC (((..((((((((((((......))))))))))))..)))..------.................................(((........))) ( -21.10) >consensus GCGCGUGUACGUACAUAACACUCUAUGUAUGUACAUAUGUAC______AUUCAUAGUUUCACAGUUCUCCCACUAUUUUUCGGCUUGAUUCUGCC (((..((((((((((((......))))))))))))..))).........................................(((........))) (-19.70 = -20.03 + 0.33)

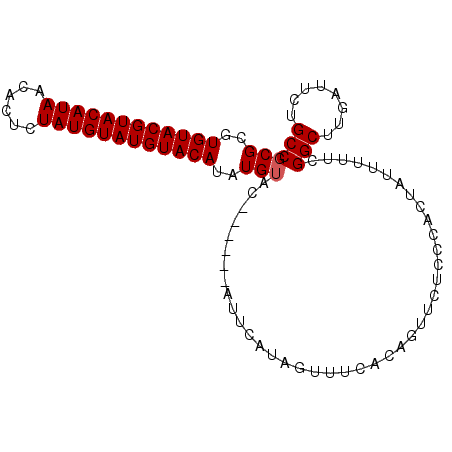
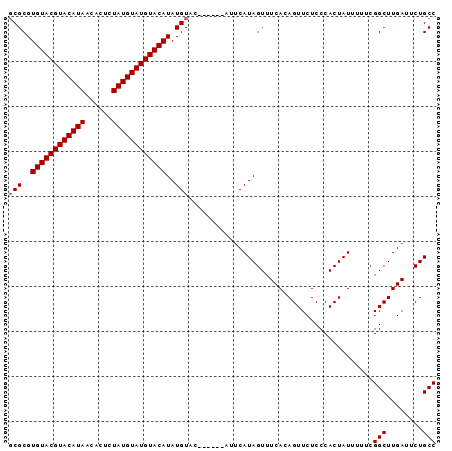
| Location | 14,426,948 – 14,427,044 |
|---|---|
| Length | 96 |
| Sequences | 5 |
| Columns | 99 |
| Reading direction | reverse |
| Mean pairwise identity | 95.00 |
| Mean single sequence MFE | -24.74 |
| Consensus MFE | -19.46 |
| Energy contribution | -19.78 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.17 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.93 |
| SVM RNA-class probability | 0.885115 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14426948 96 - 22224390 UGGAUACGUAUAAUAUAUUUAUGCUCGUGCAGAUAGAUAUCUAUUUGUUCAGCACACAAUCUUCGUACAUAUACGAUUUUUGGGGGCACAUCCAGU--- (((((..((((((.....))))))..(.((((((((....)))))))).).((...(((...(((((....)))))...)))...))..)))))..--- ( -26.20) >DroSec_CAF1 6348 94 - 1 GGGAUACGUAU--UAUAUUUAUGCUCGUGCAGAUAGAUAUCUAUUUGUUCAGCACACUAUCUUCGUACAUAUACGAUUUUUAGGGGCACAUCCAGU--- .((((..((..--........((((.(.((((((((....)))))))).)))))..(((...(((((....)))))....)))..))..))))...--- ( -22.50) >DroSim_CAF1 6360 94 - 1 UGGAUACGUAU--UAUAUUUAUGCUCGUGCAGAUAGAUAUCUAUUUGUUCAGCACACUAUCUUCGUACAUAUACGAUUUUUGGGGGCACAUCCAGU--- (((((..((..--........((((.(.((((((((....)))))))).)))))..(((...(((((....)))))....)))..))..)))))..--- ( -22.90) >DroEre_CAF1 6087 94 - 1 UGGAUACGUAU--UAUAUUUAUGCUCGUGCAGAUAGAUAUCUAUUUGCUCAGCCCACUAUCUUGGUACAUAUACGAUUUUUGGGGGCACAUCCAGU--- (((((..((((--.......))))..(.((((((((....)))))))).).((((.(((...(.(((....))).)....)))))))..)))))..--- ( -27.20) >DroYak_CAF1 6426 97 - 1 UGGAUACGUAU--UAUAUUUAUGCUCGUGCAGAUAGAUAUCUAUUUGUUCAGCCCACUAUCUUGGUACAUAUACGAUUUUUGGGGGCACAUCCAGUGGC (((((..((((--.......))))..(.((((((((....)))))))).).((((.(((...(.(((....))).)....)))))))..)))))..... ( -24.90) >consensus UGGAUACGUAU__UAUAUUUAUGCUCGUGCAGAUAGAUAUCUAUUUGUUCAGCACACUAUCUUCGUACAUAUACGAUUUUUGGGGGCACAUCCAGU___ .((((..((((...))))...((((((.((((((((....)))))))).)......(((...(((((....)))))....)))))))).))))...... (-19.46 = -19.78 + 0.32)

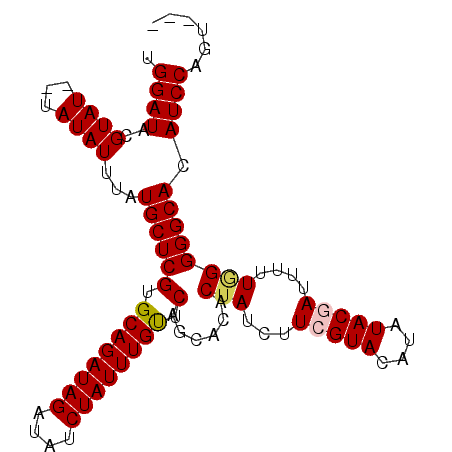
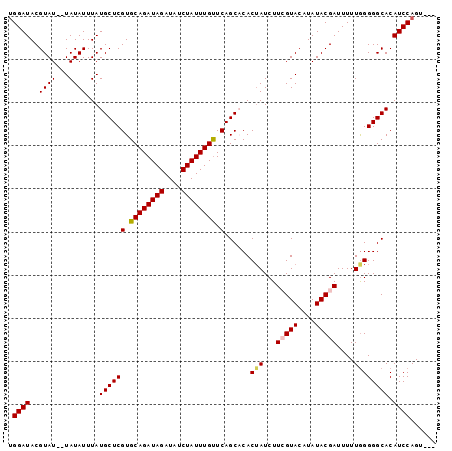
| Location | 14,426,972 – 14,427,083 |
|---|---|
| Length | 111 |
| Sequences | 5 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 94.90 |
| Mean single sequence MFE | -24.46 |
| Consensus MFE | -21.18 |
| Energy contribution | -22.06 |
| Covariance contribution | 0.88 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.52 |
| Structure conservation index | 0.87 |
| SVM decision value | 0.89 |
| SVM RNA-class probability | 0.875443 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
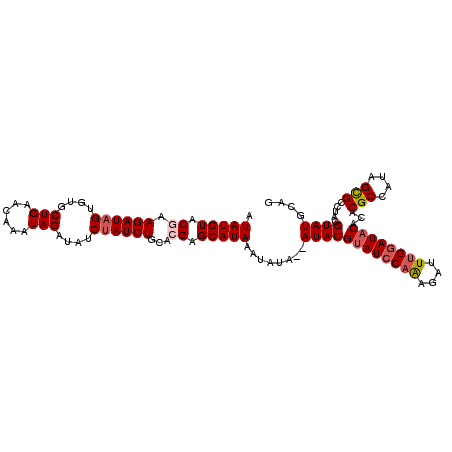
>X_DroMel_CAF1 14426972 111 + 22224390 AUAUGUACGAAGAUUGUGUGCUGAACAAAUAGAUAUCUAUCUGCACGAGCAUAAAUAUAUUAUACGUAUCCAAAGAUUUGAAUACACACGCCAUAGUGCCUACGUAUGCAG ....(((((.((....(((((((..((.((((....)))).))..).))))))............((((.(((....))).)))).(((......))).)).))))).... ( -19.30) >DroSec_CAF1 6372 109 + 1 AUAUGUACGAAGAUAGUGUGCUGAACAAAUAGAUAUCUAUCUGCACGAGCAUAAAUAUA--AUACGUAUCCCAAGAUUUGGAUUCACACGCCAUAGCGCCUACGUAUGCAG .(((((.((.((((((....(((......)))....))))))...)).)))))......--(((((((..(((.....))).......(((....)))..))))))).... ( -22.80) >DroSim_CAF1 6384 109 + 1 AUAUGUACGAAGAUAGUGUGCUGAACAAAUAGAUAUCUAUCUGCACGAGCAUAAAUAUA--AUACGUAUCCAAAGAUUUGGAUACACACGCCAUAGCGCCUACGUAUGCAG .(((((.((.((((((....(((......)))....))))))...)).)))))......--((((((((((((....))))))))...(((....))).....)))).... ( -27.20) >DroEre_CAF1 6111 109 + 1 AUAUGUACCAAGAUAGUGGGCUGAGCAAAUAGAUAUCUAUCUGCACGAGCAUAAAUAUA--AUACGUAUCCAGAGAUUUGGAUACACACGCCAUAGCGCUUACGUAUGCAG (((((((........(((.((((.(((.((((....)))).))).).))).........--....((((((((....)))))))).)))((....))...))))))).... ( -28.30) >DroYak_CAF1 6453 109 + 1 AUAUGUACCAAGAUAGUGGGCUGAACAAAUAGAUAUCUAUCUGCACGAGCAUAAAUAUA--AUACGUAUCCAAAGAUUUGGAUACACACGCCAUAGCGCCUACGUAUGCAC ...(((....((((((....(((......)))....))))))..))).(((((......--....((((((((....))))))))...(((....)))......))))).. ( -24.70) >consensus AUAUGUACGAAGAUAGUGUGCUGAACAAAUAGAUAUCUAUCUGCACGAGCAUAAAUAUA__AUACGUAUCCAAAGAUUUGGAUACACACGCCAUAGCGCCUACGUAUGCAG .(((((.((.((((((....(((......)))....))))))...)).)))))........((((((((((((....))))))))...(((....))).....)))).... (-21.18 = -22.06 + 0.88)


| Location | 14,426,972 – 14,427,083 |
|---|---|
| Length | 111 |
| Sequences | 5 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 94.90 |
| Mean single sequence MFE | -30.44 |
| Consensus MFE | -26.33 |
| Energy contribution | -27.21 |
| Covariance contribution | 0.88 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.70 |
| Structure conservation index | 0.86 |
| SVM decision value | 3.36 |
| SVM RNA-class probability | 0.999082 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
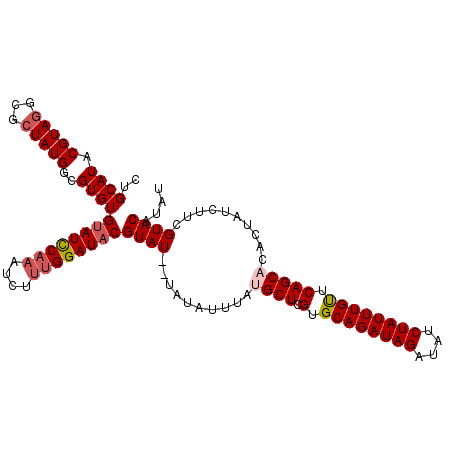
>X_DroMel_CAF1 14426972 111 - 22224390 CUGCAUACGUAGGCACUAUGGCGUGUGUAUUCAAAUCUUUGGAUACGUAUAAUAUAUUUAUGCUCGUGCAGAUAGAUAUCUAUUUGUUCAGCACACAAUCUUCGUACAUAU .....((((.((((......))((((((((((((....))))))))((((((.....))))))..(.((((((((....)))))))).).)))).....)).))))..... ( -28.40) >DroSec_CAF1 6372 109 - 1 CUGCAUACGUAGGCGCUAUGGCGUGUGAAUCCAAAUCUUGGGAUACGUAU--UAUAUUUAUGCUCGUGCAGAUAGAUAUCUAUUUGUUCAGCACACUAUCUUCGUACAUAU .....((((.((((((....))))(((.((((........))))......--........((((.(.((((((((....)))))))).))))))))...)).))))..... ( -27.30) >DroSim_CAF1 6384 109 - 1 CUGCAUACGUAGGCGCUAUGGCGUGUGUAUCCAAAUCUUUGGAUACGUAU--UAUAUUUAUGCUCGUGCAGAUAGAUAUCUAUUUGUUCAGCACACUAUCUUCGUACAUAU .....((((.(((.((....))((((((((((((....))))))))....--.........(((.(.((((((((....)))))))).))))))))..))).))))..... ( -31.50) >DroEre_CAF1 6111 109 - 1 CUGCAUACGUAAGCGCUAUGGCGUGUGUAUCCAAAUCUCUGGAUACGUAU--UAUAUUUAUGCUCGUGCAGAUAGAUAUCUAUUUGCUCAGCCCACUAUCUUGGUACAUAU ..(((((.(((.((((....))))..(((((((......)))))))....--..))).)))))..(.((((((((....)))))))).)...(((......)))....... ( -32.10) >DroYak_CAF1 6453 109 - 1 GUGCAUACGUAGGCGCUAUGGCGUGUGUAUCCAAAUCUUUGGAUACGUAU--UAUAUUUAUGCUCGUGCAGAUAGAUAUCUAUUUGUUCAGCCCACUAUCUUGGUACAUAU ((((....((((((((....))))..((((((((....))))))))....--.........(((.(.((((((((....)))))))).))))...))))....)))).... ( -32.90) >consensus CUGCAUACGUAGGCGCUAUGGCGUGUGUAUCCAAAUCUUUGGAUACGUAU__UAUAUUUAUGCUCGUGCAGAUAGAUAUCUAUUUGUUCAGCACACUAUCUUCGUACAUAU ..((((.(((((...)))))..))))((((((((....))))))))((((..........((((.(.((((((((....)))))))).)))))..........)))).... (-26.33 = -27.21 + 0.88)


| Location | 14,427,008 – 14,427,122 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 92.70 |
| Mean single sequence MFE | -22.56 |
| Consensus MFE | -20.16 |
| Energy contribution | -20.84 |
| Covariance contribution | 0.68 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.13 |
| Structure conservation index | 0.89 |
| SVM decision value | -0.03 |
| SVM RNA-class probability | 0.516476 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
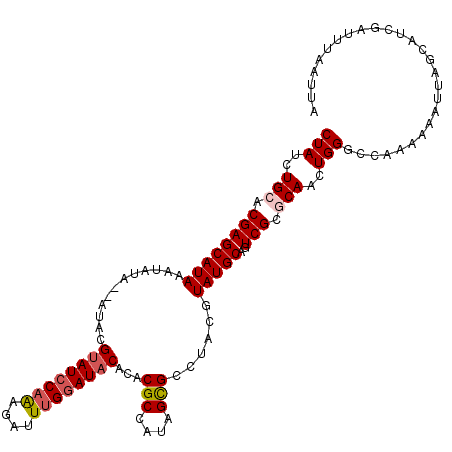
>X_DroMel_CAF1 14427008 114 + 22224390 CUAUCUGCACGAGCAUAAAUAUAUUAUACGUAUCCAAAGAUUUGAAUACACACGCCAUAGUGCCUACGUAUGCAGUCGCGCAAGUGGGCCAAAAAAUUAGCAACGAUUUAAUUA (((..(((.((((((((............((((.(((....))).)))).(((......)))......)))))..))).)))..)))........................... ( -20.60) >DroSec_CAF1 6408 112 + 1 CUAUCUGCACGAGCAUAAAUAUA--AUACGUAUCCCAAGAUUUGGAUUCACACGCCAUAGCGCCUACGUAUGCAGUCGCGCAACUGGGCCAAAAAAUUAGCAUCGAUUUAAUUA .........(((((.........--....(.((((........)))).)....((....))(((((.((.(((......))))))))))..........)).)))......... ( -18.80) >DroSim_CAF1 6420 112 + 1 CUAUCUGCACGAGCAUAAAUAUA--AUACGUAUCCAAAGAUUUGGAUACACACGCCAUAGCGCCUACGUAUGCAGUCGCGCAACUGGGCCAAAAAAUUAGCAUCGAUUUAAUUA .........(((((.........--....((((((((....))))))))....((....))(((((.((.(((......))))))))))..........)).)))......... ( -25.90) >DroEre_CAF1 6147 108 + 1 CUAUCUGCACGAGCAUAAAUAUA--AUACGUAUCCAGAGAUUUGGAUACACACGCCAUAGCGCUUACGUAUGCAGUCGCACAACUGGUCCAAAAAAUUAGCAUCGACUUA---- ............(((((......--....((((((((....))))))))...(((....)))......)))))(((((.....(((((.......)))))...)))))..---- ( -22.50) >DroYak_CAF1 6489 108 + 1 CUAUCUGCACGAGCAUAAAUAUA--AUACGUAUCCAAAGAUUUGGAUACACACGCCAUAGCGCCUACGUAUGCACUCGCACAACUGGGCCAAAAAAUUAGCAUCGAUUUA---- .........(((((.........--....((((((((....))))))))....((....))(((((.((.(((....)))..)))))))..........)).))).....---- ( -25.00) >consensus CUAUCUGCACGAGCAUAAAUAUA__AUACGUAUCCAAAGAUUUGGAUACACACGCCAUAGCGCCUACGUAUGCAGUCGCGCAACUGGGCCAAAAAAUUAGCAUCGAUUUAAUUA (((..(((.((((((((............((((((((....))))))))...(((....)))......)))))..))).)))..)))........................... (-20.16 = -20.84 + 0.68)


| Location | 14,427,008 – 14,427,122 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 92.70 |
| Mean single sequence MFE | -32.34 |
| Consensus MFE | -30.44 |
| Energy contribution | -30.88 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.10 |
| Structure conservation index | 0.94 |
| SVM decision value | 3.55 |
| SVM RNA-class probability | 0.999380 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
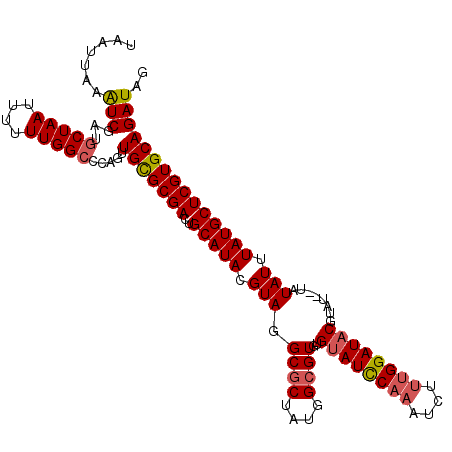
>X_DroMel_CAF1 14427008 114 - 22224390 UAAUUAAAUCGUUGCUAAUUUUUUGGCCCACUUGCGCGACUGCAUACGUAGGCACUAUGGCGUGUGUAUUCAAAUCUUUGGAUACGUAUAAUAUAUUUAUGCUCGUGCAGAUAG .......(((...(((((....))))).....(((((((((((....))))((((......))))((((((((....))))))))((((((.....)))))))))))))))).. ( -31.70) >DroSec_CAF1 6408 112 - 1 UAAUUAAAUCGAUGCUAAUUUUUUGGCCCAGUUGCGCGACUGCAUACGUAGGCGCUAUGGCGUGUGAAUCCAAAUCUUGGGAUACGUAU--UAUAUUUAUGCUCGUGCAGAUAG .......(((...(((((....))))).....(((((((..((((((((((...)))))((((((...((((.....))))))))))..--......))))))))))))))).. ( -29.70) >DroSim_CAF1 6420 112 - 1 UAAUUAAAUCGAUGCUAAUUUUUUGGCCCAGUUGCGCGACUGCAUACGUAGGCGCUAUGGCGUGUGUAUCCAAAUCUUUGGAUACGUAU--UAUAUUUAUGCUCGUGCAGAUAG .......(((...(((((....))))).....(((((((..(((((.(((.((((....))))..((((((((....))))))))....--..))).))))))))))))))).. ( -36.10) >DroEre_CAF1 6147 108 - 1 ----UAAGUCGAUGCUAAUUUUUUGGACCAGUUGUGCGACUGCAUACGUAAGCGCUAUGGCGUGUGUAUCCAAAUCUCUGGAUACGUAU--UAUAUUUAUGCUCGUGCAGAUAG ----...(((....((((....))))......((..(((..(((((.(((.((((....))))..(((((((......)))))))....--..))).))))))))..))))).. ( -30.50) >DroYak_CAF1 6489 108 - 1 ----UAAAUCGAUGCUAAUUUUUUGGCCCAGUUGUGCGAGUGCAUACGUAGGCGCUAUGGCGUGUGUAUCCAAAUCUUUGGAUACGUAU--UAUAUUUAUGCUCGUGCAGAUAG ----...(((...(((((....))))).....((..(((..(((((.(((.((((....))))..((((((((....))))))))....--..))).))))))))..))))).. ( -33.70) >consensus UAAUUAAAUCGAUGCUAAUUUUUUGGCCCAGUUGCGCGACUGCAUACGUAGGCGCUAUGGCGUGUGUAUCCAAAUCUUUGGAUACGUAU__UAUAUUUAUGCUCGUGCAGAUAG .......(((...(((((....))))).....(((((((..(((((.(((.((((....))))..((((((((....))))))))........))).))))))))))))))).. (-30.44 = -30.88 + 0.44)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:51:32 2006