
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 1,604,686 – 1,604,839 |
| Length | 153 |
| Max. P | 0.580043 |

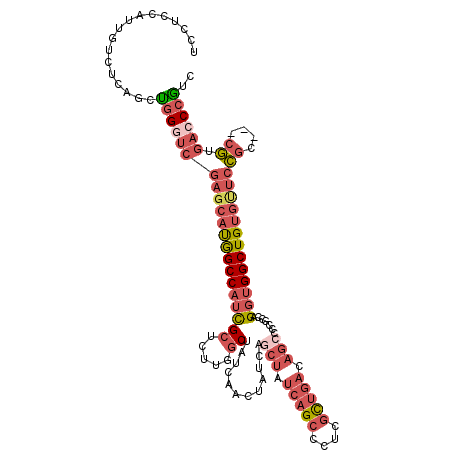
| Location | 1,604,686 – 1,604,799 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 83.63 |
| Mean single sequence MFE | -37.87 |
| Consensus MFE | -27.35 |
| Energy contribution | -29.72 |
| Covariance contribution | 2.37 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.82 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.09 |
| SVM RNA-class probability | 0.580043 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1604686 113 + 22224390 UCCUCCAUUGUCUCAGCCGGGUCGAGCAUGGCCAUCGCUCUUGGCUAUCAACUAUCAGCUAUCAGCCCUCGCUGACAGGCCCGCCAGGUGGCUGUGCUCCGC---CGUGACCCGUC .................((((((((((((((((((((((..(((.......)))..)))..(((((....)))))..((....)).)))))))))))))...---...)))))).. ( -43.10) >DroPse_CAF1 2013 107 + 1 -ACGCCAUUGUCUCAGGUCGCGGUUGCUCAGCCAUUGCCAUU-GCUAUCAGCAAUCAGCCAGCAGGCACCAUCUAC-------CUGGCCGGCUGUCUUCUACUAUCAUGACCCAUC -..............(((((.(((....(((((...((((((-((.....))))...(((....))).........-------.)))).))))).........))).))))).... ( -27.52) >DroSec_CAF1 503 113 + 1 UCCUCCAUUGUCUCAGCUGGGUCGAGCAUGGCCAUCGCUCUUGACUAUCAACUAUCAGCUAUCAGCCCUCGCUGACAGCCCCGCCAGGUGGCUGUGUUCCGC---CGUGACCCGUC ..................((((((((((((((((((((..(((.....)))......(((.(((((....))))).)))...))..)))))))))))))...---...)))))... ( -38.20) >DroSim_CAF1 16458 113 + 1 UCCUCCAUUGUCUCAGCUGGGUCGAGCAUGGCCAUCGCUCUUGGCUAUCAACUAUCAGCUAUCAGCCCUCGCUGACAGCCCCGCCAGGUGGCUGUGUUCCGC---CGUGACCCGUC ..................((((((((((((((((((((.....))............(((.(((((....))))).))).......)))))))))))))...---...)))))... ( -39.50) >DroEre_CAF1 1682 113 + 1 UCCUCCAUUGUCUCAGGUGGCUCGACCACGGCCAUCGCUCUUGGCUAUCAACUAUCAGCUAUCAGCCCUCGCUGACAGCCCCUCCAGGUGGCUGUGUUCCGC---CGUGACCCGUC .........(((...(((((...(((.(((((((((((.....))............(((.(((((....))))).))).......))))))))))))))))---)..)))..... ( -39.40) >DroYak_CAF1 1869 113 + 1 UCCUCCAUUGUCUUAGCUGGGUCGAGCAUGGCCAUCGCUCUUGGCUAUCAACUAUCAGCUAUCAGCCCUCGCUGACAGCCCCACCAGGUGGCUGUGUUCCGC---CGUGACCCGUC ..................((((((((((((((((((((.....))............(((.(((((....))))).))).......)))))))))))))...---...)))))... ( -39.50) >consensus UCCUCCAUUGUCUCAGCUGGGUCGAGCAUGGCCAUCGCUCUUGGCUAUCAACUAUCAGCUAUCAGCCCUCGCUGACAGCCCCGCCAGGUGGCUGUGUUCCGC___CGUGACCCGUC .................(((((((((((((((((((((.....))............(((.(((((....))))).))).......)))))))))))))((....)).)))))).. (-27.35 = -29.72 + 2.37)

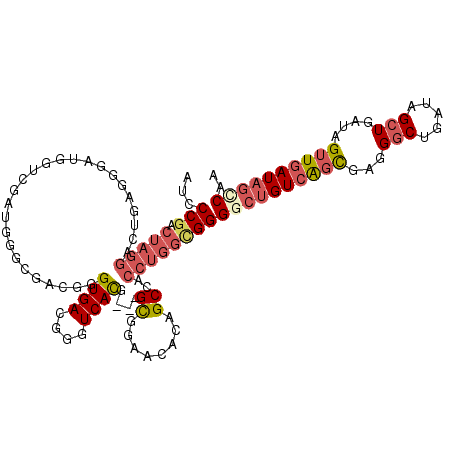
| Location | 1,604,726 – 1,604,839 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 78.80 |
| Mean single sequence MFE | -41.13 |
| Consensus MFE | -23.90 |
| Energy contribution | -25.52 |
| Covariance contribution | 1.61 |
| Combinations/Pair | 1.22 |
| Mean z-score | -1.62 |
| Structure conservation index | 0.58 |
| SVM decision value | 0.08 |
| SVM RNA-class probability | 0.574639 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
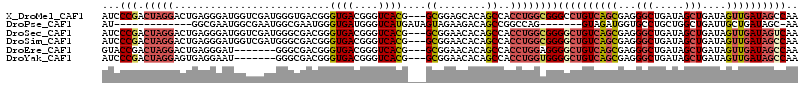
>X_DroMel_CAF1 1604726 113 - 22224390 AUCCCGACUAGGACUGAGGGAUGGUCGAUGGGUGACGGGUGACGGGUCACG---GCGGAGCACAGCCACCUGGCGGGCCUGUCAGCGAGGGCUGAUAGCUGAUAGUUGAUAGCCAA ..((((((((...(....)..)))))....(....))))(((((((((.((---.(((.(........)))).))))))))))).....(((((.((((.....)))).))))).. ( -41.80) >DroPse_CAF1 2052 95 - 1 AU-------------GGCGAAUGGCGAAUGGCGAAUGGGUGAUGGGUCAUGAUAGUAGAAGACAGCCGGCCAG-------GUAGAUGGUGCCUGCUGGCUGAUUGCUGAUAGC-AA ..-------------((((((((((..((.((......)).))..)))))............((((((((.((-------(((.....))))))))))))).)))))......-.. ( -33.00) >DroSec_CAF1 543 113 - 1 AUCCCGACUAGGACUGAGGGAUGGUCGAUGGGCGACGGGUGACGGGUCACG---GCGGAACACAGCCACCUGGCGGGGCUGUCAGCGAGGGCUGAUAGCUGAUAGUUGAUAGUCAA ..((((.(((((...........((((.....))))..((((....))))(---((........))).)))))))))(((((((((....)))))))))((((........)))). ( -45.40) >DroSim_CAF1 16498 113 - 1 AUCCCGACUAGGACUGAGGGAUGGUCGAUGGGCGACGGGUGACGGGUCACG---GCGGAACACAGCCACCUGGCGGGGCUGUCAGCGAGGGCUGAUAGCUGAUAGUUGAUAGCCAA ...(((.(((((...........((((.....))))..((((....))))(---((........))).))))))))((((((((((...(((.....)))....)))))))))).. ( -45.80) >DroEre_CAF1 1722 106 - 1 GUACCGACUAGGACUGAGGGAU-------GGGCGACGGGUGACGGGUCACG---GCGGAACACAGCCACCUGGAGGGGCUGUCAGCGAGGGCUGAUAGCUGAUAGUUGAUAGCCAA ...((..(((((..........-------.........((((....))))(---((........))).))))).))((((((((((...(((.....)))....)))))))))).. ( -38.80) >DroYak_CAF1 1909 106 - 1 AUCCCGACUAGGAGUGAGGAAU-------GGGCGACGGGUGACGGGUCACG---GCGGAACACAGCCACCUGGUGGGGCUGUCAGCGAGGGCUGAUAGCUGAUAGUUGAUAGCCAA ...((.((((((.((((.....-------.(....).(....)...))))(---((........))).))))))))((((((((((...(((.....)))....)))))))))).. ( -42.00) >consensus AUCCCGACUAGGACUGAGGGAUGGUCGAUGGGCGACGGGUGACGGGUCACG___GCGGAACACAGCCACCUGGCGGGGCUGUCAGCGAGGGCUGAUAGCUGAUAGUUGAUAGCCAA ...(((.(((((..........................((((....))))....((........))..))))))))((((((((((...(((.....)))....)))))))))).. (-23.90 = -25.52 + 1.61)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:47:37 2006