
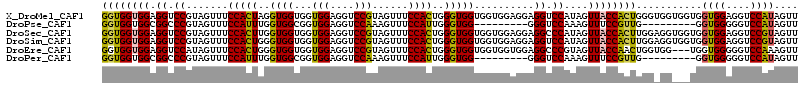
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 14,308,197 – 14,308,459 |
| Length | 262 |
| Max. P | 0.983991 |

| Location | 14,308,197 – 14,308,311 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 86.72 |
| Mean single sequence MFE | -46.08 |
| Consensus MFE | -34.64 |
| Energy contribution | -36.12 |
| Covariance contribution | 1.48 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.85 |
| Structure conservation index | 0.75 |
| SVM decision value | 1.96 |
| SVM RNA-class probability | 0.983881 |
| Prediction | RNA |
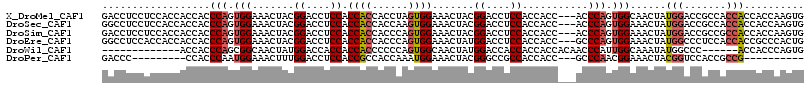
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14308197 114 + 22224390 GUGAGGGCUGCAAACUGGGGUGGUGGAGGUCCGUAAUUACCACUUGGUGGUGGUGGCGGUCCAUAGUUGCCACUGGGU---GGUGGUGGAGGUCCGUAGUUUCCACUAGGUGGUGGU ....(((((....(((.....)))...)))))...(((((((((((((((..(..((((.((....(..(((((....---)))))..).)).))))..)..))))))))))))))) ( -51.50) >DroSec_CAF1 20841 114 + 1 GUAAGGGCUGCAAACUGGGGUGGUGGAGGUCCGUAAUUACCACUUGGUGGUGGUGGCGGUCCAUAGUUUCCACUGGGU---GGUGGUGGAGGUCCGUAGUUUCCACUUGGUGGUGGU ......((..(..((..((.(((..(.((.((...(((((((((..((((..((((....)))).....))))..)))---))))))...)).))....)..)))))..)).)..)) ( -45.80) >DroSim_CAF1 18149 114 + 1 GUGAGGGCUGCAAACUGGGGUGGUGGAGGUCCGUAAUUGCCACUUGGUGGUGGCGGCGGUCCAUAGUUUCCACUGGGU---GGUGGUGGAGGUCCGUAGUUUCCACUGGGUGGUGGU ....(((((....(((.....)))...)))))...((..(((((..((((..((.((((.((......((((((....---...)))))))).)))).))..))))..)))))..)) ( -50.20) >DroEre_CAF1 17897 114 + 1 GUGAGGGCUGCGAACUGGGGUGGUGGGGGUCCGUAGAUUCCAGUGGGCGGUGGUGGAGGGCCAUAGUUUCCACUGGGC---GGUGGUGGAGGUCCAUAGUUUCCACUGGGUGGUGGU ......((..(.(.(..(((..(((((..((((..(..(((((((((..(((((.....)))))....))))))))).---..)..))))..))))).....)).)..).).)..)) ( -40.20) >DroWil_CAF1 21116 111 + 1 GUGAGGGCAGCAAAUUGCGGUGGUGGUGGUCCAUAGUUACCACUGGGUGGU------GGGCCAUAUUUGCCAAUGGGUUGUGGUGGUGGUGGUCCAUAGUUGCCACUGGGGGGUGGU .(.((((((((......(((((((..(........)..)))))))....((------(((((((..(..(((........)))..)..))))))))).)))))).)).)........ ( -42.70) >consensus GUGAGGGCUGCAAACUGGGGUGGUGGAGGUCCGUAAUUACCACUUGGUGGUGGUGGCGGUCCAUAGUUUCCACUGGGU___GGUGGUGGAGGUCCGUAGUUUCCACUGGGUGGUGGU ....(((((....(((.....)))...)))))...(((((((((.(((((.....((((.((....(..(((((.......)))))..).)).)))).....))))).))))))))) (-34.64 = -36.12 + 1.48)

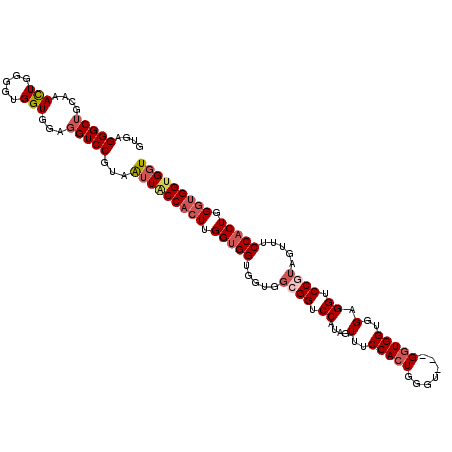

| Location | 14,308,197 – 14,308,311 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 86.72 |
| Mean single sequence MFE | -25.21 |
| Consensus MFE | -17.00 |
| Energy contribution | -18.64 |
| Covariance contribution | 1.64 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.38 |
| SVM RNA-class probability | 0.711837 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14308197 114 - 22224390 ACCACCACCUAGUGGAAACUACGGACCUCCACCACC---ACCCAGUGGCAACUAUGGACCGCCACCACCACCAAGUGGUAAUUACGGACCUCCACCACCCCAGUUUGCAGCCCUCAC .((((......)))).......(((..(((......---.....(((((...........))))).(((((...)))))......)))..)))........................ ( -24.90) >DroSec_CAF1 20841 114 - 1 ACCACCACCAAGUGGAAACUACGGACCUCCACCACC---ACCCAGUGGAAACUAUGGACCGCCACCACCACCAAGUGGUAAUUACGGACCUCCACCACCCCAGUUUGCAGCCCUUAC .((((......))))(((((..((...(((((....---.....))))).....(((((((..(((((......))))).....)))...))))...))..)))))........... ( -25.70) >DroSim_CAF1 18149 114 - 1 ACCACCACCCAGUGGAAACUACGGACCUCCACCACC---ACCCAGUGGAAACUAUGGACCGCCGCCACCACCAAGUGGCAAUUACGGACCUCCACCACCCCAGUUUGCAGCCCUCAC .((((......))))(((((..((...(((((....---.....))))).....(((......(((((......)))))......((....)).)))))..)))))........... ( -27.70) >DroEre_CAF1 17897 114 - 1 ACCACCACCCAGUGGAAACUAUGGACCUCCACCACC---GCCCAGUGGAAACUAUGGCCCUCCACCACCGCCCACUGGAAUCUACGGACCCCCACCACCCCAGUUCGCAGCCCUCAC ...........(((((..((((((...(((((....---.....)))))..))))))...)))))....((..(((((.......((....))......)))))..))......... ( -25.72) >DroWil_CAF1 21116 111 - 1 ACCACCCCCCAGUGGCAACUAUGGACCACCACCACCACAACCCAUUGGCAAAUAUGGCCC------ACCACCCAGUGGUAACUAUGGACCACCACCACCGCAAUUUGCUGCCCUCAC ..........((.((((....(((.(((.....(((((........(((.......))).------........))))).....))).)))........((.....))))))))... ( -22.03) >consensus ACCACCACCCAGUGGAAACUACGGACCUCCACCACC___ACCCAGUGGAAACUAUGGACCGCCACCACCACCAAGUGGUAAUUACGGACCUCCACCACCCCAGUUUGCAGCCCUCAC .((((......))))(((((..((.......((((.........)))).......((.(((.....(((((...))))).....))).)).......))..)))))........... (-17.00 = -18.64 + 1.64)


| Location | 14,308,237 – 14,308,351 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 78.39 |
| Mean single sequence MFE | -44.50 |
| Consensus MFE | -27.25 |
| Energy contribution | -29.00 |
| Covariance contribution | 1.75 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.61 |
| SVM decision value | 1.50 |
| SVM RNA-class probability | 0.959416 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14308237 114 + 22224390 CACUUGGUGGUGGUGGCGGUCCAUAGUUGCCACUGGGU---GGUGGUGGAGGUCCGUAGUUUCCACUAGGUGGUGGUGGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGAGGAGGUC ((((..((((..(..((((.((...(..((((((.(((---((..((((....)))).....))))).))))))..)...)).))))..)..))))..))))............... ( -46.90) >DroSec_CAF1 20881 114 + 1 CACUUGGUGGUGGUGGCGGUCCAUAGUUUCCACUGGGU---GGUGGUGGAGGUCCGUAGUUUCCACUUGGUGGUGGUGGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGAGGAGGCC ((((..((((..(..(((((((((.....((((..(((---((..((((....)))).....)))))..))))..)))))...))))..)..))))..))))..(((.......))) ( -47.00) >DroSim_CAF1 18189 114 + 1 CACUUGGUGGUGGCGGCGGUCCAUAGUUUCCACUGGGU---GGUGGUGGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGAGGAGGUC ((((..((((..((.(((((((((.....(((((.(((---((..((((....)))).....))))).)))))..)))))...)))).))..))))..))))............... ( -48.30) >DroEre_CAF1 17937 114 + 1 CAGUGGGCGGUGGUGGAGGGCCAUAGUUUCCACUGGGC---GGUGGUGGAGGUCCAUAGUUUCCACUGGGUGGUGGUGGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGUGGAGGCC (((((((..(((((.....)))))....))))))).((---((..((((....))))..((((((((.......)))))))).)))).((((((((((........)))))))))). ( -43.50) >DroWil_CAF1 21156 98 + 1 CACUGGGUGGU------GGGCCAUAUUUGCCAAUGGGUUGUGGUGGUGGUGGUCCAUAGUUGCCACUGGGGGGUGGUGGUGGUCCAUAGUUGCCGCUGGGUGGU------------- ((((.(((((.------.((((((....(((....)))....))))).((((.((((....((((((....)))))).)))).))))..)..))))).))))..------------- ( -45.00) >DroPer_CAF1 25680 95 + 1 ----------CGGCGGUGGACCGUAGUUUCCGUUGGGC---GGUGGUGGCGGCCCGUAGUUUCCAUUUGGUGGCGGUGGAGGUCCAAAGUUUCCAUUGGGUGG---------GGGUC ----------.(((((.((((....)))))))))((((---.((....)).))))....(..(((((..(((((..(((....)))..)...))))..)))))---------..).. ( -36.30) >consensus CACUUGGUGGUGGUGGCGGUCCAUAGUUUCCACUGGGU___GGUGGUGGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGAGGAGGUC ((((..((((.....((((.((....(((((((((........))))))))).........((((((.......)))))))).)))).....))))..))))............... (-27.25 = -29.00 + 1.75)



| Location | 14,308,237 – 14,308,351 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 78.39 |
| Mean single sequence MFE | -24.23 |
| Consensus MFE | -13.93 |
| Energy contribution | -14.35 |
| Covariance contribution | 0.42 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.56 |
| SVM RNA-class probability | 0.780734 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14308237 114 - 22224390 GACCUCCUCCACCACCACCCAGUGGAAACUACGGACCUCCACCACCACCUAGUGGAAACUACGGACCUCCACCACC---ACCCAGUGGCAACUAUGGACCGCCACCACCACCAAGUG ...............(((...((((......(((...((((((((......)))).......((....)).((((.---.....))))......)))))))......))))...))) ( -23.30) >DroSec_CAF1 20881 114 - 1 GGCCUCCUCCACCACCACCCAGUGGAAACUACGGACCUCCACCACCACCAAGUGGAAACUACGGACCUCCACCACC---ACCCAGUGGAAACUAUGGACCGCCACCACCACCAAGUG (((.(((.......((((...((((.......((....))...........(((((..(....)...)))))..))---))...)))).......)))..)))..(((......))) ( -27.54) >DroSim_CAF1 18189 114 - 1 GACCUCCUCCACCACCACCCAGUGGAAACUACGGACCUCCACCACCACCCAGUGGAAACUACGGACCUCCACCACC---ACCCAGUGGAAACUAUGGACCGCCGCCACCACCAAGUG ...............(((...((((......(((...((((((((......)))).......((....)).((((.---.....))))......))))...)))...))))...))) ( -24.90) >DroEre_CAF1 17937 114 - 1 GGCCUCCACCACCACCACCCAGUGGAAACUACGGACCUCCACCACCACCCAGUGGAAACUAUGGACCUCCACCACC---GCCCAGUGGAAACUAUGGCCCUCCACCACCGCCCACUG (((........((((......)))).......(((..((((((((......))))......))))..)))......---)))((((((......(((.......)))....)))))) ( -28.90) >DroWil_CAF1 21156 98 - 1 -------------ACCACCCAGCGGCAACUAUGGACCACCACCACCCCCCAGUGGCAACUAUGGACCACCACCACCACAACCCAUUGGCAAAUAUGGCCC------ACCACCCAGUG -------------..((((((..(....)..)))....(((.......(((((((......(((.......))).......)))))))......)))...------........))) ( -17.04) >DroPer_CAF1 25680 95 - 1 GACCC---------CCACCCAAUGGAAACUUUGGACCUCCACCGCCACCAAAUGGAAACUACGGGCCGCCACCACC---GCCCAACGGAAACUACGGUCCACCGCCG---------- ((((.---------....((((.(....).)))).......((((((.....))).......((((.(......).---))))..))).......))))........---------- ( -23.70) >consensus GACCUCCUCCACCACCACCCAGUGGAAACUACGGACCUCCACCACCACCCAGUGGAAACUACGGACCUCCACCACC___ACCCAGUGGAAACUAUGGACCGCCACCACCACCAAGUG ..................(((.(((.......((....)).((((......)))).......((....))...........))).)))......(((.......))).......... (-13.93 = -14.35 + 0.42)
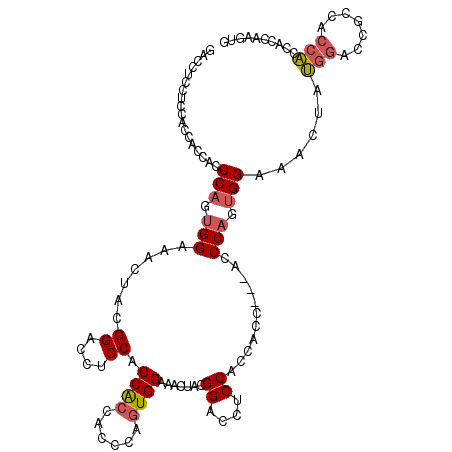
| Location | 14,308,275 – 14,308,391 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 81.47 |
| Mean single sequence MFE | -39.44 |
| Consensus MFE | -27.96 |
| Energy contribution | -27.21 |
| Covariance contribution | -0.75 |
| Combinations/Pair | 1.25 |
| Mean z-score | -1.63 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.35 |
| SVM RNA-class probability | 0.701906 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14308275 116 + 22224390 GGUGGUGGAGGUCCGUAGUUUCCACUAGGUGGUGGUGGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGAGGAGGUCCAUAGUUACCACUGGGUGGUGGUGGUGGAGGUCCAUAGUU ....(((((..(((((..(..(((((.((((((((((((..(((....(..(((((....)))))..).)))..)))))...))))))).)))))..)..)))))..))))).... ( -46.10) >DroPse_CAF1 26796 98 + 1 GGUGGUGGCGGCCCGUAGUUUCCAUUUGGUGGCGGUGGAGGUCCAAAGUUUCCAUUGGGUGG---------GGGUCCAAAGUUUCCGUUG---------GGUGGGGGUCCAUAGUU ....((((..((((.((.(..(((((..(((((..(((....)))..)...))))..)))))---------..)(((((........)))---------)))).)))))))).... ( -33.60) >DroSec_CAF1 20919 116 + 1 GGUGGUGGAGGUCCGUAGUUUCCACUUGGUGGUGGUGGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGAGGAGGCCCAUAGUUACCACUUGGAGGUGGUGGUGGAGGUCCGUAGUU ......(((..(((((.....((((((...((((((((.((.((....((((((((.......)))))))).)).))....))))))))...))))))..)))))..)))...... ( -39.70) >DroSim_CAF1 18227 116 + 1 GGUGGUGGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGAGGAGGUCCAUAGUUACCACUUGGAGGUGGUGGUGGAGGUCCGUAGUU ......(((..(((((..(..((.(..((((((((((((..(((....(..(((((....)))))..).)))..)))))...)))))))..).))..)..)))))..)))...... ( -42.10) >DroEre_CAF1 17975 113 + 1 GGUGGUGGAGGUCCAUAGUUUCCACUGGGUGGUGGUGGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGUGGAGGCCCGUAGUUACCAACUGGUGG---UGGUGGGGGUCCAAAGUU .....((((..(((((.....((((..(.((((..(...((.((.......(((((....))))).......)).))...)..)))).)..))))---..)))))..))))..... ( -41.54) >DroPer_CAF1 25708 98 + 1 GGUGGUGGCGGCCCGUAGUUUCCAUUUGGUGGCGGUGGAGGUCCAAAGUUUCCAUUGGGUGG---------GGGUCCAAAGUUUCCGUUG---------GGUGGGGGUCCAUAGUU ....((((..((((.((.(..(((((..(((((..(((....)))..)...))))..)))))---------..)(((((........)))---------)))).)))))))).... ( -33.60) >consensus GGUGGUGGAGGUCCGUAGUUUCCACUUGGUGGUGGUGGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGAGGAGGUCCAUAGUUACCACUGGG_GG___UGGUGGAGGUCCAUAGUU ((((((((.((.((.......(((((..((((...(((....)))......))))..)))))..........)).))....))))))))...........((((....)))).... (-27.96 = -27.21 + -0.75)


| Location | 14,308,275 – 14,308,391 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 81.47 |
| Mean single sequence MFE | -27.27 |
| Consensus MFE | -16.06 |
| Energy contribution | -16.12 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.11 |
| Mean z-score | -3.59 |
| Structure conservation index | 0.59 |
| SVM decision value | 1.43 |
| SVM RNA-class probability | 0.953019 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14308275 116 - 22224390 AACUAUGGACCUCCACCACCACCACCCAGUGGUAACUAUGGACCUCCUCCACCACCACCCAGUGGAAACUACGGACCUCCACCACCACCUAGUGGAAACUACGGACCUCCACCACC .....((((..(((...(((((......))))).....((((..((((((((.........)))))......)))..))))((((......)))).......)))..))))..... ( -30.60) >DroPse_CAF1 26796 98 - 1 AACUAUGGACCCCCACC---------CAACGGAAACUUUGGACCC---------CCACCCAAUGGAAACUUUGGACCUCCACCGCCACCAAAUGGAAACUACGGGCCGCCACCACC .....(((...(((..(---------(((.(....).))))....---------....((((.(....).))))...((((...........))))......)))...)))..... ( -22.90) >DroSec_CAF1 20919 116 - 1 AACUACGGACCUCCACCACCACCUCCAAGUGGUAACUAUGGGCCUCCUCCACCACCACCCAGUGGAAACUACGGACCUCCACCACCACCAAGUGGAAACUACGGACCUCCACCACC ......(((..(((...(((((......))))).....((((..((((((((.........)))))......)))..))))((((......)))).......)))..)))...... ( -29.30) >DroSim_CAF1 18227 116 - 1 AACUACGGACCUCCACCACCACCUCCAAGUGGUAACUAUGGACCUCCUCCACCACCACCCAGUGGAAACUACGGACCUCCACCACCACCCAGUGGAAACUACGGACCUCCACCACC ......(((..(((...(((((......))))).....((((..((((((((.........)))))......)))..))))((((......)))).......)))..)))...... ( -30.20) >DroEre_CAF1 17975 113 - 1 AACUUUGGACCCCCACCA---CCACCAGUUGGUAACUACGGGCCUCCACCACCACCACCCAGUGGAAACUACGGACCUCCACCACCACCCAGUGGAAACUAUGGACCUCCACCACC .....((((..(((....---..(((....)))......)))..))))...((((......)))).......(((..((((((((......))))......))))..)))...... ( -27.70) >DroPer_CAF1 25708 98 - 1 AACUAUGGACCCCCACC---------CAACGGAAACUUUGGACCC---------CCACCCAAUGGAAACUUUGGACCUCCACCGCCACCAAAUGGAAACUACGGGCCGCCACCACC .....(((...(((..(---------(((.(....).))))....---------....((((.(....).))))...((((...........))))......)))...)))..... ( -22.90) >consensus AACUAUGGACCCCCACCA___CC_CCAAGUGGUAACUAUGGACCUCCUCCACCACCACCCAGUGGAAACUACGGACCUCCACCACCACCAAGUGGAAACUACGGACCUCCACCACC ......(((..(((...............((((.....................(((.....))).......((....))))))(((.....))).......)))..)))...... (-16.06 = -16.12 + 0.06)


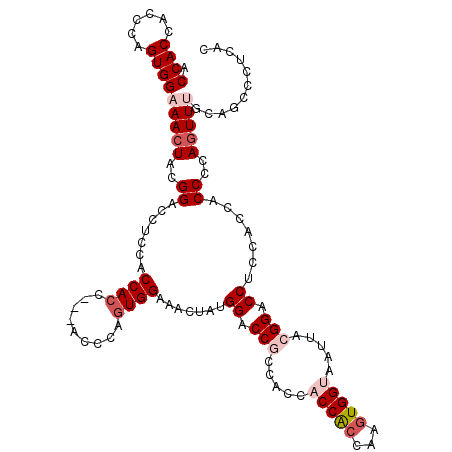
| Location | 14,308,311 – 14,308,419 |
|---|---|
| Length | 108 |
| Sequences | 4 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 90.74 |
| Mean single sequence MFE | -49.15 |
| Consensus MFE | -44.51 |
| Energy contribution | -45.20 |
| Covariance contribution | 0.69 |
| Combinations/Pair | 1.12 |
| Mean z-score | -4.93 |
| Structure conservation index | 0.91 |
| SVM decision value | 1.96 |
| SVM RNA-class probability | 0.983991 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14308311 108 + 22224390 GGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGAGGAGGUCCAUAGUUACCACUGGGUGGUGGUGGUGGAGGUCCAUAGUUACCACUUGGUGGUGGUGGUGGAGGUCCA ...((.((....((((((((.......))))))))...(((((..(((((((..((((((((..((((....))))..))))))))..)))))))..))))))).)). ( -54.00) >DroSec_CAF1 20955 108 + 1 GGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGAGGAGGCCCAUAGUUACCACUUGGAGGUGGUGGUGGAGGUCCGUAGUUACCACUUGGUGGUGGUGGUGGAGGUCCG (((..(((((..(..((((..((((((..(...(((...((((...(((((((....)))))))))))...)))...)..))))))..))))..)..)))))..))). ( -47.80) >DroSim_CAF1 18263 108 + 1 GGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGAGGAGGUCCAUAGUUACCACUUGGAGGUGGUGGUGGAGGUCCGUAGUUACCACUUGGUGGUGGUGGUGGAGGUCCG (((..(((((..(..((((..((((((..(...(((..(((((...(((((((....))))))))))))..)))...)..))))))..))))..)..)))))..))). ( -51.50) >DroEre_CAF1 18011 102 + 1 GGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGUGGAGGCCCGUAGUUACCAACUGGUGG---UGGUGGGGGUCCAAAGUUACCAAUUGGUGGU---GGUGGGGGUCCG ...(((((.((.(..((((..(.((((..(..((((..((((..(..(((....)))..---)..))))..))))..)..)))).)..)))).---).)).))).)). ( -43.30) >consensus GGAGGUCCGUAGUUUCCACUGGGUGGUGGUGGAGGAGGCCCAUAGUUACCACUUGGAGGUGGUGGUGGAGGUCCAUAGUUACCACUUGGUGGUGGUGGUGGAGGUCCG (((..(((((.....((((..((((((..(...(((..(((((...(((((((....))))))))))))..)))...)..))))))..)))).....)))))..))). (-44.51 = -45.20 + 0.69)


| Location | 14,308,311 – 14,308,419 |
|---|---|
| Length | 108 |
| Sequences | 4 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 90.74 |
| Mean single sequence MFE | -30.16 |
| Consensus MFE | -24.80 |
| Energy contribution | -25.11 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.08 |
| Mean z-score | -4.45 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.58 |
| SVM RNA-class probability | 0.965643 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14308311 108 - 22224390 UGGACCUCCACCACCACCACCAAGUGGUAACUAUGGACCUCCACCACCACCACCCAGUGGUAACUAUGGACCUCCUCCACCACCACCCAGUGGAAACUACGGACCUCC .(((..(((.......((((...(((((......(((..((((..(((((......))))).....))))..)))......)))))...)))).......)))..))) ( -30.64) >DroSec_CAF1 20955 108 - 1 CGGACCUCCACCACCACCACCAAGUGGUAACUACGGACCUCCACCACCACCUCCAAGUGGUAACUAUGGGCCUCCUCCACCACCACCCAGUGGAAACUACGGACCUCC .(((..(((...(((((......)))))......(((..((((..(((((......))))).....))))..)))(((((.........)))))......)))..))) ( -29.60) >DroSim_CAF1 18263 108 - 1 CGGACCUCCACCACCACCACCAAGUGGUAACUACGGACCUCCACCACCACCUCCAAGUGGUAACUAUGGACCUCCUCCACCACCACCCAGUGGAAACUACGGACCUCC .(((..(((...(((((......)))))......(((..((((..(((((......))))).....))))..)))(((((.........)))))......)))..))) ( -30.50) >DroEre_CAF1 18011 102 - 1 CGGACCCCCACC---ACCACCAAUUGGUAACUUUGGACCCCCACCA---CCACCAGUUGGUAACUACGGGCCUCCACCACCACCACCCAGUGGAAACUACGGACCUCC .(((..(((...---...((((((((((.....(((....)))...---..))))))))))......)))..)))....((((......))))...(....)...... ( -29.90) >consensus CGGACCUCCACCACCACCACCAAGUGGUAACUACGGACCUCCACCACCACCACCAAGUGGUAACUAUGGACCUCCUCCACCACCACCCAGUGGAAACUACGGACCUCC .(((..(((.......((((...(((((......(((..((((((((.........)))))......)))..)))......)))))...)))).......)))..))) (-24.80 = -25.11 + 0.31)



| Location | 14,308,351 – 14,308,459 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 75.14 |
| Mean single sequence MFE | -40.28 |
| Consensus MFE | -21.65 |
| Energy contribution | -22.02 |
| Covariance contribution | 0.37 |
| Combinations/Pair | 1.25 |
| Mean z-score | -2.14 |
| Structure conservation index | 0.54 |
| SVM decision value | 1.40 |
| SVM RNA-class probability | 0.949867 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14308351 108 + 22224390 CAUAGUUACCACUGGGUGGUGGUGGUGGAGGUCCAUAGUUACCACUUGGUGGUGGUGGUGGAGGUCCA---GAGUGUCCGAUAGUCUGGUACUUCUGGGCGUGCACAGAUC .....(((((((..((((((((..((((....))))..))))))))..)))))))..(((.(.(((((---(((((.(((......)))))).))))))).).)))..... ( -52.60) >DroSec_CAF1 20995 108 + 1 CAUAGUUACCACUUGGAGGUGGUGGUGGAGGUCCGUAGUUACCACUUGGUGGUGGUGGUGGAGGUCCG---GAGUGUCCGAUAGUCUGGUACUUCUGGGCGUGCACAGAAC ......(((((((....)))))))(((.(.((((....((((((((....)))))))).(((((((((---((.(((...))).))))).)))))))))).).)))..... ( -43.90) >DroSim_CAF1 18303 108 + 1 CAUAGUUACCACUUGGAGGUGGUGGUGGAGGUCCGUAGUUACCACUUGGUGGUGGUGGUGGAGGUCCG---GAGUGUCCGAUAGUCUGGUACUUCUGGGCGUGCACAGAAC ......(((((((....)))))))(((.(.((((....((((((((....)))))))).(((((((((---((.(((...))).))))).)))))))))).).)))..... ( -43.90) >DroEre_CAF1 18051 102 + 1 CGUAGUUACCAACUGGUGG---UGGUGGGGGUCCAAAGUUACCAAUUGGUGGU---GGUGGGGGUCCG---GAGUGUCCGAUGGUCUGGUACUUCUGGGCAUGCACAGAGC .....(((((((.((((..---(..(((....)))..)..)))).))))))).---.(((.(.(((((---(((((.(((......)))))).))))))).).)))..... ( -37.00) >DroWil_CAF1 21254 82 + 1 -----------------------GGUGGUGGUCCAUAGUUACCACUGGGUGGUGGCGGUGG---CAAA---GAGUGUCCGAUAGUCUGGUACUUUUGAGCGUGCACCGAGC -----------------------..((((((.((((.((((((((...)))))))).))))---).((---(((((.(((......)))))))))).......)))))... ( -28.60) >DroAna_CAF1 84576 92 + 1 -------------------UGGUGGCGGAGGGCCGUAAUUGCCACUGGGCGGCGGAGGUGGAGGUCCAGCCGAGUGGCCAAUGGUCUGGUACUUCUGAGCAUGCACCGAGC -------------------.((((.((((((((((..(((((((((.(((...(((........))).))).))))).))))....)))).))))))......)))).... ( -35.70) >consensus CAUAGUUACCACUUGG_GGUGGUGGUGGAGGUCCAUAGUUACCACUUGGUGGUGGUGGUGGAGGUCCA___GAGUGUCCGAUAGUCUGGUACUUCUGGGCGUGCACAGAGC ........................((((....))))..(((((((...)))))))..(((.(.(((((...(((((.(((......)))))))).))))).).)))..... (-21.65 = -22.02 + 0.37)

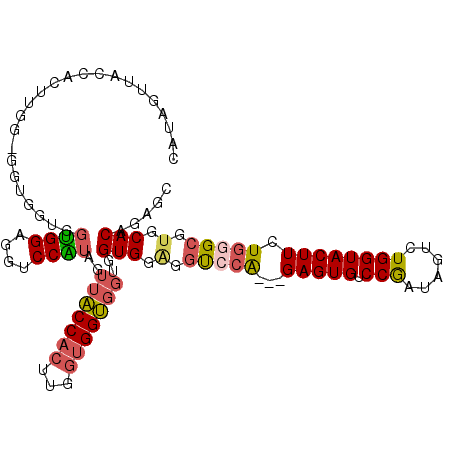
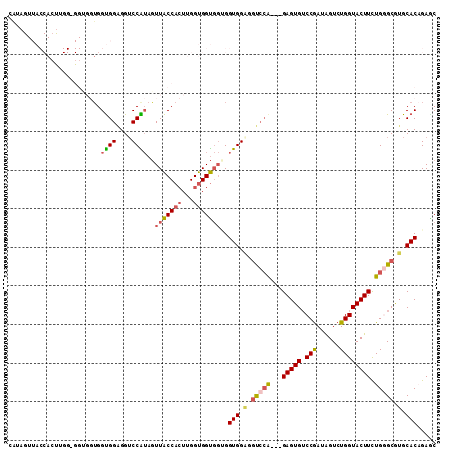
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:50:15 2006