
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 14,045,469 – 14,045,600 |
| Length | 131 |
| Max. P | 0.999932 |

| Location | 14,045,469 – 14,045,560 |
|---|---|
| Length | 91 |
| Sequences | 5 |
| Columns | 101 |
| Reading direction | forward |
| Mean pairwise identity | 86.84 |
| Mean single sequence MFE | -32.18 |
| Consensus MFE | -28.74 |
| Energy contribution | -28.98 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.04 |
| Mean z-score | -3.69 |
| Structure conservation index | 0.89 |
| SVM decision value | 4.36 |
| SVM RNA-class probability | 0.999880 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14045469 91 + 22224390 ----------GCGCUCUUCGCUCUGCUCACGCACUGUUAACCGAACGUUAACAGUGCAAGAGAGCGCCAGCUCUCAAUGUUGCAUUUGUUUACAGUACAUU ----------(..((.(..((..(((..(((((((((((((.....)))))))))))..((((((....))))))...)).)))...))..).))..)... ( -32.20) >DroSec_CAF1 14208 91 + 1 ----------GCGCUCUUCGCUCUGCUCACGCACUGUUAACCGAACGUUAACAGUGCAAGAGAGCGCCAGCUCUCAAUGUUGCAUUUGUUUACAGUACAUU ----------(..((.(..((..(((..(((((((((((((.....)))))))))))..((((((....))))))...)).)))...))..).))..)... ( -32.20) >DroSim_CAF1 14565 91 + 1 ----------GCGCUCUUCGCUCUGCUCACGCACUGUUAACCGAACGUUAACAGUGCAAGAGAGCGCCAGCUCUCCAUGUUGCAUUUGUUUACAGUACAUU ----------(..((.(..((..(((..(((((((((((((.....)))))))))))..((((((....))))))...)).)))...))..).))..)... ( -31.30) >DroEre_CAF1 14418 101 + 1 CUCGCCAUCGGCGCUCUUCGCUCCGCUCACGCACUGUUAACCCAGCGUUAACAGUGUGAGAGAGCGCCAGCACACAAUGUUGCAUAUCGUUACAGUACAUU .........(((((((((.((...))...((((((((((((.....)))))))))))).))))))))).(((........))).................. ( -35.70) >DroYak_CAF1 14181 101 + 1 CUCUCCAUCGCCGCUCUUCGCUCUGCUCACGCACUGUUAACCGAGCAUUAACAGUGCGAGAGAGCGUCAGCUUACAAUGUUGCAUUUCUUUACAGUACAUU ............(((...((((((.(((..((((((((((.......)))))))))))))))))))..)))....(((((.((...........))))))) ( -29.50) >consensus __________GCGCUCUUCGCUCUGCUCACGCACUGUUAACCGAACGUUAACAGUGCAAGAGAGCGCCAGCUCUCAAUGUUGCAUUUGUUUACAGUACAUU ..........((((((((.((...))....(((((((((((.....)))))))))))..))))))))((((.......))))................... (-28.74 = -28.98 + 0.24)


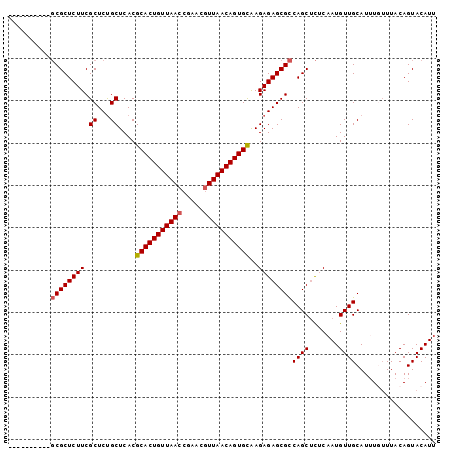
| Location | 14,045,469 – 14,045,560 |
|---|---|
| Length | 91 |
| Sequences | 5 |
| Columns | 101 |
| Reading direction | reverse |
| Mean pairwise identity | 86.84 |
| Mean single sequence MFE | -36.88 |
| Consensus MFE | -34.04 |
| Energy contribution | -33.88 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.72 |
| Structure conservation index | 0.92 |
| SVM decision value | 4.64 |
| SVM RNA-class probability | 0.999932 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14045469 91 - 22224390 AAUGUACUGUAAACAAAUGCAACAUUGAGAGCUGGCGCUCUCUUGCACUGUUAACGUUCGGUUAACAGUGCGUGAGCAGAGCGAAGAGCGC---------- (((((..((((......)))))))))....(((..((((((((((((((((((((.....)))))))))))..))).))))))...)))..---------- ( -35.20) >DroSec_CAF1 14208 91 - 1 AAUGUACUGUAAACAAAUGCAACAUUGAGAGCUGGCGCUCUCUUGCACUGUUAACGUUCGGUUAACAGUGCGUGAGCAGAGCGAAGAGCGC---------- (((((..((((......)))))))))....(((..((((((((((((((((((((.....)))))))))))..))).))))))...)))..---------- ( -35.20) >DroSim_CAF1 14565 91 - 1 AAUGUACUGUAAACAAAUGCAACAUGGAGAGCUGGCGCUCUCUUGCACUGUUAACGUUCGGUUAACAGUGCGUGAGCAGAGCGAAGAGCGC---------- ......((((..((...........(((((((....))))))).(((((((((((.....)))))))))))))..)))).((.....))..---------- ( -35.30) >DroEre_CAF1 14418 101 - 1 AAUGUACUGUAACGAUAUGCAACAUUGUGUGCUGGCGCUCUCUCACACUGUUAACGCUGGGUUAACAGUGCGUGAGCGGAGCGAAGAGCGCCGAUGGCGAG ..(((..((((......)))).(((((.(((((..((((((((((((((((((((.....)))))))))..))))).))))))...))))))))))))).. ( -41.60) >DroYak_CAF1 14181 101 - 1 AAUGUACUGUAAAGAAAUGCAACAUUGUAAGCUGACGCUCUCUCGCACUGUUAAUGCUCGGUUAACAGUGCGUGAGCAGAGCGAAGAGCGGCGAUGGAGAG .......((((......)))).((((((..(((..((((((((((((((((((((.....)))))))))))..))).))))))...))).))))))..... ( -37.10) >consensus AAUGUACUGUAAACAAAUGCAACAUUGAGAGCUGGCGCUCUCUUGCACUGUUAACGUUCGGUUAACAGUGCGUGAGCAGAGCGAAGAGCGC__________ (((((..((((......)))))))))....(((..((((((((((((((((((((.....)))))))))))..))).))))))...)))............ (-34.04 = -33.88 + -0.16)


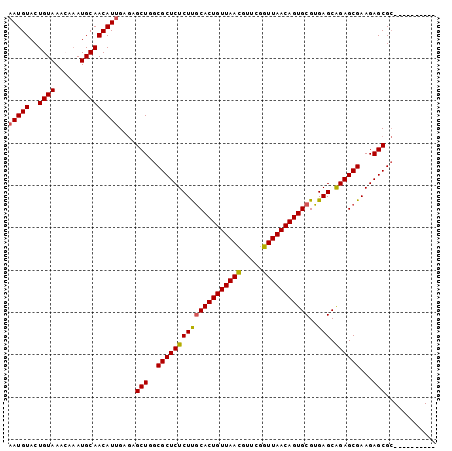
| Location | 14,045,482 – 14,045,600 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 82.54 |
| Mean single sequence MFE | -39.72 |
| Consensus MFE | -32.72 |
| Energy contribution | -33.60 |
| Covariance contribution | 0.88 |
| Combinations/Pair | 1.07 |
| Mean z-score | -4.06 |
| Structure conservation index | 0.82 |
| SVM decision value | 3.63 |
| SVM RNA-class probability | 0.999470 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14045482 118 + 22224390 UGCUCACGCACUGUUAACCGAACGUUAACAGUGCAAGAGAGCGCCAGCUCUCAAUGUUGCAUUUGUUUACAGUACAUUGGCCGAAAUGCAGCAACAUAUGCAAUGAAUUCAGGAUUUA (((....(((((((((((.....)))))))))))..((((((....))))))..((((((((((.....(((....)))....))))))))))......)))................ ( -40.80) >DroSec_CAF1 14221 118 + 1 UGCUCACGCACUGUUAACCGAACGUUAACAGUGCAAGAGAGCGCCAGCUCUCAAUGUUGCAUUUGUUUACAGUACAUUGGCCGAAAUGCAGCAACAUAUGCAAUGAAUUCACGGUUUC (((....(((((((((((.....)))))))))))..((((((....))))))..((((((((((.....(((....)))....))))))))))......)))................ ( -40.80) >DroSim_CAF1 14578 118 + 1 UGCUCACGCACUGUUAACCGAACGUUAACAGUGCAAGAGAGCGCCAGCUCUCCAUGUUGCAUUUGUUUACAGUACAUUGGCCGAAAUGCAGCAACAUAUGCAAUGAAUUCACGAUUUC (((....(((((((((((.....)))))))))))..((((((....))))))..((((((((((.....(((....)))....))))))))))......)))................ ( -39.90) >DroEre_CAF1 14441 92 + 1 CGCUCACGCACUGUUAACCCAGCGUUAACAGUGUGAGAGAGCGCCAGCACACAAUGUUGCAUAUCGUUACAGUACAUUGGUCGAAAUGCAGC-------------------------- (((((.((((((((((((.....))))))))))))...)))))............(((((((.(((...(((....)))..))).)))))))-------------------------- ( -35.00) >DroYak_CAF1 14204 118 + 1 UGCUCACGCACUGUUAACCGAGCAUUAACAGUGCGAGAGAGCGUCAGCUUACAAUGUUGCAUUUCUUUACAGUACAUUGGGCGAAAUGCAGCAGUACAUGCAAUGACUUUACGAUGCA ..(((.(((((((((((.......))))))))))).))).(((((.((......(((((((((((....(((....)))...)))))))))))......))...(......)))))). ( -42.10) >consensus UGCUCACGCACUGUUAACCGAACGUUAACAGUGCAAGAGAGCGCCAGCUCUCAAUGUUGCAUUUGUUUACAGUACAUUGGCCGAAAUGCAGCAACAUAUGCAAUGAAUUCACGAUUUA .......(((((((((((.....)))))))))))..((((((....))))))..((((((((((.....(((....)))....))))))))))......................... (-32.72 = -33.60 + 0.88)


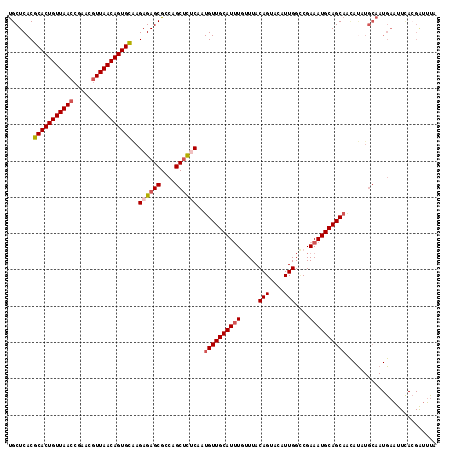
| Location | 14,045,482 – 14,045,600 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 82.54 |
| Mean single sequence MFE | -39.04 |
| Consensus MFE | -31.50 |
| Energy contribution | -32.54 |
| Covariance contribution | 1.04 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.27 |
| Structure conservation index | 0.81 |
| SVM decision value | 3.08 |
| SVM RNA-class probability | 0.998382 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 14045482 118 - 22224390 UAAAUCCUGAAUUCAUUGCAUAUGUUGCUGCAUUUCGGCCAAUGUACUGUAAACAAAUGCAACAUUGAGAGCUGGCGCUCUCUUGCACUGUUAACGUUCGGUUAACAGUGCGUGAGCA .....((.(..((((((((((.((((((((((((......))))))..)).)))).))))))...))))..).)).((((...((((((((((((.....)))))))))))).)))). ( -40.10) >DroSec_CAF1 14221 118 - 1 GAAACCGUGAAUUCAUUGCAUAUGUUGCUGCAUUUCGGCCAAUGUACUGUAAACAAAUGCAACAUUGAGAGCUGGCGCUCUCUUGCACUGUUAACGUUCGGUUAACAGUGCGUGAGCA ....(((.(..((((((((((.((((((((((((......))))))..)).)))).))))))...))))..)))).((((...((((((((((((.....)))))))))))).)))). ( -40.60) >DroSim_CAF1 14578 118 - 1 GAAAUCGUGAAUUCAUUGCAUAUGUUGCUGCAUUUCGGCCAAUGUACUGUAAACAAAUGCAACAUGGAGAGCUGGCGCUCUCUUGCACUGUUAACGUUCGGUUAACAGUGCGUGAGCA ......((.......((((((.((((((((((((......))))))..)).)))).))))))((((((((((....))))))).(((((((((((.....)))))))))))))).)). ( -40.70) >DroEre_CAF1 14441 92 - 1 --------------------------GCUGCAUUUCGACCAAUGUACUGUAACGAUAUGCAACAUUGUGUGCUGGCGCUCUCUCACACUGUUAACGCUGGGUUAACAGUGCGUGAGCG --------------------------(((((((......((((((..((((......)))))))))).)))).)))((((...(.((((((((((.....)))))))))).).)))). ( -33.10) >DroYak_CAF1 14204 118 - 1 UGCAUCGUAAAGUCAUUGCAUGUACUGCUGCAUUUCGCCCAAUGUACUGUAAAGAAAUGCAACAUUGUAAGCUGACGCUCUCUCGCACUGUUAAUGCUCGGUUAACAGUGCGUGAGCA ...........((((((((((((.....((((((((...((......))....))))))))))).)))))..))))((((...((((((((((((.....)))))))))))).)))). ( -40.70) >consensus GAAAUCGUGAAUUCAUUGCAUAUGUUGCUGCAUUUCGGCCAAUGUACUGUAAACAAAUGCAACAUUGAGAGCUGGCGCUCUCUUGCACUGUUAACGUUCGGUUAACAGUGCGUGAGCA ................((((........))))..(((((((((((..((((......))))))))))...))))).((((...((((((((((((.....)))))))))))).)))). (-31.50 = -32.54 + 1.04)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:47:01 2006