
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 1,514,832 – 1,514,980 |
| Length | 148 |
| Max. P | 0.988601 |

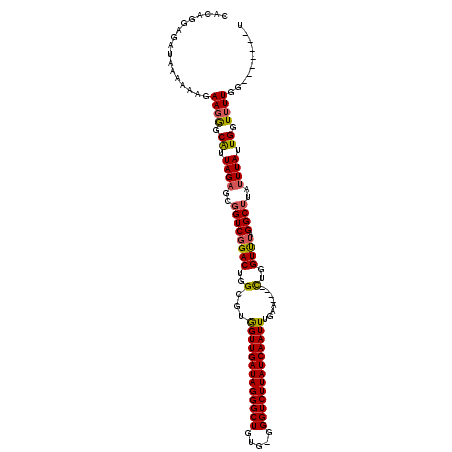
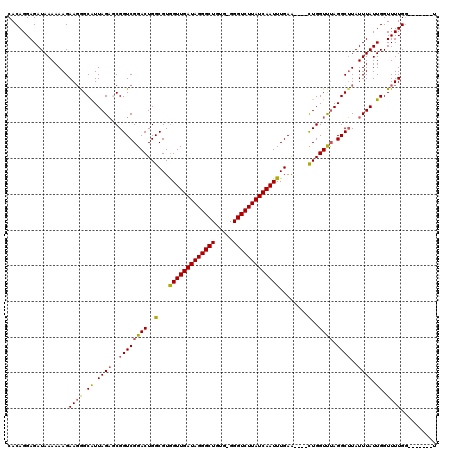
| Location | 1,514,832 – 1,514,940 |
|---|---|
| Length | 108 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.60 |
| Mean single sequence MFE | -29.30 |
| Consensus MFE | -24.12 |
| Energy contribution | -23.96 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.55 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.56 |
| SVM RNA-class probability | 0.782243 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1514832 108 - 22224390 CACAGGAGAUAAAAAAGAAGGGCAUUAGAGCGGUCGGACUGGCGUGGUUGAUAGGGCUGUG-GGGUCUUAUCAAUUUGAA----CUGGUUAAGGCUUAUUUAUUGGUUUUGG-------U .................((((.((.((((..((((.(((..(...(((((((((((((...-.)))))))))))))....----)..)))..))))..)))).)).))))..-------. ( -27.00) >DroSec_CAF1 4292 108 - 1 CACAGGAGAUAAAAAAGAAGAGCAUUAGAGCGGUCGGACUGGCGUGGUUGAUAGGGCUGUG-GGGUCUUAUCAAUUUGAA----CUGGUUUAGGCUUAUUUAUUGGUUUUGG-------U .................((((.((.((((..((((((((..(...(((((((((((((...-.)))))))))))))....----)..)))).))))..)))).)).))))..-------. ( -28.40) >DroSim_CAF1 5514 108 - 1 CACAGGAGAUAAAAAAGAAGAGCAUUAGAGCGGUCGGACUGGCGUGGUUGAUAGGGCUGUG-GGGUCUUAUCAAUUUGAA----CUGGUCUAGGCUUAUUUAUUGGUUUUGG-------U .................((((.((.((((..((((((((..(...(((((((((((((...-.)))))))))))))....----)..)))).))))..)))).)).))))..-------. ( -31.10) >DroEre_CAF1 4326 115 - 1 CACAGGAGAUAAAAAAGAAUGGCGUUAGAUCGGUCGGACUGGCGUAGUUGAUAGGGCUGUG-GGGUCUUAUCAAUUUGAA----CUGGUUUAGGCUUAUUUAUUGGUUUUGGUAUUUGGU ......(((((....(((((.(...(((((.((((((((..(...(((((((((((((...-.)))))))))))))....----)..)))).)))).))))).).)))))..)))))... ( -29.00) >DroYak_CAF1 5869 113 - 1 CACAGGAGAUAAAAAAGAAGGGCGUUAGUUCGGUCGGACUGGCGUGGUUGAUAGGGCUGCGUGGGUCUUAUCAAUUUGAGAACUUUGGUUUAGGCAUAUUUAUUGGUUUUGA-------U ..(((((((((((...((((.((((((((((....))))))))))(((((((((((((.....)))))))))))))......)))).((....))...))))))..))))).-------. ( -31.00) >consensus CACAGGAGAUAAAAAAGAAGGGCAUUAGAGCGGUCGGACUGGCGUGGUUGAUAGGGCUGUG_GGGUCUUAUCAAUUUGAA____CUGGUUUAGGCUUAUUUAUUGGUUUUGG_______U .................((((.((.((((..((((((((..(...(((((((((((((.....)))))))))))))........)..)))).))))..)))).)).)))).......... (-24.12 = -23.96 + -0.16)

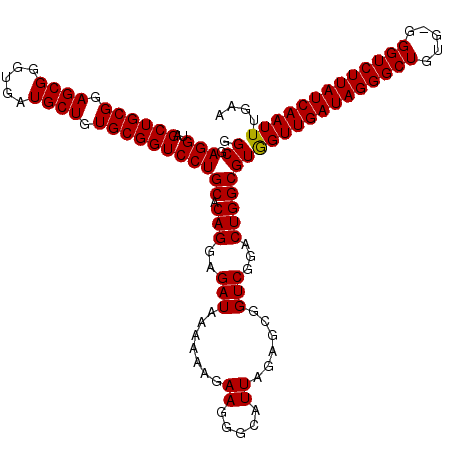
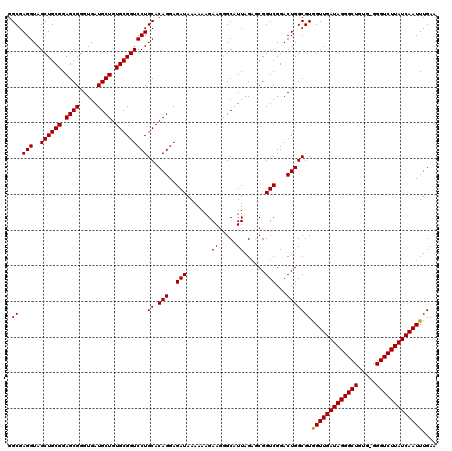
| Location | 1,514,861 – 1,514,980 |
|---|---|
| Length | 119 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.48 |
| Mean single sequence MFE | -39.28 |
| Consensus MFE | -35.92 |
| Energy contribution | -35.76 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.81 |
| Structure conservation index | 0.91 |
| SVM decision value | 2.13 |
| SVM RNA-class probability | 0.988601 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1514861 119 - 22224390 GGCGAGGUAGCUGCGGAGCGGGUGAUGCUGUGCGGUCCUGCACAGGAGAUAAAAAAGAAGGGCAUUAGAGCGGUCGGACUGGCGUGGUUGAUAGGGCUGUG-GGGUCUUAUCAAUUUGAA .((.(((..((((((.((((.....)))).)))))))))((.(((..(((...........((......)).)))...)))))))(((((((((((((...-.))))))))))))).... ( -36.30) >DroSec_CAF1 4321 119 - 1 GGCGAGGUAGCUGCGGAGCGGGUGAUGCUGUGCGGUCCUGCACAGGAGAUAAAAAAGAAGAGCAUUAGAGCGGUCGGACUGGCGUGGUUGAUAGGGCUGUG-GGGUCUUAUCAAUUUGAA .((.(((..((((((.((((.....)))).)))))))))((.(((..(((...........((......)).)))...)))))))(((((((((((((...-.))))))))))))).... ( -37.40) >DroSim_CAF1 5543 119 - 1 GGCGAGGUAGCUGCGGAGCGGGUGAUGCUGUGCGGUCCUGCACAGGAGAUAAAAAAGAAGAGCAUUAGAGCGGUCGGACUGGCGUGGUUGAUAGGGCUGUG-GGGUCUUAUCAAUUUGAA .((.(((..((((((.((((.....)))).)))))))))((.(((..(((...........((......)).)))...)))))))(((((((((((((...-.))))))))))))).... ( -37.40) >DroEre_CAF1 4362 119 - 1 GGCGAGGUAGCUGCGGAGCGGGUGAUGCUGUGCGGUCCUGCACAGGAGAUAAAAAAGAAUGGCGUUAGAUCGGUCGGACUGGCGUAGUUGAUAGGGCUGUG-GGGUCUUAUCAAUUUGAA .((.(((..((((((.((((.....)))).)))))))))))....................(((((((.((.....)))))))))(((((((((((((...-.))))))))))))).... ( -40.40) >DroYak_CAF1 5902 120 - 1 GGCGAGGUAGCUGCGGAGCGGGUGAUGCUGUGCGGUCCUGCACAGGAGAUAAAAAAGAAGGGCGUUAGUUCGGUCGGACUGGCGUGGUUGAUAGGGCUGCGUGGGUCUUAUCAAUUUGAG .((.(((..((((((.((((.....)))).)))))))))))....................((((((((((....))))))))))(((((((((((((.....))))))))))))).... ( -44.90) >consensus GGCGAGGUAGCUGCGGAGCGGGUGAUGCUGUGCGGUCCUGCACAGGAGAUAAAAAAGAAGGGCAUUAGAGCGGUCGGACUGGCGUGGUUGAUAGGGCUGUG_GGGUCUUAUCAAUUUGAA .((.(((..((((((.((((.....)))).)))))))))((.(((..(((.......((.....))......)))...)))))))(((((((((((((.....))))))))))))).... (-35.92 = -35.76 + -0.16)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:46:55 2006