
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 13,649,744 – 13,649,873 |
| Length | 129 |
| Max. P | 0.999960 |

| Location | 13,649,744 – 13,649,852 |
|---|---|
| Length | 108 |
| Sequences | 5 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 90.75 |
| Mean single sequence MFE | -33.06 |
| Consensus MFE | -28.38 |
| Energy contribution | -27.58 |
| Covariance contribution | -0.80 |
| Combinations/Pair | 1.13 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.86 |
| SVM decision value | 1.49 |
| SVM RNA-class probability | 0.958366 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 13649744 108 - 22224390 GAUGACAUCAUCGUCAUCAUUUGUGUGAUGUGUUGUUAGGCUUUAGCUGCUUCUCAGCUGCAGCAAUGCCAUCGAUCUACAGUGGAUAUUGGCUAAGUAGAA-CCGCCA ((..(((.(((((.(((.....)))))))))))..)).(((....(((((.........)))))...((((...((((.....))))..)))).........-..))). ( -32.90) >DroSec_CAF1 2062 99 - 1 GAUGACAUCAUCGUCAUCAUUUGUGUGAUGUGU---UUGGCUUUAGCUGCUUCUCAGCUGCAGCAAUGCCAUCGAUCUACAGUGGAUACCGGCUAAGCGGAA------- ((((((......))))))..((((((((((.((---...(((.((((((.....)))))).)))...)))))))...)))))......(((......)))..------- ( -30.80) >DroSim_CAF1 2043 99 - 1 GAUGACAUCAUCGUCAUCAUUUGUGUGAUGUGU---UUGGCUUUAGCUGCUUCUCAGCUGCAGCAAUGCCAUCGAUCUACAGUGGAUACCGGCUAAGCGGAA------- ((((((......))))))..((((((((((.((---...(((.((((((.....)))))).)))...)))))))...)))))......(((......)))..------- ( -30.80) >DroEre_CAF1 3050 106 - 1 GAUGACAUCAUCGUCAUCAUUUGUGUGAUGUGU---UUGGCUUUAGCUGCUUCUCAGCUGCAGCAAUGCCAUCGAUCUACAGUGGACACCGGCUAAGCGGAUUUUGCGU ((((((......))))))....(((.(((.(((---((((((...(((((.........)))))..((((((.(.....).)))).))..))))))))).))).))).. ( -34.00) >DroYak_CAF1 2151 105 - 1 GAUGACAUCAUCGUCGUCAUUUGUGUGAUGUGU---UUGGCUUUAGCUGCUUCUCAGCUGCAGCAAUGCCAUCGAUCUACAGUGGAUACCGGCUACGCGGAU-CUGCGA ((((((......))))))...(((((((((.((---...(((.((((((.....)))))).)))...)))))))...))))(..(((.(((......)))))-)..).. ( -36.80) >consensus GAUGACAUCAUCGUCAUCAUUUGUGUGAUGUGU___UUGGCUUUAGCUGCUUCUCAGCUGCAGCAAUGCCAUCGAUCUACAGUGGAUACCGGCUAAGCGGAA___GC__ ((((((......))))))..((((((((((.((......(((.((((((.....)))))).)))...)))))))...)))))......(((......)))......... (-28.38 = -27.58 + -0.80)



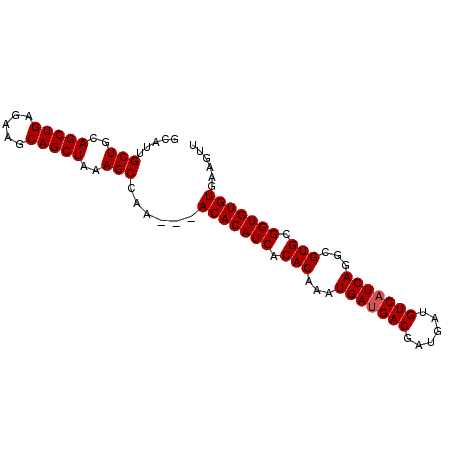
| Location | 13,649,783 – 13,649,873 |
|---|---|
| Length | 90 |
| Sequences | 5 |
| Columns | 90 |
| Reading direction | forward |
| Mean pairwise identity | 97.73 |
| Mean single sequence MFE | -32.24 |
| Consensus MFE | -32.02 |
| Energy contribution | -32.22 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.76 |
| Structure conservation index | 0.99 |
| SVM decision value | 4.90 |
| SVM RNA-class probability | 0.999960 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 13649783 90 + 22224390 GCAUUGCUGCAGCUGAGAAGCAGCUAAAGCCUAACAACACAUCACACAAAUGAUGACGAUGAUGUCAUCAGGCGUGCGGUGUGUGAAGUU .....(((..(((((.....)))))..)))......(((((((.(((...(((((((......)))))))...))).)))))))...... ( -33.10) >DroSec_CAF1 2095 87 + 1 GCAUUGCUGCAGCUGAGAAGCAGCUAAAGCCAA---ACACAUCACACAAAUGAUGACGAUGAUGUCAUCAGGCGUGCGGUGUGUGAAGUU .....(((..(((((.....)))))..)))...---(((((((.(((...(((((((......)))))))...))).)))))))...... ( -33.10) >DroSim_CAF1 2076 87 + 1 GCAUUGCUGCAGCUGAGAAGCAGCUAAAGCCAA---ACACAUCACACAAAUGAUGACGAUGAUGUCAUCAGGCGUGCGGUGUGUGAAGUU .....(((..(((((.....)))))..)))...---(((((((.(((...(((((((......)))))))...))).)))))))...... ( -33.10) >DroEre_CAF1 3090 87 + 1 GCAUUGCUGCAGCUGAGAAGCAGCUAAAGCCAA---ACACAUCACACAAAUGAUGACGAUGAUGUCAUCAGGCGUGCGGUGUGUGAAGUU .....(((..(((((.....)))))..)))...---(((((((.(((...(((((((......)))))))...))).)))))))...... ( -33.10) >DroYak_CAF1 2190 87 + 1 GCAUUGCUGCAGCUGAGAAGCAGCUAAAGCCAA---ACACAUCACACAAAUGACGACGAUGAUGUCAUCAGGCGUGCGGUGUGUGAAGUU .....(((..(((((.....)))))..)))...---(((((((.(((...(((.(((......))).)))...))).)))))))...... ( -28.80) >consensus GCAUUGCUGCAGCUGAGAAGCAGCUAAAGCCAA___ACACAUCACACAAAUGAUGACGAUGAUGUCAUCAGGCGUGCGGUGUGUGAAGUU .....(((..(((((.....)))))..)))......(((((((.(((...(((((((......)))))))...))).)))))))...... (-32.02 = -32.22 + 0.20)

| Location | 13,649,783 – 13,649,873 |
|---|---|
| Length | 90 |
| Sequences | 5 |
| Columns | 90 |
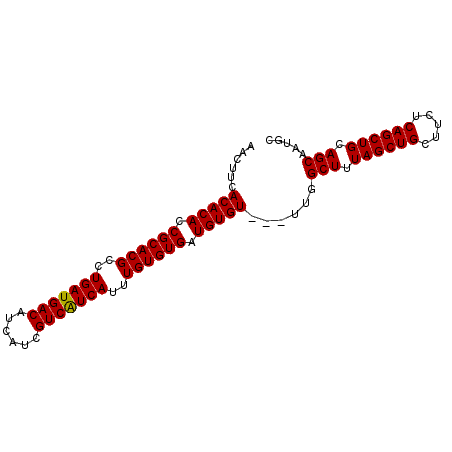
| Reading direction | reverse |
| Mean pairwise identity | 97.73 |
| Mean single sequence MFE | -34.36 |
| Consensus MFE | -34.30 |
| Energy contribution | -34.14 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.04 |
| Mean z-score | -5.25 |
| Structure conservation index | 1.00 |
| SVM decision value | 3.38 |
| SVM RNA-class probability | 0.999109 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 13649783 90 - 22224390 AACUUCACACACCGCACGCCUGAUGACAUCAUCGUCAUCAUUUGUGUGAUGUGUUGUUAGGCUUUAGCUGCUUCUCAGCUGCAGCAAUGC (((...(((((.((((((..(((((((......)))))))..)))))).))))).)))..(((.((((((.....)))))).)))..... ( -35.00) >DroSec_CAF1 2095 87 - 1 AACUUCACACACCGCACGCCUGAUGACAUCAUCGUCAUCAUUUGUGUGAUGUGU---UUGGCUUUAGCUGCUUCUCAGCUGCAGCAAUGC ......(((((.((((((..(((((((......)))))))..)))))).)))))---...(((.((((((.....)))))).)))..... ( -34.30) >DroSim_CAF1 2076 87 - 1 AACUUCACACACCGCACGCCUGAUGACAUCAUCGUCAUCAUUUGUGUGAUGUGU---UUGGCUUUAGCUGCUUCUCAGCUGCAGCAAUGC ......(((((.((((((..(((((((......)))))))..)))))).)))))---...(((.((((((.....)))))).)))..... ( -34.30) >DroEre_CAF1 3090 87 - 1 AACUUCACACACCGCACGCCUGAUGACAUCAUCGUCAUCAUUUGUGUGAUGUGU---UUGGCUUUAGCUGCUUCUCAGCUGCAGCAAUGC ......(((((.((((((..(((((((......)))))))..)))))).)))))---...(((.((((((.....)))))).)))..... ( -34.30) >DroYak_CAF1 2190 87 - 1 AACUUCACACACCGCACGCCUGAUGACAUCAUCGUCGUCAUUUGUGUGAUGUGU---UUGGCUUUAGCUGCUUCUCAGCUGCAGCAAUGC ......(((((.((((((..(((((((......)))))))..)))))).)))))---...(((.((((((.....)))))).)))..... ( -33.90) >consensus AACUUCACACACCGCACGCCUGAUGACAUCAUCGUCAUCAUUUGUGUGAUGUGU___UUGGCUUUAGCUGCUUCUCAGCUGCAGCAAUGC ......(((((.((((((..(((((((......)))))))..)))))).)))))......(((.((((((.....)))))).)))..... (-34.30 = -34.14 + -0.16)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:42:49 2006