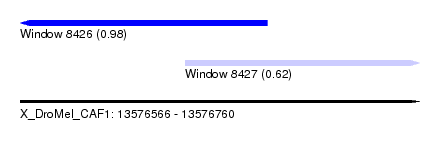
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 13,576,566 – 13,576,760 |
| Length | 194 |
| Max. P | 0.975389 |

| Location | 13,576,566 – 13,576,686 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.39 |
| Mean single sequence MFE | -40.24 |
| Consensus MFE | -29.26 |
| Energy contribution | -29.47 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.57 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.73 |
| SVM decision value | 1.75 |
| SVM RNA-class probability | 0.975389 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
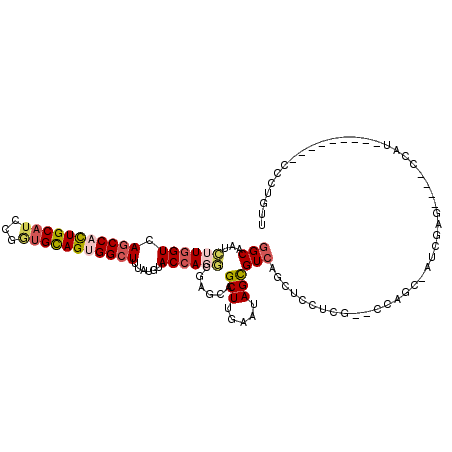
>X_DroMel_CAF1 13576566 120 - 22224390 AAAGCCACUGCACCGGAUGCAGUGGUUAACCAAGAUUGCCGACGCCUGCCAGGUAAAGAAGGGCGGUGAUUUGGCAUCGACCGUUUAUGAUUUCCUUGACAACGGUAACGAUAUGGUCAA ..((((((((((.....)))))))))).((((.....(((((((((.(((...........)))))))..)))))((((((((((...............))))))..)))).))))... ( -41.26) >DroVir_CAF1 984 120 - 1 GGAGCCACUGCGGCGCAUGCAAUGGCUGACGAAAAUCGCCGAUGCCUGCCAGCUAAAGAAGGGCGGUGACCUGGCCUCCGUGCUAUACACAUUUCUAAACAAUGGUGAUCCCAUGAUCAA (((((((.((((.....)))).))))).......((((((((.(((.(((..(....)...)))(....)..))).)).(((.....))).............)))))).))........ ( -34.30) >DroEre_CAF1 968 120 - 1 AAAGCCACUGCACCGGAUGCAGUGGCUAACCAAGAUCGCCGACGCCUGCCAGAUGAAGAAGGGCGGUGAAUUGGCAUCGACCGUGUACGAUUUCCUCGACAAUGGUAACUCGAUGGUGAA ..((((((((((.....)))))))))).((((.(((.(((((((((.(((...........)))))))..)))))))).(((((((.(((.....))))).))))).......))))... ( -46.50) >DroWil_CAF1 978 120 - 1 GGAGCCAUUGCAUCGCAUGCAGUGGUUGACCAAAAUUGCCGAUGCUUGCCAAAUGAAAAAGGGUGGCGAAUUGGCAUCAGUUGUAUACGAAUAUCUCGAUAAUGGCAAUCCAAUGGUUAA ..(((((((((((...)))))))))))(((((..(((((((((((((((((..(......)..)))))....)))))).........(((.....))).....))))))....))))).. ( -41.50) >DroYak_CAF1 980 120 - 1 AAAGCCACUGCACCGGAUGCAGUGGCUAACCAAGAUCGCCGACGCCUGUCAGAUGAAGAAGGGCGGUGAACUGGCAUCGACUGUGUACGAUUUCCUCGACAACGGUCAUUCGAUGGUUAA ...(((((((((.....)))))))))((((((.(((.((((.(((((.((.......)).)))))(....))))))))((((((((.(((.....))))).))))))......)))))). ( -49.60) >DroMoj_CAF1 961 120 - 1 GGAGCCACUUCGUCGAAUGCAAUGGCUUACAAAAAUCGCCGAUGCCUGCCAAAUGAAGAAGGGCGGCGACUUAGCAUCCGUGAUCUAUAAAUUUUUAAAUAAUGGGGACCCCAUGGUAAA ...(((...(((.((.((((..((((...........)))).((((.(((...........))))))).....)))).)))))..................(((((...))))))))... ( -28.30) >consensus AAAGCCACUGCACCGGAUGCAGUGGCUAACCAAAAUCGCCGACGCCUGCCAGAUGAAGAAGGGCGGUGAAUUGGCAUCGACCGUAUACGAAUUCCUCGACAAUGGUAACCCCAUGGUCAA ..((((((((((.....))))))))))(((((.....(((((((((.(((...........)))))))..)))))....(((((...(((.....)))...))))).......))))).. (-29.26 = -29.47 + 0.20)


| Location | 13,576,646 – 13,576,760 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 72.02 |
| Mean single sequence MFE | -35.78 |
| Consensus MFE | -20.36 |
| Energy contribution | -20.92 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.22 |
| Mean z-score | -1.40 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.622020 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 13576646 114 + 22224390 GGCAAUCUUGGUUAACCACUGCAUCCGGUGCAGUGGCUUUAUAUACCAGGCGAGCAGCUUGAAUAGCGUCAUUUCAUCG--CCAGC-AUCGGGCCCACUAUCCUUUUUCCCCUUGUU (((...((((((.(((((((((((...))))))))).)).....)))))).((...(((.....))).))........)--))(((-(..(((...............)))..)))) ( -31.26) >DroVir_CAF1 1064 104 + 1 GGCGAUUUUCGUCAGCCAUUGCAUGCGCCGCAGUGGCUCCAUGUACCAGCAGAGGAGCUUCAUUAGCGUCAGCUGCUCGCGUUGCCUGUCCAA----CCAG---------CUCUGAU ((((.....)))).((((((((.......))))))))(((.(((....)))..)))(((.....)))(((((..(((.(.((((......)))----))))---------).))))) ( -34.40) >DroPse_CAF1 1032 104 + 1 GGCAAUUUUGGUCAGCCAUUGCAUACGAUGUAGGGGCUUUGUGUACCACACGAGCAGCUUGAAUAGCGUCACCUGCUCCCGCUGGCUAUCGAG----CCAC---------UCCUGCU (((.....(((((((((((((....)))))..((((..(((((.....)))))((((..(((......))).))))))))))))))))....)----))..---------....... ( -33.70) >DroEre_CAF1 1048 105 + 1 GGCGAUCUUGGUUAGCCACUGCAUCCGGUGCAGUGGCUUUAUAUACCAGGCGAGUAGCUUAAAAAGUGUGAGUUCGUCG--GCAGC-AUCUAGCUCACUAU---------CCCUGUU (((((.((((((.(((((((((((...)))))))))))......)))((((.....)))).........))).)))))(--((((.-...(((....))).---------..))))) ( -34.90) >DroYak_CAF1 1060 105 + 1 GGCGAUCUUGGUUAGCCACUGCAUCCGGUGCAGUGGCUUUAUGUACCAGGCGAGCAGCUUAAAGAGCGUCAGUUCGUCG--GCAGC-AUCAGGUUCGCCAU---------CCUUGUU (((((.((((((.(((((((((((...)))))))))))...(((....(((((((.((((...))))....))))))).--)))..-)))))).)))))..---------....... ( -46.70) >DroPer_CAF1 1032 104 + 1 GGCAAUUUUGGUCAGCCAUUGCAUACGAUGUAGGGGCUUUGUGUACCACACGAGCAGCUUGAAUAGCGUCACCUGCUCCCGCUGGCUAUCGAG----CCAC---------UCCUGCU (((.....(((((((((((((....)))))..((((..(((((.....)))))((((..(((......))).))))))))))))))))....)----))..---------....... ( -33.70) >consensus GGCAAUCUUGGUCAGCCACUGCAUCCGGUGCAGUGGCUUUAUGUACCAGGCGAGCAGCUUGAAUAGCGUCAGCUCCUCG__CCAGC_AUCGAG____CCAU_________CCCUGUU (((...((((((.(((((((((((...)))))))))))......))))))......(((.....))))))............................................... (-20.36 = -20.92 + 0.56)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:42:05 2006