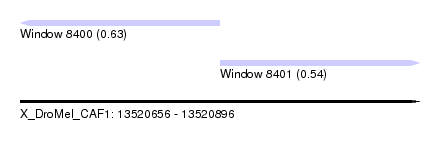
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 13,520,656 – 13,520,896 |
| Length | 240 |
| Max. P | 0.632278 |

| Location | 13,520,656 – 13,520,776 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.33 |
| Mean single sequence MFE | -45.07 |
| Consensus MFE | -28.35 |
| Energy contribution | -27.97 |
| Covariance contribution | -0.38 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.06 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.632278 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
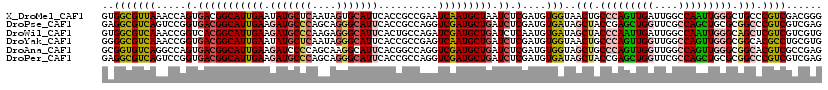
>X_DroMel_CAF1 13520656 120 - 22224390 GUUUCCGUACCCAGGGUGGAUGUGCUCGACAGAAUCUGGCAUUGCAGAAUAUGCAAUCGAGGAUACGCAUGGUCCUGGCCUACAUCUUUGCCCAGUUGACGUUGUGGGUAAGAAACAGGC (((((..(((((((((((((((((((((((((...)))...(((((.....)))))))))).........(((....))))))))))..)))).(....)....)))))).))))).... ( -39.90) >DroPse_CAF1 5345 120 - 1 GCUUCCGAACCCAGGGCGGCUGUGCCCGCCAGAAUUUGGCCCUGCAGAACAUCCAAUCACGCCUGCGCAUGGUCCUGGCGUACAUUUUCGCCCAGCUUAUGCUGUGGGUGAGGAAUCGAC (.((((..(((((((((((..(((..((((((.....((((.(((((...............)))))...))))))))))..)))..))))))(((....))).)))))..)))).)... ( -50.86) >DroEre_CAF1 3754 120 - 1 GAUUCUGGACGCAGGGUGGCUGUGCCCGUCAGAAUCUGGCAUUGCAGAAUAUGCAAUCGAGGAUACGCAUGGUCCUGGCCUACAUCUUUGCCCAGUUGAUGUUGUGGGUAAGGAACAGGC ((((((((.....((((......)))).)))))))).(((((((((.....))))))..(((((.......))))).)))....((((((((((..........))))))))))...... ( -42.60) >DroWil_CAF1 3710 120 - 1 GAUUUCGCACCCAGGGCGGAUGUGCUCGACAAAAUUUGGCUUUGCAGAAUAUUCAAUCGAGAAUUCGCAUGGUUCUGGCCUAUAUUUUUGCUCAGCUCAUGCUUUGGGUGAGAAAUCGUC ((((((.((((((((((..(((.(((.(((((((...((((.(((.((((.(((....))).))))))).......)))).....))))).))))).))))))))))))).))))))... ( -45.30) >DroAna_CAF1 7900 120 - 1 GUUUCCGCACCCAAGGUGGCUGCGCCCGCCAGAAUCUGGCUCUGCAGAACAUCCAAUCUCGCAUCCGCAUGGUCCUGGCCUACAUCUUUGCCCAGCUUAUGCUCUGGGUGAGGAAUAGAC ..((((.((((((.((((.(((((...(((((...)))))..)))))..)))).............((((((..((((.(.........).)))).))))))..)))))).))))..... ( -40.90) >DroPer_CAF1 6558 120 - 1 GCUUCCGAACCCAGGGCGGCUGUGCCCGCCAGAAUUUGGCCCUGCAGAACAUCCAAUCACGCCUGCGCAUGGUCCUGGCGUACAUUUUCGCCCAGCUUAUGCUGUGGGUGAGGAAUCGAC (.((((..(((((((((((..(((..((((((.....((((.(((((...............)))))...))))))))))..)))..))))))(((....))).)))))..)))).)... ( -50.86) >consensus GAUUCCGAACCCAGGGCGGCUGUGCCCGCCAGAAUCUGGCACUGCAGAACAUCCAAUCGAGCAUACGCAUGGUCCUGGCCUACAUCUUUGCCCAGCUUAUGCUGUGGGUGAGGAAUAGAC ..((((..(((((((((((..(((...(((((.....((((.(((.((........))..(....)))).)))))))))...)))..))))))(((....))).)))))..))))..... (-28.35 = -27.97 + -0.38)



| Location | 13,520,776 – 13,520,896 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 83.44 |
| Mean single sequence MFE | -51.07 |
| Consensus MFE | -33.41 |
| Energy contribution | -34.17 |
| Covariance contribution | 0.76 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.05 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.541680 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
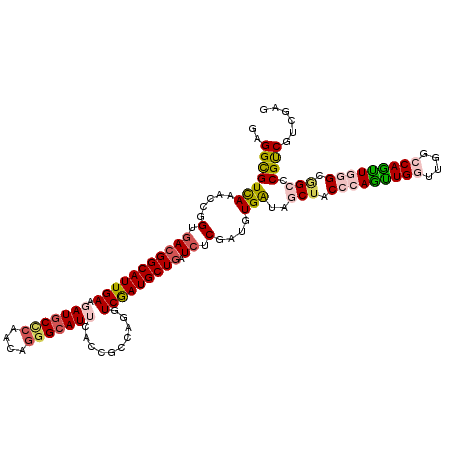
>X_DroMel_CAF1 13520776 120 + 22224390 GUGGCGUUAAACCAGUGACGGCAUUGAAUAUGCUCAAUAGUGCAUUCACCGCCGAAUCAAUGCUAAUCUCGAUGUGGUAACUGCCCAGUUGAUUGGCCAAUUGGGCUGCCCGUCGACGGG .((((((........(((((((..(((((..(((....)))..)))))..))))..))))))))).((((((((.((((...(((((((((......))))))))))))))))))).)). ( -42.80) >DroPse_CAF1 5465 120 + 1 GAGGCGUCAGUCCGGUGACGGCAUUGAAGAUGCCCAGCAGGGCAUUCACCGCCAGGUCGAUGCUGAUCUCGAUGUGAUAGCUACCGAGCUGGUUCGCCAGCUGCGCGGCCCGUCGUCGAG ..((((((((((((....))).))))).(((((((....)))))))...)))).(((((....)))))((((((.....(((..((((((((....)))))).)).)))....)))))). ( -53.00) >DroWil_CAF1 3830 120 + 1 GUGGCGUCAAACCGGUCACGGCAUUGAAGAUGCCCAAGAGGGCAUUCACUGCCAGAUCGAUGCUGAUCUCAAUGUGAUAGCUACCCAAUUGAUUGGCCAAUUGGGCAGCUCGUCGUCGUG ..((((.(......(((((((((.(((..((((((....))))))))).))))((((((....))))))....)))))((((.((((((((......)))))))).)))).).))))... ( -51.20) >DroYak_CAF1 3788 120 + 1 GGGGCGUCAAACCGGUGACGGCAUUGAAUAUGCUCAAUAGGGCAUUCACCGCCGAGUCAAUGCUGAUCUCGAUGUGGUAACUGCCCAGUUGGUUGGCCAGUUGGGCGGCACGCCUGCGUG .(((((((((.(((....)))..))))..((((((....))))))..((((((((((((....))).))))..)))))..(((((((..(((....)))..)))))))..)))))..... ( -52.30) >DroAna_CAF1 8020 120 + 1 GCGGUGUCAGGCCAGUGACGGCAUUGAAGAUCCCCAGCAAGGCAUUCACGGCCAGGUCGAUGCUGAUCUCGAUGUGGUAGCUGCCCAGUUGGUUGGCCAGUUGGGCGGCACGUCGCCGAG .(((((.(..((((((.((.(((((((.((((........((((((.((......)).))))))))))))))))).)).)))(((((..(((....)))..))))))))..).))))).. ( -54.10) >DroPer_CAF1 6678 120 + 1 GAGGCGUCAGUCCGGUGACGGCAUUGAAGAUGCCCAGCAGGGCAUUCACCGCCAGGUCGAUGCUGAUCUCGAUGUGAUAGCUACCGAGCUGGUUCGCCAGCUGCGCGGCCCGUCGUCGAG ..((((((((((((....))).))))).(((((((....)))))))...)))).(((((....)))))((((((.....(((..((((((((....)))))).)).)))....)))))). ( -53.00) >consensus GAGGCGUCAAACCGGUGACGGCAUUGAAGAUGCCCAACAGGGCAUUCACCGCCAGGUCGAUGCUGAUCUCGAUGUGAUAGCUACCCAGUUGGUUGGCCAGUUGGGCGGCCCGUCGUCGAG ..(((((((.....(.(((((((((((.(((((((....)))))))..........))))))))).)).)....)))..(((.(((((((((....))))))))).))).))))...... (-33.41 = -34.17 + 0.76)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:41:42 2006