
| Sequence ID | X_DroMel_CAF1 |
|---|---|
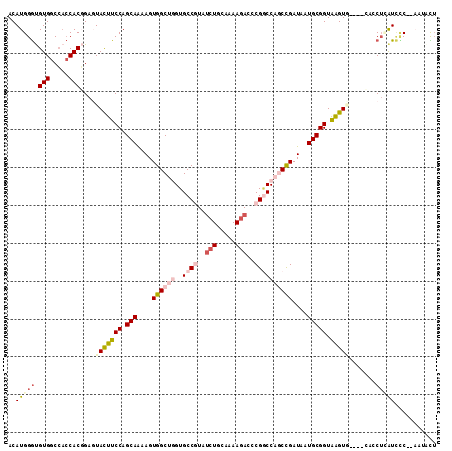
| Location | 13,242,428 – 13,242,541 |
| Length | 113 |
| Max. P | 0.907807 |

| Location | 13,242,428 – 13,242,541 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 79.72 |
| Mean single sequence MFE | -40.88 |
| Consensus MFE | -20.86 |
| Energy contribution | -22.83 |
| Covariance contribution | 1.97 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.71 |
| Structure conservation index | 0.51 |
| SVM decision value | 1.06 |
| SVM RNA-class probability | 0.907807 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
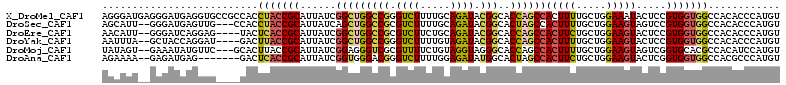
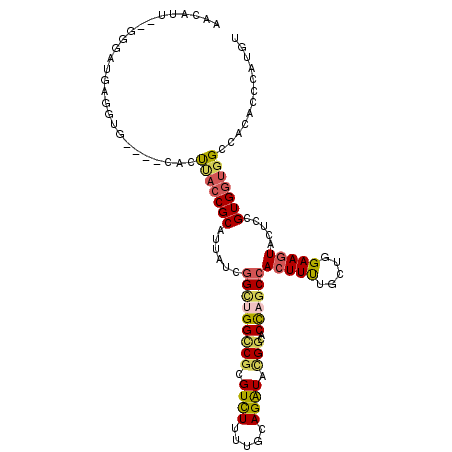
>X_DroMel_CAF1 13242428 113 + 22224390 ACAUGGGUGUGGCCACCACGGAGUAUUUCCAGCAAAAGUGGCUGGUGCCGUAUCUGCAAAAGACCCGGCCAGCCGAUAAUGCGGUAGGUGGCGGCACCUCAUCCCUCAUCCCU ..((((((((.((((((((((((...)))).(((....((((((..((((..(((.....)))..))))))))))....))).)).)))))).))))).)))........... ( -50.80) >DroSec_CAF1 3389 108 + 1 ACAUGGGUGUGGCCACCACGGACUACUUCCAGCAAAAGUGGCUAGUGCCGUAUCUGCAAAAGACGCGGCCAGCUGAUAAUGCGGUAGGUGG---CAACUCAUCCC--AAUGCU ...((((((..(((((((((((.....))).(((....(((((.(.((((..(((.....)))..))))))))))....))).)).)))))---)....)).)))--)..... ( -43.40) >DroEre_CAF1 6171 107 + 1 ACAUGGGUGUGGCCACCACGGAGUACUUCCAGCAGAAGUGGCUGGUGCCGUAUCUGCAGAAGACGCGGCCAGCCGAUAAUGCGGUGAGUA----CUCCUGAUCCC--AAUGUU (((((((.((((...))))(((((((((((.(((....((((((..((((..(((.....)))..))))))))))....))))).)))))----))))....)))--.)))). ( -51.80) >DroYak_CAF1 3456 107 + 1 ACAUGGGUGUGGCCACCACGGAGUACUUCCAGCAAAAGUGGCUGGUGCCGUAUCUACAAAAGACCCGGCCAGCCGAUAAUGCGGUAAGUC----AUCCUGGUAGC--UAAAUU .........((((.((((.(((..((((((.(((....((((((..((((..(((.....)))..))))))))))....))))).)))).----.))))))).))--)).... ( -41.30) >DroMoj_CAF1 3539 108 + 1 ACAUGGAUGUGGCGUGCACCGACUACUUCCAGCAAAAGUGGCUGGUGCCCUACCUACAGAAAACGCGACCCUCCGAUAAUGCGGUAAGUGC---GAACAUAUUUC--ACUAUA ..((((.(((((.((((((((.((((((.......)))))).))))))...))))))).....((((.....(((......)))....)))---)..........--.)))). ( -30.20) >DroAna_CAF1 5448 104 + 1 ACAUGGGCGUGGCCACCACCGAGUACUUCCAGCAGAAGUGGCUAGUGCCAUAUCUCCAAAAGACCCGUCCCACCGAUAAUGCGGUGAGUC-------CUCAUCUC--UUUUCU ....((((((((...))))((.((.(((...(.(((.(((((....))))).))).)..))))).))...(((((......))))).)))-------).......--...... ( -27.80) >consensus ACAUGGGUGUGGCCACCACGGAGUACUUCCAGCAAAAGUGGCUGGUGCCGUAUCUGCAAAAGACCCGGCCAGCCGAUAAUGCGGUAAGUG____CACCUCAUCCC__AAUACU ..(((((.(((.....)))....(((((((.(((....((((((..((((..(((.....)))..))))))))))....))))).))))).......)))))........... (-20.86 = -22.83 + 1.97)


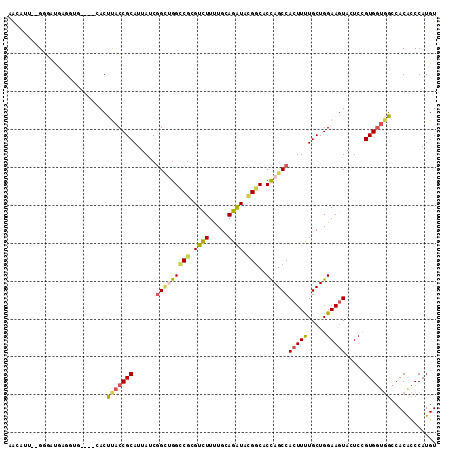
| Location | 13,242,428 – 13,242,541 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 79.72 |
| Mean single sequence MFE | -40.75 |
| Consensus MFE | -22.62 |
| Energy contribution | -22.85 |
| Covariance contribution | 0.23 |
| Combinations/Pair | 1.36 |
| Mean z-score | -2.21 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.64 |
| SVM RNA-class probability | 0.809232 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 13242428 113 - 22224390 AGGGAUGAGGGAUGAGGUGCCGCCACCUACCGCAUUAUCGGCUGGCCGGGUCUUUUGCAGAUACGGCACCAGCCACUUUUGCUGGAAAUACUCCGUGGUGGCCACACCCAUGU ........(((.((..(.((((((((.....(((.....(((((((((..(((.....)))..))))..))))).....))).(((.....)))))))))))).))))).... ( -45.60) >DroSec_CAF1 3389 108 - 1 AGCAUU--GGGAUGAGUUG---CCACCUACCGCAUUAUCAGCUGGCCGCGUCUUUUGCAGAUACGGCACUAGCCACUUUUGCUGGAAGUAGUCCGUGGUGGCCACACCCAUGU .((((.--((..((.((..---((((..((..(...........((((..(((.....)))..)))).(((((.......)))))..)..))..))))..))))..)).)))) ( -38.30) >DroEre_CAF1 6171 107 - 1 AACAUU--GGGAUCAGGAG----UACUCACCGCAUUAUCGGCUGGCCGCGUCUUCUGCAGAUACGGCACCAGCCACUUCUGCUGGAAGUACUCCGUGGUGGCCACACCCAUGU .((((.--(((....((((----((((..(.(((.....(((((((((..(((.....)))..))))..))))).....))).)..))))))))((((...)))).))))))) ( -47.40) >DroYak_CAF1 3456 107 - 1 AAUUUA--GCUACCAGGAU----GACUUACCGCAUUAUCGGCUGGCCGGGUCUUUUGUAGAUACGGCACCAGCCACUUUUGCUGGAAGUACUCCGUGGUGGCCACACCCAUGU ......--((((((((((.----.((((.(((((.....(((((((((..(((.....)))..))))..))))).....))).))))))..))).)))))))........... ( -42.80) >DroMoj_CAF1 3539 108 - 1 UAUAGU--GAAAUAUGUUC---GCACUUACCGCAUUAUCGGAGGGUCGCGUUUUCUGUAGGUAGGGCACCAGCCACUUUUGCUGGAAGUAGUCGGUGCACGCCACAUCCAUGU ......--...(((((..(---(((((..(((......)))..))).))).....(((.(((...(((((.((.(((((.....))))).)).)))))..))))))..))))) ( -30.80) >DroAna_CAF1 5448 104 - 1 AGAAAA--GAGAUGAG-------GACUCACCGCAUUAUCGGUGGGACGGGUCUUUUGGAGAUAUGGCACUAGCCACUUCUGCUGGAAGUACUCGGUGGUGGCCACGCCCAUGU ......--...(((.(-------(.(((((((......))))))).(((((((((..((((..((((....))))..))).)..)))).)))))((((...)))).))))).. ( -39.60) >consensus AACAUU__GGGAUGAGGUG____CACUUACCGCAUUAUCGGCUGGCCGCGUCUUUUGCAGAUACGGCACCAGCCACUUUUGCUGGAAGUACUCCGUGGUGGCCACACCCAUGU ..........................(((((((......(((((((((.((((.....)))).)))..))))))(((((.....))))).....)))))))............ (-22.62 = -22.85 + 0.23)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:39:00 2006