
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 13,165,506 – 13,165,626 |
| Length | 120 |
| Max. P | 0.975407 |

| Location | 13,165,506 – 13,165,626 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.00 |
| Mean single sequence MFE | -44.53 |
| Consensus MFE | -37.05 |
| Energy contribution | -36.92 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.83 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.509898 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 13165506 120 + 22224390 AGACCCUGCGUCUCGUCCAGGCCUUCCAGUACACCGACAAGUACGGCGAGGUCUGCCCCGCCAACUGGAAGCCCGGCCAGAAGACCAUGGUGGCCGAUCCCACCAAGUCCAAGGAGUACU ..(((((...(((.(.((.(((.(((((((((........))))((((.((....)).))))...)))))))).))).))).(((..((((((......)))))).)))..))).))... ( -44.70) >DroVir_CAF1 2588 120 + 1 AGACACUGCGUCUGGUCCAGGCAUUCCAGUAUACCGACAAGUAUGGCGAGGUGUGCCCAGCCAACUGGAAGCCCGGCCAGAAGACCAUGGUGGCCGAUCCCACCAAGUCCAAGGAGUACU ....(((.(.(((((.((.(((.(((((((((((......))))(((..((....))..))).)))))))))).))))))).(((..((((((......)))))).)))...).)))... ( -49.60) >DroEre_CAF1 450 120 + 1 AGACCCUGCGUCUCGUUCAGGCCUUCCAGUACACCGACAAGUACGGCGAGGUCUGCCCCGCCAACUGGAAGCCCGGCCAGAAGACCAUGGUGGCCGAUCCCACCAAGUCCAAGGAGUACU ..(((((...(((.(..(.(((.(((((((((........))))((((.((....)).))))...)))))))).)..)))).(((..((((((......)))))).)))..))).))... ( -42.70) >DroYak_CAF1 449 120 + 1 AGACACUGCGUCUCGUUCAGGCCUUCCAGUACACCGAUAAGUACGGCGAGGUGUGCCCCGCCAACUGGAAGCCCGGCCAGAAGACCAUGGUGGCCGAUCCCACCAAGUCCAAGGAGUACU .(((..((.((((((..(.(((.(((((((((........))))((((.((....)).))))...)))))))).)..).).))))))((((((......)))))).)))........... ( -42.30) >DroMoj_CAF1 450 120 + 1 AGACACUGCGUCUAGUGCAGGCCUUCCAGUACACUGACAAGUAUGGCGAGGUGUGCCCAGCCAACUGGAAGCCCGGCCAGAAGACCAUGGUGGCCGAUCCCACCAAGUCCAAGGAGUACU ((((.....))))(((((.((.((((((((..(((....))).((((..((....))..)))))))))))).))..((....(((..((((((......)))))).)))...)).))))) ( -45.00) >DroAna_CAF1 450 120 + 1 AGACCAUCCGUUUGGUUCAGGCCUUCCAGUACACCGAUAAGUACGGUGAGGUGUGCCCCGCCAACUGGAAGCCCGGCCAGAAGACCAUGGUCGCUGAUCCCACCAAGUCCAAGGAGUACU .((((((...((((((...((.((((((((.(((((.......))))).((((.....)))).)))))))).)).)))))).....))))))..........((........))...... ( -42.90) >consensus AGACACUGCGUCUCGUUCAGGCCUUCCAGUACACCGACAAGUACGGCGAGGUGUGCCCCGCCAACUGGAAGCCCGGCCAGAAGACCAUGGUGGCCGAUCCCACCAAGUCCAAGGAGUACU .(((.....((((.(.((.(((.(((((((((........))))((((.((....)).))))...)))))))).)).)...))))..((((((......)))))).)))........... (-37.05 = -36.92 + -0.14)



| Location | 13,165,506 – 13,165,626 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.00 |
| Mean single sequence MFE | -51.47 |
| Consensus MFE | -46.50 |
| Energy contribution | -46.83 |
| Covariance contribution | 0.34 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.83 |
| Structure conservation index | 0.90 |
| SVM decision value | 1.75 |
| SVM RNA-class probability | 0.975407 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 13165506 120 - 22224390 AGUACUCCUUGGACUUGGUGGGAUCGGCCACCAUGGUCUUCUGGCCGGGCUUCCAGUUGGCGGGGCAGACCUCGCCGUACUUGUCGGUGUACUGGAAGGCCUGGACGAGACGCAGGGUCU ......(((((((((((((((......)))))).)))).(((.((((((((((((((.(((((((....))))))).((((....)))).))))))).)))))).).)))..)))))... ( -58.00) >DroVir_CAF1 2588 120 - 1 AGUACUCCUUGGACUUGGUGGGAUCGGCCACCAUGGUCUUCUGGCCGGGCUUCCAGUUGGCUGGGCACACCUCGCCAUACUUGUCGGUAUACUGGAAUGCCUGGACCAGACGCAGUGUCU ..((((.(..(((((((((((......)))))).)))))((((((((((((((((((.(((.(((....))).)))(((((....)))))))))))).)))))).))))).).))))... ( -55.40) >DroEre_CAF1 450 120 - 1 AGUACUCCUUGGACUUGGUGGGAUCGGCCACCAUGGUCUUCUGGCCGGGCUUCCAGUUGGCGGGGCAGACCUCGCCGUACUUGUCGGUGUACUGGAAGGCCUGAACGAGACGCAGGGUCU ......(((((((((((((((......)))))).)))).((((..((((((((((((.(((((((....))))))).((((....)))).))))))).)))))..).)))..)))))... ( -53.20) >DroYak_CAF1 449 120 - 1 AGUACUCCUUGGACUUGGUGGGAUCGGCCACCAUGGUCUUCUGGCCGGGCUUCCAGUUGGCGGGGCACACCUCGCCGUACUUAUCGGUGUACUGGAAGGCCUGAACGAGACGCAGUGUCU .((.(((...(((((((((((......)))))).)))))...(..((((((((((((.(((((((....))))))).((((....)))).))))))).)))))..))))))......... ( -50.60) >DroMoj_CAF1 450 120 - 1 AGUACUCCUUGGACUUGGUGGGAUCGGCCACCAUGGUCUUCUGGCCGGGCUUCCAGUUGGCUGGGCACACCUCGCCAUACUUGUCAGUGUACUGGAAGGCCUGCACUAGACGCAGUGUCU ..........((((.((((((......))))))((((((....((.((.((((((((.(((.(((....))).)))(((((....))))))))))))).)).))...)))).))..)))) ( -48.20) >DroAna_CAF1 450 120 - 1 AGUACUCCUUGGACUUGGUGGGAUCAGCGACCAUGGUCUUCUGGCCGGGCUUCCAGUUGGCGGGGCACACCUCACCGUACUUAUCGGUGUACUGGAAGGCCUGAACCAAACGGAUGGUCU .((..((((..........))))...))((((((((((....))))((.((((((((..((((((....))))((((.......)))))))))))))).))....((....)))))))). ( -43.40) >consensus AGUACUCCUUGGACUUGGUGGGAUCGGCCACCAUGGUCUUCUGGCCGGGCUUCCAGUUGGCGGGGCACACCUCGCCGUACUUGUCGGUGUACUGGAAGGCCUGAACGAGACGCAGGGUCU ......((..(((((((((((......)))))).)))))...)).((((((((((((((((((((....))))))))((((....)))).))))))).)))))....((((.....)))) (-46.50 = -46.83 + 0.34)

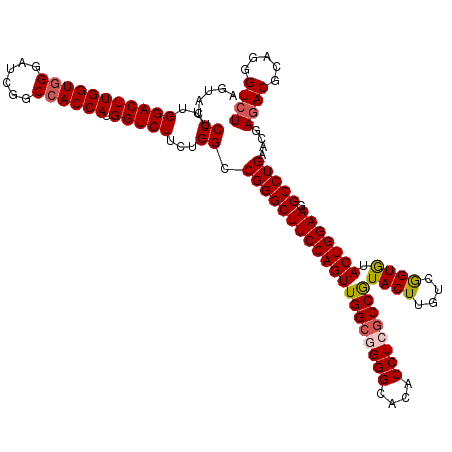
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:38:32 2006