
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 13,161,587 – 13,161,686 |
| Length | 99 |
| Max. P | 0.619778 |

| Location | 13,161,587 – 13,161,686 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | forward |
| Mean pairwise identity | 78.98 |
| Mean single sequence MFE | -34.08 |
| Consensus MFE | -25.26 |
| Energy contribution | -25.73 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.20 |
| Mean z-score | -1.09 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.619778 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 13161587 99 + 22224390 GU-GUGGCAC------AAACAGUUUACCUGCAGCUGCAGGAUCUGCUCAGUGACUAUGUGCAGCCAUGCGUCAUGGACUGCAAGGUGGGUGUGCGCACCUAUCUGG ..-(((((((------...(((....(((((....)))))..)))....))).)))).((((((((((...))))).)))))(((((((((....))))))))).. ( -40.60) >DroVir_CAF1 34954 99 + 1 CU-GUGUUAC------UUUCAGUCUAUUUGCAGCUGCAGGAUCUGCUUAGCGACUAUGUGCAGCCCUGCGUGAUGGACUGCAAAGUGGGUGUGCGCACCUAUCUGG ..-.....((------((((((((((((((((((((((.(...(((...)))....).))))))..)))).))))))))).)))))(((((....)))))...... ( -34.90) >DroGri_CAF1 26185 100 + 1 GC-CCGGCAU-U----UUGCAGUCUAUUUGCAGCUGCAGGAUCUGUUGAGCGACUACGUGCAGCCGUGCGUUAUGGACUGCAAGGUGGGCGUGCGCACCUAUCUGG ..-((((...-.----.((((((((((.((((((((((.(....(((....)))..).))))))..))))..))))))))))(((((.(....).)))))..)))) ( -42.50) >DroYak_CAF1 22947 96 + 1 ----------UACCACACACAGUUUAUUUGCAGCUGCAGGAUCUGCUCAGUGACUAUGUGCAGCCGUGCGUGAUGGACUGCAAGGUGGGCGUGCGCACCUAUCUGG ----------..(((....(((((((((((((((((((((.((........))))...))))))..)))).)))))))))..(((((.(....).)))))...))) ( -31.10) >DroMoj_CAF1 33317 99 + 1 UU-CGUUUAC------UUUCAGUUUAUCUGCAGCUGCAAGAUCUGCUCAGUGACUAUGUGCAGCCGUGCGUCAUGGAUUGCAAAGUGGGUGUGCGCACCUACCUGG ..-...((((------(...(((..((((((....)).))))..))).))))).....((((((((((...))))).)))))..(((((((....))))))).... ( -29.60) >DroAna_CAF1 26536 100 + 1 CUUGAAA---UUC---CUCCAGUGUAUCUGCAACUACAGGAUCUGUUGAGCGACUAUGUCCAGCCGUGUGUGAUGGACUGCAAGGUGGGUGUUCGCACUUAUCUGG .......---...---...((((.((((((((......((((..(((....)))...)))).....)))).)))).))))..(((((((((....))))))))).. ( -25.80) >consensus CU_GUGG_AC______UUACAGUUUAUCUGCAGCUGCAGGAUCUGCUCAGCGACUAUGUGCAGCCGUGCGUGAUGGACUGCAAGGUGGGUGUGCGCACCUAUCUGG ...................(((((((..((((((((((((.((........))))...))))))..))))...)))))))..(((((((((....))))))))).. (-25.26 = -25.73 + 0.48)

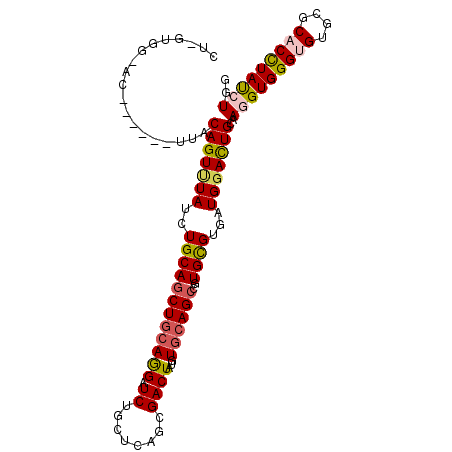
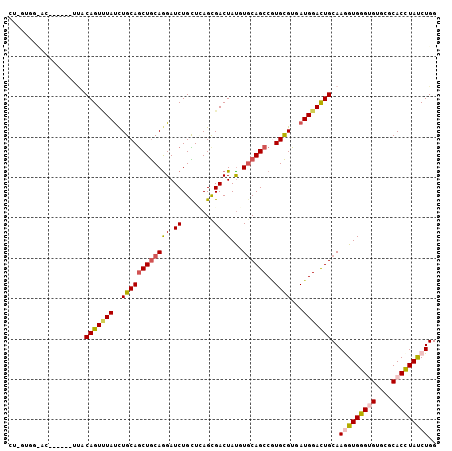
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:38:26 2006