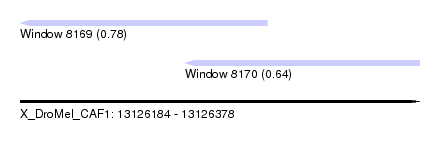
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 13,126,184 – 13,126,378 |
| Length | 194 |
| Max. P | 0.783876 |

| Location | 13,126,184 – 13,126,304 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.18 |
| Mean single sequence MFE | -41.43 |
| Consensus MFE | -26.41 |
| Energy contribution | -27.38 |
| Covariance contribution | 0.98 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.61 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.57 |
| SVM RNA-class probability | 0.783876 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 13126184 120 - 22224390 UGUGGAGCUGCAUUUGCAUUUGCAUCUGGAGCUGCAUCUGCGGCAUUGUCUGGCGCUGCAGCUCGAAGUUGUUCCGCUUGGCCAGGCGCUUCUGCUUCUUGAAGAACUUUCGGGCCACGU .(((((((.((...(((....)))....((((((((...(((.((.....)).)))))))))))...)).))))))).(((((.((..((((........))))..))....)))))... ( -48.30) >DroSec_CAF1 3911 120 - 1 UGUGGAGCUGCAUUUGCAUUUGCAUCUGGAGCUGCAUCUGCGGCAUUGUCUGGCGCUGCAGCUCGAAGUUGUUCCGCUUAGCCAGGCGCUUCUGCUUUUUAAAGAACUUUCGGGCCACGU .((((.((((((..((((.((.(....).)).))))..))))))...(((((((((.(((((.....)))))...))...)))))))((....))...................)))).. ( -41.40) >DroWil_CAF1 3915 102 - 1 U------CUGCAUUUGC---UGCAUAUG---------CUGCGGCAUGGUCUGACGCUGCAAUUCGAAAUUGUUCCGUUUGGCCAAUCGUUUUUGCUUCUUAAAGAACUUGCGGGCCACAU (------(((((..(((---((((....---------.)))))))(((((.((((..(((((.....)))))..)))).)))))...((((((........)))))).))))))...... ( -33.60) >DroYak_CAF1 4065 120 - 1 UGUGGAGCUGCAUUUGCAUUUGCAUCUGCAGCUGCAUCUGCGGCAUUGUCUGGCGCUGCAGCUCGAAGUUGUUCCGCUUGGCCAGGCGCUUCUGCUUCUUGAAGAACUUUCGGGCCACGU .(((((((((((..(((....)))..)))))))....(((((((..........)))))))(((((((((.(((.((((....))))((....)).....))).))).)))))))))).. ( -47.90) >DroMoj_CAF1 3939 93 - 1 UGUGCAUCU------------------------GCUG---GGGCAGCGCCUGGCGCUGCAGCUCGAAGUUGUUGCGCUUGGCCAGACGCUUCUGUUUCUUGAAGAACUUGCGCGCCACAU .((((((((------------------------((((---...))))(((.(((((.(((((.....))))).))))).))).)))..((((........))))....)))))....... ( -34.90) >DroAna_CAF1 4207 120 - 1 CAUGGAGCUGCAUCUGCAUCUGCAUCUGGAGCUGCAUCUGGGGCAUGGUCUGGCGCUGAAGCUCGAAGUUGUUCCGCUUGGCGAGGCGCUUCUGUUUCUUGAAGAACUUCCGGGCCACGU .(((.((((.((..(((....)))..)).)))).)))..((((((...((.(((......))).))...))))))((((((.(((...((((........))))..)))))))))..... ( -42.50) >consensus UGUGGAGCUGCAUUUGCAUUUGCAUCUGGAGCUGCAUCUGCGGCAUUGUCUGGCGCUGCAGCUCGAAGUUGUUCCGCUUGGCCAGGCGCUUCUGCUUCUUGAAGAACUUUCGGGCCACGU .(((.((((.((..(((....)))..)).)))).)))....(((...(((.((((..(((((.....)))))..)))).)))......((((........)))).........))).... (-26.41 = -27.38 + 0.98)

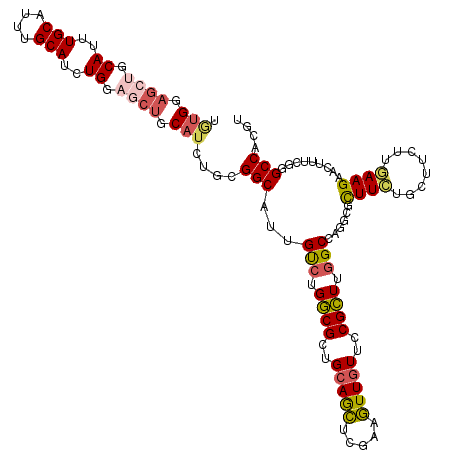
| Location | 13,126,264 – 13,126,378 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 82.38 |
| Mean single sequence MFE | -42.97 |
| Consensus MFE | -29.32 |
| Energy contribution | -31.67 |
| Covariance contribution | 2.35 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.21 |
| SVM RNA-class probability | 0.636195 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
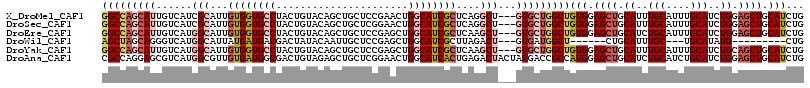
>X_DroMel_CAF1 13126264 114 - 22224390 GGCCAGCAUUGUCAUCGCAUUGUGGUGCCUACUGUACAGCUGCUCCGAACUGGCAUCGCUCAGGCU---GUGCUGGCUGUGGAGCUGCAUUUGCAUUUGCAUCUGGAGCUGCAUCUG (((((((((.(((........((((((((....(((....)))........))))))))...))).---)))))))))(((.((((.((..(((....)))..)).)))).)))... ( -44.30) >DroSec_CAF1 3991 114 - 1 GGCCAGCAUUGUCAUCGCAUUGUGGUGCCUACUGUACAGCUGCUCGGAACUGGCAUCGCUCAGGCU---GUGCUGGCUGUGGAGCUGCAUUUGCAUUUGCAUCUGGAGCUGCAUCUG (((((((((.(((........((((((((..(((..(....)..)))....))))))))...))).---)))))))))(((.((((.((..(((....)))..)).)))).)))... ( -46.70) >DroEre_CAF1 16716 114 - 1 GGCCAGCAUUGUCAUGGCAUUGUGGUGCCUACUGUACAGCUGCUCCGAGCUGGCAUCGCUCAAGCU---GUGCUGGCUGUGGAGCUGCAUCUGCAUUUGCAUCUGGAGCUGCAUCUG (((((((........(((((....)))))......((((((.....((((.......)))).))))---)))))))))(((.((((.((..(((....)))..)).)))).)))... ( -46.70) >DroWil_CAF1 3995 96 - 1 AGCUAGCAGGGUCAUGGCAUUAUGAUGACGACUAUACAAUUGCUCCGAGCUGGCAUCGCUUAGACU---GUGAUGGCU------CUGCAUUUGC---UGCAUAUG---------CUG (((((((((.(((((.(.....).))))).......(((.(((...((((...((((((.......---)))))))))------).))).))))---))).)).)---------)). ( -29.00) >DroYak_CAF1 4145 114 - 1 GGCCAGCAUUGUCAUGGCAUUGUGGUGCCUACUGUACAGCUGCUCCGAGCUGGCAUCGCUCAAGCU---GUGCUGGCUGUGGAGCUGCAUUUGCAUUUGCAUCUGCAGCUGCAUCUG (((((((........(((((....)))))......((((((.....((((.......)))).))))---)))))))))(((.(((((((..(((....)))..))))))).)))... ( -52.00) >DroAna_CAF1 4287 117 - 1 CGCCAGGAGCGUCAUGGCGUUGUGAUGGCGACUGUAGAGCUGCUCGGAACUGGCAUCACUGAGACUACUAUGACCGCCAUGGAGCUGCAUCUGCAUCUGCAUCUGGAGCUGCAUCUG ..((((..(((((((.((...)).)))))((.((((((((.((((.(...((((.(((..(......)..)))..))))).)))).)).)))))))).))..))))........... ( -39.10) >consensus GGCCAGCAUUGUCAUGGCAUUGUGGUGCCUACUGUACAGCUGCUCCGAACUGGCAUCGCUCAGGCU___GUGCUGGCUGUGGAGCUGCAUUUGCAUUUGCAUCUGGAGCUGCAUCUG (((((((((......(((...((((((((......................))))))))....)))...)))))))))(((.((((.((..(((....)))..)).)))).)))... (-29.32 = -31.67 + 2.35)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:38:14 2006