
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 13,101,315 – 13,101,430 |
| Length | 115 |
| Max. P | 0.710033 |

| Location | 13,101,315 – 13,101,430 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 81.94 |
| Mean single sequence MFE | -57.47 |
| Consensus MFE | -37.83 |
| Energy contribution | -38.33 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.69 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.14 |
| SVM RNA-class probability | 0.600602 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 13101315 115 + 22224390 CGGAUGUCCAAGGGGCAGUGGUGCCGACUGCGUUCGUCCCAGCGGCCGAUGGCCCUGUUUGUGCAGAUGCACCUGCGCCACCUGGGCGUCCGUUAUGGGCACAUUGUGCUGGUGG ((((((((((.(((((.(((((((...(((((..((...(((.((((...)))))))..)))))))..))))).))))).)))))))))))).(((.(((((...))))).))). ( -56.30) >DroVir_CAF1 7880 112 + 1 CGGAUGACCCAGGGGCAGCGGCGCCGACUGGGUGCGUCCGAGUGGCCGGUGGCCCUGCUUGUGCAGAUGCACCUGGGCCACCUGGGCGUCUGUGAUGGGCACAU---GCCCGGCG ((((((((((((.(((......)))..)))))).))))))....(((((((((((.....((((....))))..)))))))).((((((.(((.....))).))---))))))). ( -66.80) >DroPse_CAF1 3002 115 + 1 CGGAUGUCCCAGGGGCAGCGGGGCCGACUGAGUCCUGCCCAAGGGUCGGUGGCCCUGCUUGUGCAGAUGCACCUGAGCCACCUGUGCAUCCGUGAUUGGUGUGUUGUGCGGGUGU (((((((..((((((((((((((((.(((((.((((.....)))))))))))))))))).((((....))))....))).)))).)))))))..(((.((.......)).))).. ( -56.80) >DroMoj_CAF1 4377 112 + 1 GGGAUGGCCUAGGGGUAGGGGCGCCGAUUGGGUGCGCCCCAGUGGACGGUGGCCCUGCUUAUGCAGAUGCACCUGCGCCACCUGGGCGUCCGUGAUGGGCGCGU---GCGCGUGG .(((((.(((((((((((((.(((((((((((.....))))))...))))).)))))))...((((......))))....))))))))))).......((((..---..)))).. ( -57.90) >DroAna_CAF1 4794 115 + 1 CGGAUGUCCCAGGGGCAGUGGCGCCGACUGUGUCCUGCCCAGCGGCCGGUGGCCCUGCUUGUGAAGAUGCACCUGCGCCACCUGGGCAUCGGUGAUGGGAACAUUGUGCUGCUGC ..((((.((((.((((((.(((((.....))))))))))).......((((((...((..(((......)))..)))))))))))))))).(..(((....)))..)........ ( -50.20) >DroPer_CAF1 2993 115 + 1 CGGAUGUCCCAGGGGCAGCGGGGCCGACUGAGUCCUGCCCAAGGGUCGGUGGCCCUGCUUGUGCAGAUGCACCUGAGCCACCUGUGCAUCCGUGAUUGGUGUGUUGUGCGGGUGU (((((((..((((((((((((((((.(((((.((((.....)))))))))))))))))).((((....))))....))).)))).)))))))..(((.((.......)).))).. ( -56.80) >consensus CGGAUGUCCCAGGGGCAGCGGCGCCGACUGAGUCCGGCCCAGCGGCCGGUGGCCCUGCUUGUGCAGAUGCACCUGCGCCACCUGGGCAUCCGUGAUGGGCACAUUGUGCGGGUGG ((((((.((((((((((((((.(((.((((...(((......))).))))))).))))).((((....))))....))).))))))))))))....................... (-37.83 = -38.33 + 0.50)



| Location | 13,101,315 – 13,101,430 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 81.94 |
| Mean single sequence MFE | -49.21 |
| Consensus MFE | -30.89 |
| Energy contribution | -33.37 |
| Covariance contribution | 2.47 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.15 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.37 |
| SVM RNA-class probability | 0.710033 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 13101315 115 - 22224390 CCACCAGCACAAUGUGCCCAUAACGGACGCCCAGGUGGCGCAGGUGCAUCUGCACAAACAGGGCCAUCGGCCGCUGGGACGAACGCAGUCGGCACCACUGCCCCUUGGACAUCCG ......((((...))))......((((.(.(((((.((((..(((((..((((.....(((((((...)))).)))(......)))))...)))))..)))).))))).).)))) ( -47.50) >DroVir_CAF1 7880 112 - 1 CGCCGGGC---AUGUGCCCAUCACAGACGCCCAGGUGGCCCAGGUGCAUCUGCACAAGCAGGGCCACCGGCCACUCGGACGCACCCAGUCGGCGCCGCUGCCCCUGGGUCAUCCG .(((((((---.((((.....))))...)))).((((((((..((((....)))).....)))))))))))....((((.(.((((((..((((....)))).))))))).)))) ( -59.80) >DroPse_CAF1 3002 115 - 1 ACACCCGCACAACACACCAAUCACGGAUGCACAGGUGGCUCAGGUGCAUCUGCACAAGCAGGGCCACCGACCCUUGGGCAGGACUCAGUCGGCCCCGCUGCCCCUGGGACAUCCG .......................((((((....((((((((..((((....)))).....))))))))..(((..(((((((((...)))(....).))))))..))).)))))) ( -47.90) >DroMoj_CAF1 4377 112 - 1 CCACGCGC---ACGCGCCCAUCACGGACGCCCAGGUGGCGCAGGUGCAUCUGCAUAAGCAGGGCCACCGUCCACUGGGGCGCACCCAAUCGGCGCCCCUACCCCUAGGCCAUCCC ....((((---..(((((.(((..((....)).))))))))..))))..((((....))))((((..........(((((((.........)))))))........))))..... ( -46.67) >DroAna_CAF1 4794 115 - 1 GCAGCAGCACAAUGUUCCCAUCACCGAUGCCCAGGUGGCGCAGGUGCAUCUUCACAAGCAGGGCCACCGGCCGCUGGGCAGGACACAGUCGGCGCCACUGCCCCUGGGACAUCCG ((....)).................((((((((((.((((..(((((...........(((((((...)))).)))(((........))).)))))..)))))))))).)))).. ( -45.50) >DroPer_CAF1 2993 115 - 1 ACACCCGCACAACACACCAAUCACGGAUGCACAGGUGGCUCAGGUGCAUCUGCACAAGCAGGGCCACCGACCCUUGGGCAGGACUCAGUCGGCCCCGCUGCCCCUGGGACAUCCG .......................((((((....((((((((..((((....)))).....))))))))..(((..(((((((((...)))(....).))))))..))).)))))) ( -47.90) >consensus CCACCCGCACAACGCGCCCAUCACGGACGCCCAGGUGGCGCAGGUGCAUCUGCACAAGCAGGGCCACCGGCCACUGGGCAGGACCCAGUCGGCGCCGCUGCCCCUGGGACAUCCG .......................((((.(((((((.(((...(((((.(((((....))))).....((((...((((.....)))))))))))))...))))))))).).)))) (-30.89 = -33.37 + 2.47)

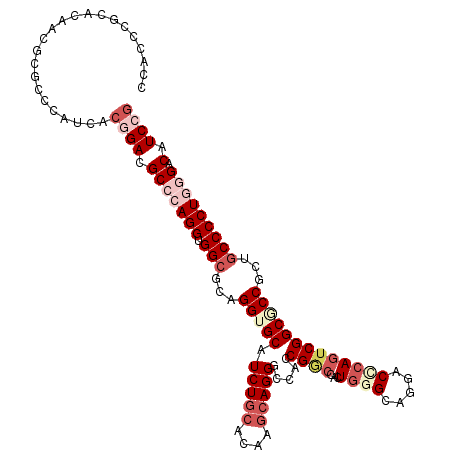
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:38:03 2006