
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 12,925,977 – 12,926,069 |
| Length | 92 |
| Max. P | 0.772358 |

| Location | 12,925,977 – 12,926,069 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | forward |
| Mean pairwise identity | 82.25 |
| Mean single sequence MFE | -26.81 |
| Consensus MFE | -17.82 |
| Energy contribution | -18.43 |
| Covariance contribution | 0.61 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.16 |
| SVM RNA-class probability | 0.613147 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
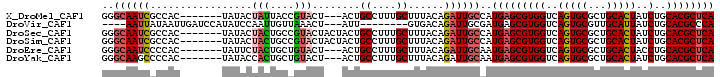
>X_DroMel_CAF1 12925977 92 + 22224390 GGGCAAUCGCCAC-------UAUACUAUUACCGUACU---ACUGCCUUUGCUUUACAGAUUGCCAUGAGCGUGGUCAGUGCGCUGCACUAUCUGCACGCUCA .(((((((.....-------..(((.......)))..---...((....))......))))))).(((((((((..(((((...)))))..)..)))))))) ( -28.00) >DroVir_CAF1 2797 87 + 1 ----AAUUAUAAUUGAUCCAUAUCCAAUUGUUAAACU---AUU--------GUGACAGAUUGCGAUGAGCGUGGUCAGUGCGUUGCAUUAUCUGCACGCCCA ----....(((((((.........)))))))......---...--------(((.((((((((((((.((.......)).)))))))..))))))))..... ( -20.50) >DroSec_CAF1 2654 95 + 1 GGGCAAUCGCCAC-------UAUACUACUGCCGUACUACUACUGCCUUUGCUUUACAGAUUGCCAUGAGCGUGGUCAGUGCGCUGCACUAUCUGCACGCUCA .(((((((.....-------.........((.(((....))).))............))))))).(((((((((..(((((...)))))..)..)))))))) ( -29.27) >DroSim_CAF1 2691 95 + 1 GGGCAAUCGCCAC-------UAUACUACUGCCGUACUACUACUGCCUUUGCUUUACAGAUUGCCAUGAGCGUGGUCAGUGCGCUGCACUAUCUGCACGCUCA .(((((((.....-------.........((.(((....))).))............))))))).(((((((((..(((((...)))))..)..)))))))) ( -29.27) >DroEre_CAF1 2618 92 + 1 GGGCAAUCCCCAC-------UAUUCUACUGCUGUACU---ACUGCCUUUGCUUUACAGAUUGCAAUGAGCGUGGUCAGUGCGCUGCACUACCUGCACGCUCA (((.....)))..-------.........((((.(((---((.(((.((((..........)))).).)))))))))))(((.((((.....)))))))... ( -26.50) >DroYak_CAF1 2569 92 + 1 GGGCAAGCCCCAC-------UAUACCACUGCUGUACU---ACUGCCUUUGCUUUACAGAUUGCAAUGAGCGUGGUCAGUGCGCUGCACUAUCUGCACGCUCA (((....)))...-------.........((((.(((---((.(((.((((..........)))).).)))))))))))(((.((((.....)))))))... ( -27.30) >consensus GGGCAAUCGCCAC_______UAUACUACUGCCGUACU___ACUGCCUUUGCUUUACAGAUUGCCAUGAGCGUGGUCAGUGCGCUGCACUAUCUGCACGCUCA ..((((((.................(((....)))........((....))......))))))..(((((((((..(((((...)))))..)..)))))))) (-17.82 = -18.43 + 0.61)
| Location | 12,925,977 – 12,926,069 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | reverse |
| Mean pairwise identity | 82.25 |
| Mean single sequence MFE | -30.23 |
| Consensus MFE | -22.18 |
| Energy contribution | -22.77 |
| Covariance contribution | 0.58 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.53 |
| SVM RNA-class probability | 0.772358 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 12925977 92 - 22224390 UGAGCGUGCAGAUAGUGCAGCGCACUGACCACGCUCAUGGCAAUCUGUAAAGCAAAGGCAGU---AGUACGGUAAUAGUAUA-------GUGGCGAUUGCCC ((((((((....((((((...))))))..)))))))).(((((((((.....))..(.((.(---(.(((.......)))))-------.)).)))))))). ( -33.10) >DroVir_CAF1 2797 87 - 1 UGGGCGUGCAGAUAAUGCAACGCACUGACCACGCUCAUCGCAAUCUGUCAC--------AAU---AGUUUAACAAUUGGAUAUGGAUCAAUUAUAAUU---- ...(((((((.....))).))))..((((((.((.....))....((((.(--------(((---.(.....).))))))))))).))).........---- ( -18.40) >DroSec_CAF1 2654 95 - 1 UGAGCGUGCAGAUAGUGCAGCGCACUGACCACGCUCAUGGCAAUCUGUAAAGCAAAGGCAGUAGUAGUACGGCAGUAGUAUA-------GUGGCGAUUGCCC ((((((((....((((((...))))))..)))))))).(((((((((.....))..(.((.((.((.(((....))).))))-------.)).)))))))). ( -35.10) >DroSim_CAF1 2691 95 - 1 UGAGCGUGCAGAUAGUGCAGCGCACUGACCACGCUCAUGGCAAUCUGUAAAGCAAAGGCAGUAGUAGUACGGCAGUAGUAUA-------GUGGCGAUUGCCC ((((((((....((((((...))))))..)))))))).(((((((((.....))..(.((.((.((.(((....))).))))-------.)).)))))))). ( -35.10) >DroEre_CAF1 2618 92 - 1 UGAGCGUGCAGGUAGUGCAGCGCACUGACCACGCUCAUUGCAAUCUGUAAAGCAAAGGCAGU---AGUACAGCAGUAGAAUA-------GUGGGGAUUGCCC ((((((((....((((((...))))))..))))))))..(((((((.((..((....))...---..(((....))).....-------.)).))))))).. ( -30.40) >DroYak_CAF1 2569 92 - 1 UGAGCGUGCAGAUAGUGCAGCGCACUGACCACGCUCAUUGCAAUCUGUAAAGCAAAGGCAGU---AGUACAGCAGUGGUAUA-------GUGGGGCUUGCCC ...((.((((.....)))))).((((((((((((.....))...(((((..((.......))---..)))))..))))).))-------)))(((....))) ( -29.30) >consensus UGAGCGUGCAGAUAGUGCAGCGCACUGACCACGCUCAUGGCAAUCUGUAAAGCAAAGGCAGU___AGUACAGCAGUAGUAUA_______GUGGCGAUUGCCC ((((((((....((((((...))))))..))))))))..((((((......((....)).......((((.......)))).............)))))).. (-22.18 = -22.77 + 0.58)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:36:45 2006