
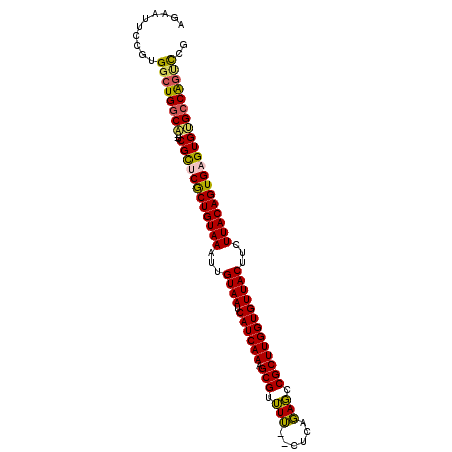
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 12,896,003 – 12,896,102 |
| Length | 99 |
| Max. P | 0.993983 |

| Location | 12,896,003 – 12,896,102 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 103 |
| Reading direction | forward |
| Mean pairwise identity | 85.42 |
| Mean single sequence MFE | -43.57 |
| Consensus MFE | -39.89 |
| Energy contribution | -39.45 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.22 |
| Mean z-score | -5.71 |
| Structure conservation index | 0.92 |
| SVM decision value | 2.44 |
| SVM RNA-class probability | 0.993983 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 12896003 99 + 22224390 AGAAUUCCGUGGCUGGCA--UCGCUCGCUGUAAAUUGUAAUCAUCAAAGCGUUUU--CUCAGAGCCGCUUGGUGUUACUUCUUACAGUGAGUGUGCCAGUCCG .......((.((((((((--.((((((((((((...((((.((((((.((((((.--....))).)))))))))))))...)))))))))))))))))))))) ( -47.00) >DroGri_CAF1 123742 91 + 1 ---------UGAAUGACGUCACGCCCGCUGUAAUAUGUAAUCAUCAAAGCGUGUAU-UU--UACUCGCUUGGUGUUACUUCUUACAGUGAGUGCGACGGCCAC ---------.......((((.(((.((((((((...((((.((((((.(((.(((.-..--))).)))))))))))))...)))))))).))).))))..... ( -35.80) >DroSec_CAF1 96848 99 + 1 AGAAUUCCGUGGCUGGCA--UCGCUCGCUGUAAAUUGUAAUCAUCAAAGCGUUUU--CUCAGAGCCGCUUGGUGUUACUUCUUACAGUGAGUGUGCCAGUCCG .......((.((((((((--.((((((((((((...((((.((((((.((((((.--....))).)))))))))))))...)))))))))))))))))))))) ( -47.00) >DroEre_CAF1 93949 99 + 1 AGAAUUCCGUGGCUGGCA--UCGCCCGCUGUAAAUUGUAAUCAUCAAAGCGUUUU--CUCAGAGCCGCUUGGUGUUACUUCUUACAGUGAGUGUGCCAGUCCG .......((.((((((((--.(((.((((((((...((((.((((((.((((((.--....))).)))))))))))))...)))))))).))))))))))))) ( -42.20) >DroYak_CAF1 105539 99 + 1 AGAAUUCCGUGGCUGGCA--UCGCUCGCUGUAAAUUGUAAUCAUCAAAGCGUUUU--CUCAGAGCCGCUUGGUGUUACUUCUUACAGUGAGUGUGCCAGUCCG .......((.((((((((--.((((((((((((...((((.((((((.((((((.--....))).)))))))))))))...)))))))))))))))))))))) ( -47.00) >DroAna_CAF1 108583 101 + 1 AAAAUUCCGUGGCUGGCA--UCGUUCACUGUAAAUUGUAACCAUCAAAGCGUUUUUUAUCAGAGCCGCUUGGUGUUACUUCUUACAGUGAGUGAGCCAGCUGG ..........(((((((.--(((((((((((((...(((((.(((((.(((.(((......))).)))))))))))))...)))))))))))))))))))).. ( -42.40) >consensus AGAAUUCCGUGGCUGGCA__UCGCUCGCUGUAAAUUGUAAUCAUCAAAGCGUUUU__CUCAGAGCCGCUUGGUGUUACUUCUUACAGUGAGUGUGCCAGUCCG ..........((((((((...((((((((((((...((((.((((((.(((.(((......))).)))))))))))))...)))))))))))))))))))).. (-39.89 = -39.45 + -0.44)


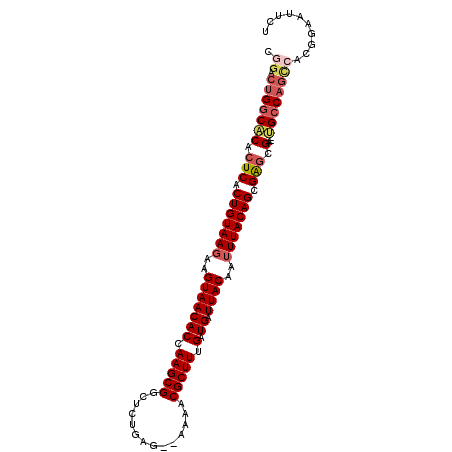
| Location | 12,896,003 – 12,896,102 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 103 |
| Reading direction | reverse |
| Mean pairwise identity | 85.42 |
| Mean single sequence MFE | -35.21 |
| Consensus MFE | -25.61 |
| Energy contribution | -26.56 |
| Covariance contribution | 0.95 |
| Combinations/Pair | 1.10 |
| Mean z-score | -3.99 |
| Structure conservation index | 0.73 |
| SVM decision value | 1.14 |
| SVM RNA-class probability | 0.921720 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 12896003 99 - 22224390 CGGACUGGCACACUCACUGUAAGAAGUAACACCAAGCGGCUCUGAG--AAAACGCUUUGAUGAUUACAAUUUACAGCGAGCGA--UGCCAGCCACGGAAUUCU .((.(((((((.(((.(((((((..(((((((.(((((........--....))))).).)).))))..))))))).))).).--)))))))).......... ( -35.50) >DroGri_CAF1 123742 91 - 1 GUGGCCGUCGCACUCACUGUAAGAAGUAACACCAAGCGAGUA--AA-AUACACGCUUUGAUGAUUACAUAUUACAGCGGGCGUGACGUCAUUCA--------- (((((.(((((.(((.((((((...(((((((.(((((.(((--..-.))).))))).).)).))))...)))))).))).))))))))))...--------- ( -33.50) >DroSec_CAF1 96848 99 - 1 CGGACUGGCACACUCACUGUAAGAAGUAACACCAAGCGGCUCUGAG--AAAACGCUUUGAUGAUUACAAUUUACAGCGAGCGA--UGCCAGCCACGGAAUUCU .((.(((((((.(((.(((((((..(((((((.(((((........--....))))).).)).))))..))))))).))).).--)))))))).......... ( -35.50) >DroEre_CAF1 93949 99 - 1 CGGACUGGCACACUCACUGUAAGAAGUAACACCAAGCGGCUCUGAG--AAAACGCUUUGAUGAUUACAAUUUACAGCGGGCGA--UGCCAGCCACGGAAUUCU .((.(((((((.(((.(((((((..(((((((.(((((........--....))))).).)).))))..))))))).))).).--)))))))).......... ( -34.60) >DroYak_CAF1 105539 99 - 1 CGGACUGGCACACUCACUGUAAGAAGUAACACCAAGCGGCUCUGAG--AAAACGCUUUGAUGAUUACAAUUUACAGCGAGCGA--UGCCAGCCACGGAAUUCU .((.(((((((.(((.(((((((..(((((((.(((((........--....))))).).)).))))..))))))).))).).--)))))))).......... ( -35.50) >DroAna_CAF1 108583 101 - 1 CCAGCUGGCUCACUCACUGUAAGAAGUAACACCAAGCGGCUCUGAUAAAAAACGCUUUGAUGGUUACAAUUUACAGUGAACGA--UGCCAGCCACGGAAUUUU ((.((((((((..((((((((((..(((((.(.(((((..............))))).)...)))))..))))))))))..))--.))))))...))...... ( -36.64) >consensus CGGACUGGCACACUCACUGUAAGAAGUAACACCAAGCGGCUCUGAG__AAAACGCUUUGAUGAUUACAAUUUACAGCGAGCGA__UGCCAGCCACGGAAUUCU .((.(((((((.(((.(((((((..(((((((.(((((..............))))).).)).))))..))))))).))).)...)))))))).......... (-25.61 = -26.56 + 0.95)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:36:33 2006