| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 12,694,171 – 12,694,327 |
| Length | 156 |
| Max. P | 0.987658 |

| Location | 12,694,171 – 12,694,287 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 71.62 |
| Mean single sequence MFE | -31.75 |
| Consensus MFE | -10.63 |
| Energy contribution | -11.37 |
| Covariance contribution | 0.74 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.00 |
| Structure conservation index | 0.33 |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.630028 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 12694171 116 - 22224390 CCGAUACAC-ACACUUGCACAC-GUUCAUACAUGUACAUGUAUGUAGAUGUGCUGGCGUGUGGAAAUUAUGGUUUUGUAAUUGGCAUAUCGCUGUCUCUAUCGCCAAACAGCUGGCCG ..(((((((-((.((.((((((-...(((((((....)))))))..).))))).)).)))))..((((((......))))))((((......))))..))))((((......)))).. ( -35.50) >DroSec_CAF1 47107 112 - 1 CCGAUACAC-ACACUUACACAC-GUAUGUACAU----AUGUAUGUGCAUGUGCUGGCGUGUGGAAAUUAUGGUUUUGUAAUUGGCAUAUCGCUGUCUCUAUCGCCAAACAGCUGGCCG ..(((((((-((.((..((((.-((((((((..----..)))))))).))))..)).)))))..((((((......))))))((((......))))..))))((((......)))).. ( -33.40) >DroSim_CAF1 33218 112 - 1 CCGAUACAC-ACACUUACACAC-GUAUGUACAU----AUGUAUGUGCAUGUGCUGGCGUGUGGAAAUUAUGGUUUUGUAAUUGGGAUAUCGCUGUCUCUAUCGCCAAACAGCUGGCCG ..(((((((-((.((..((((.-((((((((..----..)))))))).))))..)).)))))..((((((......))))))((((((....))))))))))((((......)))).. ( -35.50) >DroEre_CAF1 46404 98 - 1 UCGGU--AC-ACGCAUACACUC-GUACAUA----------------AAUGUGCUGGCGUGUGGAAAUUAUGGUUUUGUAAUUGGCAUAUCGCUGUCUCUAUCGCCAAACAUCUAACCG .((((--..-............-(((((..----------------..)))))(((((.(((((((((((......))))))((((......))))))))))))))........)))) ( -23.50) >DroYak_CAF1 50691 100 - 1 CCGAUCCAC-ACGCAUACAAAG-GUACAUA----------------CAUGUGCUGGCGUGUGGAAAUUAUGGUUUUGUAAUUGGCAUAUCGCUAUCUCUAUCGCCAUACAUCUGGCCA ....(((((-((((.......(-(((((..----------------..)))))).))))))))).....(((((.((((..(((((((..........))).))))))))...))))) ( -32.10) >DroPer_CAF1 75609 93 - 1 UCGCUACUGAAAGCUUGUAGGAGCCAGACA----------------GAUGUGU---CGGGUGCGCGUUAUACUUUUAUAAUUGCCACUGCUACGCAUCUAU------ACAUCUGUGCG .(((..(((...((((....)))))))(((----------------(((((((---.(((((((.((((((....)))))).((....))..)))))))))------))))))))))) ( -30.50) >consensus CCGAUACAC_ACACUUACACAC_GUACAUA________________CAUGUGCUGGCGUGUGGAAAUUAUGGUUUUGUAAUUGGCAUAUCGCUGUCUCUAUCGCCAAACAGCUGGCCG ................................................(((..(((((.(((((((((((......))))))(((.....)))...)))))))))).)))........ (-10.63 = -11.37 + 0.74)

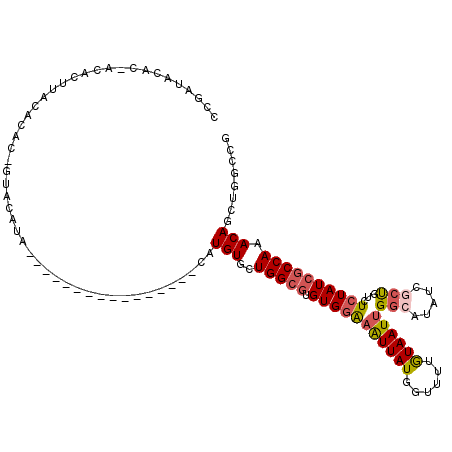
| Location | 12,694,209 – 12,694,327 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 84.62 |
| Mean single sequence MFE | -29.90 |
| Consensus MFE | -22.20 |
| Energy contribution | -24.18 |
| Covariance contribution | 1.98 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.88 |
| Structure conservation index | 0.74 |
| SVM decision value | 2.09 |
| SVM RNA-class probability | 0.987658 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 12694209 118 + 22224390 UUACAAAACCAUAAUUUCCACACGCCAGCACAUCUACAUACAUGUACAUGUAUGAACGUGUGCAAGUGUGUGUAUCGGGAUUUUUUGGCCAUGGACUGCAUUCAAAAUGCAAUUUGUU ..(((((.(((.(((..((((((((..((((((...(((((((....)))))))...))))))..)))))))....)..)))...)))........(((((.....))))).))))). ( -36.50) >DroSec_CAF1 47145 114 + 1 UUACAAAACCAUAAUUUCCACACGCCAGCACAUGCACAUACAU----AUGUACAUACGUGUGUAAGUGUGUGUAUCGGGUUUUUUUGGCCAUGGACUGCAUUCAAAAUGCAAUUUGUU ..(((((.((((......(((((((..(((((((....(((..----..)))....)))))))..))))))).....((((.....))))))))..(((((.....))))).))))). ( -32.80) >DroSim_CAF1 33256 114 + 1 UUACAAAACCAUAAUUUCCACACGCCAGCACAUGCACAUACAU----AUGUACAUACGUGUGUAAGUGUGUGUAUCGGGAUUUUUUGGCCAUGGACUGCAUUCAAAAUGCAAUUUGUU ..(((((.........((((...(((((.((((((((.(((((----((((....))))))))).))))))))...........)))))..)))).(((((.....))))).))))). ( -32.00) >DroEre_CAF1 46442 100 + 1 UUACAAAACCAUAAUUUCCACACGCCAGCACAUU----------------UAUGUACGAGUGUAUGCGUGU--ACCGAGAUUUUUUGUCCAUGGACUGCAUUCAAAAUGCAAUUUGUU ..(((((.....((((((.((((((..((((...----------------.........))))..))))))--...))))))....(((....)))(((((.....))))).))))). ( -22.30) >DroYak_CAF1 50729 102 + 1 UUACAAAACCAUAAUUUCCACACGCCAGCACAUG----------------UAUGUACCUUUGUAUGCGUGUGGAUCGGAAUUUUUUGGCCAUGGACAGCAUUCAAAAUGCAAUUUGUU ..(((((.((((....(((((((((..(((...(----------------(....))...)))..)))))))))..((..........))))))...((((.....))))..))))). ( -25.90) >consensus UUACAAAACCAUAAUUUCCACACGCCAGCACAUG_ACAUACAU____AUGUAUGUACGUGUGUAAGUGUGUGUAUCGGGAUUUUUUGGCCAUGGACUGCAUUCAAAAUGCAAUUUGUU ..(((((.(((.(((((((((((((..(((((((....(((((....)))))....)))))))..)))))))....))))))...))).........((((.....))))..))))). (-22.20 = -24.18 + 1.98)

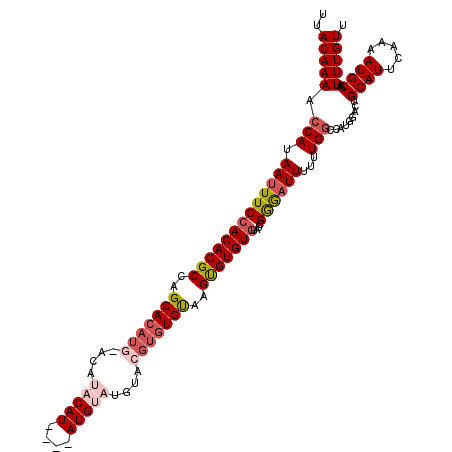

| Location | 12,694,209 – 12,694,327 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 84.62 |
| Mean single sequence MFE | -30.00 |
| Consensus MFE | -16.66 |
| Energy contribution | -17.28 |
| Covariance contribution | 0.62 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.54 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.25 |
| SVM RNA-class probability | 0.651981 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 12694209 118 - 22224390 AACAAAUUGCAUUUUGAAUGCAGUCCAUGGCCAAAAAAUCCCGAUACACACACUUGCACACGUUCAUACAUGUACAUGUAUGUAGAUGUGCUGGCGUGUGGAAAUUAUGGUUUUGUAA .(((((((((((.....)))))))((((((..........))....(((((.((.((((((...(((((((....)))))))..).))))).)).)))))......))))..)))).. ( -34.10) >DroSec_CAF1 47145 114 - 1 AACAAAUUGCAUUUUGAAUGCAGUCCAUGGCCAAAAAAACCCGAUACACACACUUACACACGUAUGUACAU----AUGUAUGUGCAUGUGCUGGCGUGUGGAAAUUAUGGUUUUGUAA .....(((((((.....)))))))...........((((((.(((.(((((.((..((((.((((((((..----..)))))))).))))..)).)))))...)))..)))))).... ( -32.20) >DroSim_CAF1 33256 114 - 1 AACAAAUUGCAUUUUGAAUGCAGUCCAUGGCCAAAAAAUCCCGAUACACACACUUACACACGUAUGUACAU----AUGUAUGUGCAUGUGCUGGCGUGUGGAAAUUAUGGUUUUGUAA .(((((((((((.....)))))))((((((..........))....(((((.((..((((.((((((((..----..)))))))).))))..)).)))))......))))..)))).. ( -32.00) >DroEre_CAF1 46442 100 - 1 AACAAAUUGCAUUUUGAAUGCAGUCCAUGGACAAAAAAUCUCGGU--ACACGCAUACACUCGUACAUA----------------AAUGUGCUGGCGUGUGGAAAUUAUGGUUUUGUAA .(((((((((((.....)))))))((((((...........((..--...))(((((.((.(((((..----------------..))))).)).)))))....))))))..)))).. ( -23.40) >DroYak_CAF1 50729 102 - 1 AACAAAUUGCAUUUUGAAUGCUGUCCAUGGCCAAAAAAUUCCGAUCCACACGCAUACAAAGGUACAUA----------------CAUGUGCUGGCGUGUGGAAAUUAUGGUUUUGUAA .(((((..((((.....))))...((((((..........))..(((((((((.......((((((..----------------..)))))).)))))))))....)))).))))).. ( -28.30) >consensus AACAAAUUGCAUUUUGAAUGCAGUCCAUGGCCAAAAAAUCCCGAUACACACACUUACACACGUACAUACAU____AUGUAUGU_CAUGUGCUGGCGUGUGGAAAUUAUGGUUUUGUAA .(((((((((((.....))))))((((((((((.........(.....)............((((((.(((..........))).)))))))))).)))))).........))))).. (-16.66 = -17.28 + 0.62)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:35:16 2006