
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 1,415,731 – 1,415,838 |
| Length | 107 |
| Max. P | 0.562460 |

| Location | 1,415,731 – 1,415,838 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.78 |
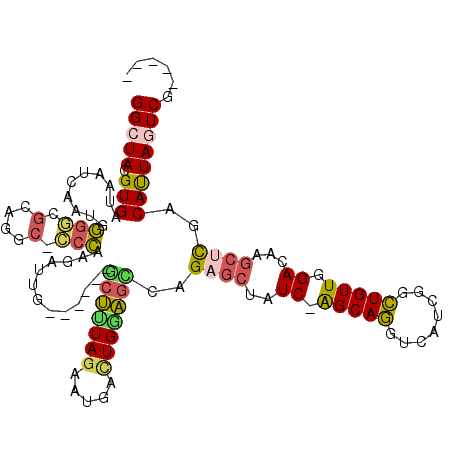
| Mean single sequence MFE | -34.98 |
| Consensus MFE | -18.44 |
| Energy contribution | -18.53 |
| Covariance contribution | 0.09 |
| Combinations/Pair | 1.40 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.53 |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.562460 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
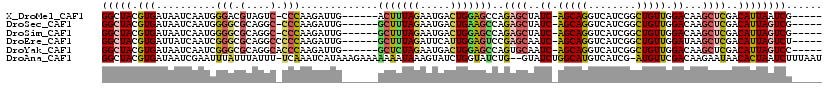
>X_DroMel_CAF1 1415731 107 + 22224390 GGCUACGUGAUAAUCAAUGGGACGUAGUC-CCCAAGAUUG------ACUUUAGAAUGACUGGAGCCAGAGCUAUC-AGCAGGUCAUCGGCUGUUGGACAAGCUCGACAUUAAUCG----- ((((((((..((.....))..))))))))-.....(((((------(((((((.....))))))...(((((.((-(((((........)))))))...)))))....)))))).----- ( -32.00) >DroSec_CAF1 22181 107 + 1 GGCUACGUGAUAAUCAAUGGGGCGCAGGC-CCCAAGAUUG------GCUUUAGAAUGACUGAAGCCAGAGCUAUC-AGCAGGUCAUCGGCUGUUGGACAAGCUCGACAUUAGUCG----- (((((.(((..((((..((((((....))-)))).))))(------(((((((.....)))))))).(((((.((-(((((........)))))))...)))))..)))))))).----- ( -44.80) >DroSim_CAF1 12799 107 + 1 GGCUACGUGAUAAUCAAUGGGGCGCAGGC-CCCAAGAUUG------GCUUUAGAAUGACUGGAGCCAGAGCUAUC-AGCAGGUCAUCGGCUGUUGGACAAGCUCGACAUUAGUCG----- (((((.(((..((((..((((((....))-)))).))))(------(((((((.....)))))))).(((((.((-(((((........)))))))...)))))..)))))))).----- ( -44.40) >DroEre_CAF1 17508 108 + 1 GGCUACGUGAUUAUCAAUCGGGCGCAGGCCCCCAAGAUUG------GCUUUAGAUUCAUUGGAGUCCGAGCAAUC-AGCAGGUCAUCGGCUGUUGGAUAAGCUCGACAUUAGUCU----- (((((.(((....((((((((((....))))....)))))------).....(((((....)))))(((((..((-(((((........)))))))....))))).)))))))).----- ( -34.50) >DroYak_CAF1 13714 108 + 1 GGCUACGUGAUAAUCAAUCGGGCGCAGGCACCCAAGAUUG------GCUCUAGAAUGACUGGAGCCAGUGCAAUC-AGCAGGUCAUCGGCUGUUGGACAAGCUCGACAUUAGUCC----- (((((.(((........((((((((.((...))...((((------(((((((.....)))))))))))))..((-(((((........)))))))....)))))))))))))).----- ( -38.30) >DroAna_CAF1 31923 116 + 1 GGCUACGUGAUAAUCGAAUUUAUUUAUUU-UCAAAUCAUAAAGAAAAAAAUAAAGUAUCUGGUAUCUG--GUAUCUGGCAUGUCAUCG-AUGUUCGACAAGAAUAACACUAAUCUUUAAU (..((.(((....((((.(((((((.(((-((..........)))))))))))).....((..(((..--(...)..).))..)))))-)(((((.....))))).)))))..)...... ( -15.90) >consensus GGCUACGUGAUAAUCAAUGGGGCGCAGGC_CCCAAGAUUG______GCUUUAGAAUGACUGGAGCCAGAGCUAUC_AGCAGGUCAUCGGCUGUUGGACAAGCUCGACAUUAGUCG_____ (((((.(((..........(((.(....).))).............(((((((.....)))))))..((((..((.(((((........))))).))...))))..))))))))...... (-18.44 = -18.53 + 0.09)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:45:46 2006