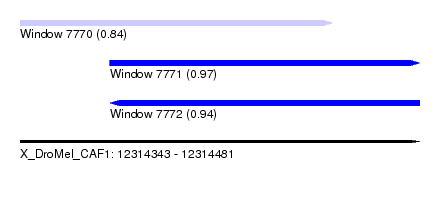
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 12,314,343 – 12,314,481 |
| Length | 138 |
| Max. P | 0.969414 |

| Location | 12,314,343 – 12,314,451 |
|---|---|
| Length | 108 |
| Sequences | 5 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 83.79 |
| Mean single sequence MFE | -32.96 |
| Consensus MFE | -24.70 |
| Energy contribution | -26.78 |
| Covariance contribution | 2.08 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.75 |
| SVM RNA-class probability | 0.841969 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
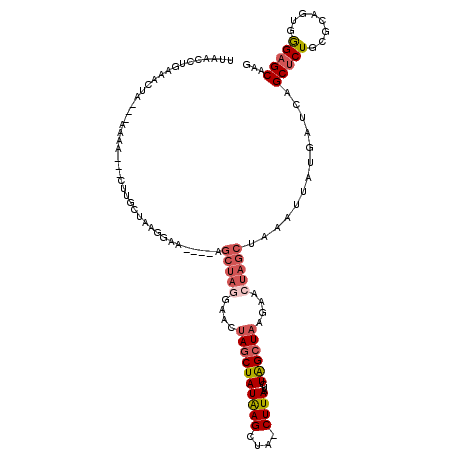
>X_DroMel_CAF1 12314343 108 + 22224390 GCGCACGCGCUGCCGUUGUCGCGCUAUCUCGCUUGCUCCCACUGCGCAGAGCUGAACAUAAUUUAGCUAGUUCUUAGCUAGAUUAAG-UAGCUCAUAGCUAGUUCCUAG-- (((((.((((.((....)).))))......(....)......))))).((((((....((((((((((((...))))))))))))..-))))))...............-- ( -33.50) >DroSec_CAF1 16669 109 + 1 GCGCACGCGCUGCCGUGUUCGCGCUAUCUCGCUUGCUCCCACUGCGCAGAGCUGCGCAUA-UCUAGCUAGUUCUUAGCCAGAUUAAG-UAGCUUAUAGCUAGUUCCUAGCG (((((((((....)))))..(((......)))...........)))).(((((((....(-(((.(((((...))))).))))...)-))))))...(((((...))))). ( -36.00) >DroSim_CAF1 17165 110 + 1 GCGCACGCGCUGCCGUUUUCGCGCUAUCUCGCUUGCUCCCACUGCGCAGAGCUGCUCAUAAUUUAGCUAGUUCUUAGCCAGAUUAAG-UAGCUUAUAGCUAGUUUCUAGCG (((((.((((..........))))......(....)......))))).((((((((..((((((.(((((...))))).))))))))-))))))...(((((...))))). ( -34.00) >DroEre_CAF1 16870 92 + 1 GCGCACGCGCUG--------------UCUCGCUCGCGCCCGCUGCGCAGAGCUGAUCAUAAUUUAGCUAGUUCUUAGCUAGAUUAAGCUAGCUUAUAGUUUAUA-----CU (((((.((((..--------------........))))....))))).((((((..(.((((((((((((...)))))))))))).).))))))..........-----.. ( -29.20) >DroYak_CAF1 16207 110 + 1 GCGCACACGCUGCCUUUUUCGCGCCAUCUCGCUUGCUCUCACUGCGCAGAGCUGAUCAUAAUUUAGCUAGUUCUUAGCUAGAUUAAG-UAGCUCAUAGCUAGUUCUUAGCU (((((......((.......(((......)))..))......))))).((((((....((((((((((((...))))))))))))..-))))))..((((((...)))))) ( -32.10) >consensus GCGCACGCGCUGCCGUUUUCGCGCUAUCUCGCUUGCUCCCACUGCGCAGAGCUGAUCAUAAUUUAGCUAGUUCUUAGCUAGAUUAAG_UAGCUUAUAGCUAGUUCCUAGCG (((((.((((..........))))......(....)......))))).((((((....((((((((((((...))))))))))))...))))))...(((((...))))). (-24.70 = -26.78 + 2.08)

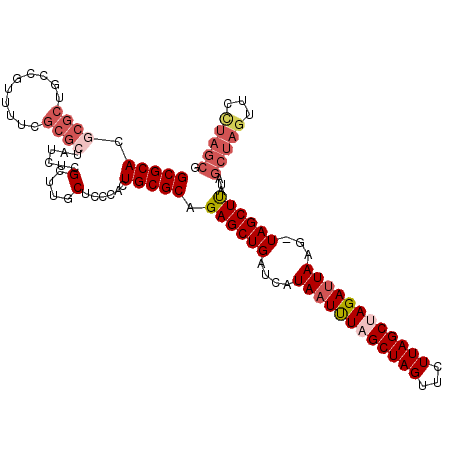

| Location | 12,314,374 – 12,314,481 |
|---|---|
| Length | 107 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.43 |
| Mean single sequence MFE | -27.80 |
| Consensus MFE | -18.40 |
| Energy contribution | -19.28 |
| Covariance contribution | 0.88 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.16 |
| Structure conservation index | 0.66 |
| SVM decision value | 1.64 |
| SVM RNA-class probability | 0.969414 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 12314374 107 + 22224390 CUUGCUCCCACUGCGCAGAGCUGAACAUAAUUUAGCUAGUUCUUAGCUAGAUUAAG-UAGCUCAUAGCUAGUUCCUAG------UUCCUUAUUUAG---UUUU---UAGUUUCAGCUUAA ...((.......))((.((((((....((((((((((((...))))))))))))..-))))))..((((((...((((------........))))---...)---)))))...)).... ( -27.20) >DroSec_CAF1 16700 111 + 1 CUUGCUCCCACUGCGCAGAGCUGCGCAUA-UCUAGCUAGUUCUUAGCCAGAUUAAG-UAGCUUAUAGCUAGUUCCUAGCGUC-GUUCCUUAGAAAG---UUUU---UAGUUUCAGGUUAG ((.(((.....((((((....))))))..-...))).))....(((((((((((((-.(((((...(((((...))))).((-........)))))---))))---))))))..))))). ( -27.90) >DroSim_CAF1 17196 109 + 1 CUUGCUCCCACUGCGCAGAGCUGCUCAUAAUUUAGCUAGUUCUUAGCCAGAUUAAG-UAGCUUAUAGCUAGUUUCUAGCG----UUCCUUAGAAAG---UUUU---UAGUUUCAGGCUUA ...((.......))((.((((((((..((((((.(((((...))))).))))))))-))))))...(((((...))))).----.......((((.---(...---.).))))..))... ( -26.10) >DroEre_CAF1 16887 108 + 1 CUCGCGCCCGCUGCGCAGAGCUGAUCAUAAUUUAGCUAGUUCUUAGCUAGAUUAAGCUAGCUUAUAGUUUAUA-----CUAUAGUUUUUCUGCAAGAAAUUUUGCGUAAUUUU------- ..(((((.....))(((((((((..(.((((((((((((...)))))))))))).).))))(((((((....)-----))))))....)))))..........))).......------- ( -29.70) >DroYak_CAF1 16238 113 + 1 CUUGCUCUCACUGCGCAGAGCUGAUCAUAAUUUAGCUAGUUCUUAGCUAGAUUAAG-UAGCUCAUAGCUAGUUCUUAGCUUUAAGUUCUCUUCAAG---UUUU---UAGUUUUAAGGUAA ..(((....(((((...((((((....((((((((((((...))))))))))))..-))))))...)).))).(((((..(((((..((.....))---..))---)))..)))))))). ( -28.10) >consensus CUUGCUCCCACUGCGCAGAGCUGAUCAUAAUUUAGCUAGUUCUUAGCUAGAUUAAG_UAGCUUAUAGCUAGUUCCUAGCG____UUCCUUAGAAAG___UUUU___UAGUUUCAGGUUAA ...(((...(((((...((((((....((((((((((((...))))))))))))...))))))...)).)))....)))......................................... (-18.40 = -19.28 + 0.88)

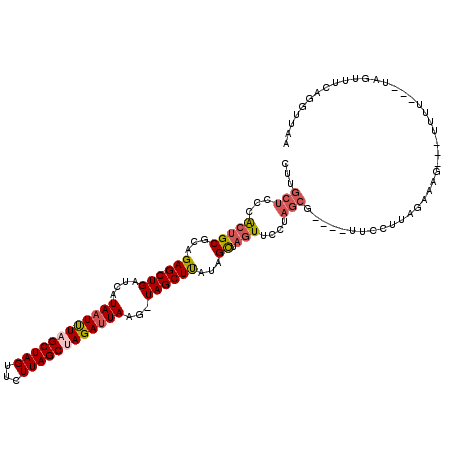
| Location | 12,314,374 – 12,314,481 |
|---|---|
| Length | 107 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.43 |
| Mean single sequence MFE | -26.68 |
| Consensus MFE | -13.48 |
| Energy contribution | -14.64 |
| Covariance contribution | 1.16 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.24 |
| Structure conservation index | 0.51 |
| SVM decision value | 1.27 |
| SVM RNA-class probability | 0.938222 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 12314374 107 - 22224390 UUAAGCUGAAACUA---AAAA---CUAAAUAAGGAA------CUAGGAACUAGCUAUGAGCUA-CUUAAUCUAGCUAAGAACUAGCUAAAUUAUGUUCAGCUCUGCGCAGUGGGAGCAAG ...(((((...(((---....---((.....))...------.)))....))))).(((((..-..((((.((((((.....)))))).)))).)))))(((((........)))))... ( -24.20) >DroSec_CAF1 16700 111 - 1 CUAACCUGAAACUA---AAAA---CUUUCUAAGGAAC-GACGCUAGGAACUAGCUAUAAGCUA-CUUAAUCUGGCUAAGAACUAGCUAGA-UAUGCGCAGCUCUGCGCAGUGGGAGCAAG ....(((((((...---....---.))))..)))...-...(((((...))))).....(((.-(((((((((((((.....))))))))-).((((((....)))))).)))))))... ( -33.20) >DroSim_CAF1 17196 109 - 1 UAAGCCUGAAACUA---AAAA---CUUUCUAAGGAA----CGCUAGAAACUAGCUAUAAGCUA-CUUAAUCUGGCUAAGAACUAGCUAAAUUAUGAGCAGCUCUGCGCAGUGGGAGCAAG ....(((((((...---....---.))))..)))..----.(((((...))))).....(((.-..((((.((((((.....)))))).))))..))).(((((........)))))... ( -25.40) >DroEre_CAF1 16887 108 - 1 -------AAAAUUACGCAAAAUUUCUUGCAGAAAAACUAUAG-----UAUAAACUAUAAGCUAGCUUAAUCUAGCUAAGAACUAGCUAAAUUAUGAUCAGCUCUGCGCAGCGGGCGCGAG -------.......(((..........(((((.....(((((-----(....)))))).(((..(.((((.((((((.....)))))).)))).)...))))))))((.....))))).. ( -27.40) >DroYak_CAF1 16238 113 - 1 UUACCUUAAAACUA---AAAA---CUUGAAGAGAACUUAAAGCUAAGAACUAGCUAUGAGCUA-CUUAAUCUAGCUAAGAACUAGCUAAAUUAUGAUCAGCUCUGCGCAGUGAGAGCAAG ..............---....---.................(((....(((.((...(((((.-..((((.((((((.....)))))).)))).....))))).))..)))...)))... ( -23.20) >consensus UUAACCUGAAACUA___AAAA___CUUGCUAAGGAA____AGCUAGGAACUAGCUAUAAGCUA_CUUAAUCUAGCUAAGAACUAGCUAAAUUAUGAUCAGCUCUGCGCAGUGGGAGCAAG .........................................(((((....((((((((((....))))...))))))....))))).............(((((........)))))... (-13.48 = -14.64 + 1.16)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:32:12 2006