
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 12,194,447 – 12,194,623 |
| Length | 176 |
| Max. P | 0.998637 |

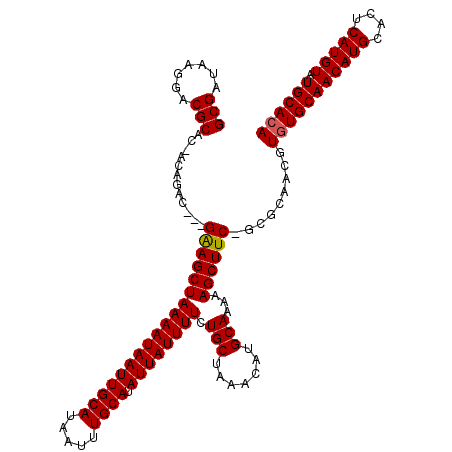
| Location | 12,194,447 – 12,194,562 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.54 |
| Mean single sequence MFE | -34.42 |
| Consensus MFE | -28.99 |
| Energy contribution | -28.93 |
| Covariance contribution | -0.05 |
| Combinations/Pair | 1.03 |
| Mean z-score | -4.00 |
| Structure conservation index | 0.84 |
| SVM decision value | 3.17 |
| SVM RNA-class probability | 0.998637 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 12194447 115 + 22224390 GCGAUAAGGACGCAC-ACAGAC---GAAGCUAAAAUAAUUGCAUAAUUUGCAUAUUAUUUUCUGCUAAACAUGCAAAAAGCUUC-GCGCAACGUGUGCAACAUGCACUCAUGUAUGCACA (((........((((-((.(.(---((((((((((((((((((.....)))).)))))))).(((.......)))...))))))-)).....)))))).(((((....))))).)))... ( -34.40) >DroSec_CAF1 20782 115 + 1 GCGAUAAGGACGCAC-ACAGAC---GAAGCUAAAAUAAUUGCAUAAUUUGCAUAUUAUUUUCUGCUAAACAUGCAAAAAGCUUC-GCGCAACGUGUGCAACAUGCACUCAUGUAUGCACA (((........((((-((.(.(---((((((((((((((((((.....)))).)))))))).(((.......)))...))))))-)).....)))))).(((((....))))).)))... ( -34.40) >DroSim_CAF1 21976 115 + 1 GCGAUAAGGACGCAC-ACAGAC---GAAGCUAAAAUAAUUGCAUAAUUUGCAUAUUAUUUUCUGCUAAACAUGCAAAAAGCUUC-GCGCAACGUGUGCAACAUGCACUCAUGUAUGCACA (((........((((-((.(.(---((((((((((((((((((.....)))).)))))))).(((.......)))...))))))-)).....)))))).(((((....))))).)))... ( -34.40) >DroEre_CAF1 20167 115 + 1 GCGAUAAGGACGCAC-ACGGAC---GGAGCUAAAAUAAUUGCAUAAUUUGCAUAUUAUUUUCUGCUAAACAUGCAAAAAGCUUC-GCGCAACGUGUGCAACAUGCACUCAUGUAUGCACA (((........((((-(((..(---((((((((((((((((((.....)))).)))))))).(((.......)))...))))))-).....))))))).(((((....))))).)))... ( -37.00) >DroYak_CAF1 5340 115 + 1 GCGAUAAGGACGCAC-ACAGAC---GAAGCUAAAAUAAUUGCAUAAUUUGCAUAUUAUUUUCUGCUAAACAUGCAAAAAGCUUC-GCGCAACGUGUGCAACAUGCACUCAUGUAUGCACA (((........((((-((.(.(---((((((((((((((((((.....)))).)))))))).(((.......)))...))))))-)).....)))))).(((((....))))).)))... ( -34.40) >DroAna_CAF1 32510 120 + 1 GCGAUAAGGGCGCUCGGCAGACACAGGAGCUAAAAUAAUUGCAUAAUUUGCAUAUUAUUUUCUGCUAAACAUGCAAAAAGCUUCCACACCGCGUUUGCAACAUGCACUCAUGUAUGCACA ((((((.(((.((...((((((.(.(((((.((((((((((((.....)))).))))))))..)))......((.....)).......))).)))))).....)).))).))).)))... ( -31.90) >consensus GCGAUAAGGACGCAC_ACAGAC___GAAGCUAAAAUAAUUGCAUAAUUUGCAUAUUAUUUUCUGCUAAACAUGCAAAAAGCUUC_GCGCAACGUGUGCAACAUGCACUCAUGUAUGCACA (((.......)))............((((((((((((((((((.....)))).)))))))).(((.......)))...)))))).........(((((((((((....))))).)))))) (-28.99 = -28.93 + -0.05)


| Location | 12,194,447 – 12,194,562 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.54 |
| Mean single sequence MFE | -36.02 |
| Consensus MFE | -28.40 |
| Energy contribution | -29.00 |
| Covariance contribution | 0.60 |
| Combinations/Pair | 1.05 |
| Mean z-score | -3.00 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.66 |
| SVM RNA-class probability | 0.813768 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 12194447 115 - 22224390 UGUGCAUACAUGAGUGCAUGUUGCACACGUUGCGC-GAAGCUUUUUGCAUGUUUAGCAGAAAAUAAUAUGCAAAUUAUGCAAUUAUUUUAGCUUC---GUCUGU-GUGCGUCCUUAUCGC .((((((......))))))...(((((((....((-((((((..((((.......))))((((((((.((((.....))))))))))))))))))---)).)))-))))........... ( -36.60) >DroSec_CAF1 20782 115 - 1 UGUGCAUACAUGAGUGCAUGUUGCACACGUUGCGC-GAAGCUUUUUGCAUGUUUAGCAGAAAAUAAUAUGCAAAUUAUGCAAUUAUUUUAGCUUC---GUCUGU-GUGCGUCCUUAUCGC .((((((......))))))...(((((((....((-((((((..((((.......))))((((((((.((((.....))))))))))))))))))---)).)))-))))........... ( -36.60) >DroSim_CAF1 21976 115 - 1 UGUGCAUACAUGAGUGCAUGUUGCACACGUUGCGC-GAAGCUUUUUGCAUGUUUAGCAGAAAAUAAUAUGCAAAUUAUGCAAUUAUUUUAGCUUC---GUCUGU-GUGCGUCCUUAUCGC .((((((......))))))...(((((((....((-((((((..((((.......))))((((((((.((((.....))))))))))))))))))---)).)))-))))........... ( -36.60) >DroEre_CAF1 20167 115 - 1 UGUGCAUACAUGAGUGCAUGUUGCACACGUUGCGC-GAAGCUUUUUGCAUGUUUAGCAGAAAAUAAUAUGCAAAUUAUGCAAUUAUUUUAGCUCC---GUCCGU-GUGCGUCCUUAUCGC .((((((......))))))...(((((((..(((.-((.(((..((((.......))))((((((((.((((.....))))))))))))))))))---)).)))-))))........... ( -34.10) >DroYak_CAF1 5340 115 - 1 UGUGCAUACAUGAGUGCAUGUUGCACACGUUGCGC-GAAGCUUUUUGCAUGUUUAGCAGAAAAUAAUAUGCAAAUUAUGCAAUUAUUUUAGCUUC---GUCUGU-GUGCGUCCUUAUCGC .((((((......))))))...(((((((....((-((((((..((((.......))))((((((((.((((.....))))))))))))))))))---)).)))-))))........... ( -36.60) >DroAna_CAF1 32510 120 - 1 UGUGCAUACAUGAGUGCAUGUUGCAAACGCGGUGUGGAAGCUUUUUGCAUGUUUAGCAGAAAAUAAUAUGCAAAUUAUGCAAUUAUUUUAGCUCCUGUGUCUGCCGAGCGCCCUUAUCGC ..((((.(((((....)))))))))...((((((.((..((.....))..(((..((((((((((((.((((.....)))))))))))..((....)).)))))..)))..)).)))))) ( -35.60) >consensus UGUGCAUACAUGAGUGCAUGUUGCACACGUUGCGC_GAAGCUUUUUGCAUGUUUAGCAGAAAAUAAUAUGCAAAUUAUGCAAUUAUUUUAGCUUC___GUCUGU_GUGCGUCCUUAUCGC ((((((.(((((....)))))))))))....(((((((((((..((((.......))))((((((((.((((.....)))))))))))))))))).........)))))........... (-28.40 = -29.00 + 0.60)

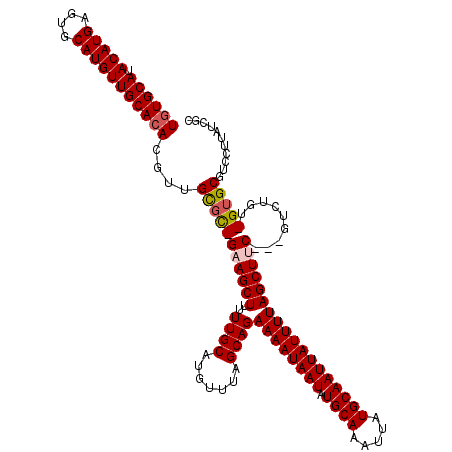
| Location | 12,194,523 – 12,194,623 |
|---|---|
| Length | 100 |
| Sequences | 4 |
| Columns | 103 |
| Reading direction | forward |
| Mean pairwise identity | 68.06 |
| Mean single sequence MFE | -24.23 |
| Consensus MFE | -14.35 |
| Energy contribution | -15.47 |
| Covariance contribution | 1.12 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.12 |
| Structure conservation index | 0.59 |
| SVM decision value | 2.68 |
| SVM RNA-class probability | 0.996281 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 12194523 100 + 22224390 CUUC-GCGCAACGUGUGCAACAUGCACUCAUGUAUGCACAUCCCCUACCCCCCCCCCCUCC--UCCACACUCUCACAGGACGAAAUGUGGGUGUGUGUAUGAG ....-.......((((((((((((....))))).)))))))................(((.--..(((((.((((((........)))))).)))))...))) ( -29.90) >DroSec_CAF1 20858 77 + 1 CUUC-GCGCAACGUGUGCAACAUGCACUCAUGUAUGCACAUCCCCC-----------------UUCCCCCUCUCCCAGGACGAGAUGUGGGUGUG-------- ...(-((((...((((((((((((....))))).))))))).....-----------------...(((.((((.......)))).).)))))))-------- ( -22.90) >DroSim_CAF1 22052 81 + 1 CUUC-GCGCAACGUGUGCAACAUGCACUCAUGUAUGCACAUCCCCC---------CU------UUCCCCCUCUCCCAGGACGAGAUAUGGGUGUGUG------ ...(-(((((..((((((((((((....))))).)))))))(((..---------.(------(((..(((.....)))..))))...)))))))))------ ( -25.50) >DroAna_CAF1 32590 95 + 1 CUUCCACACCGCGUUUGCAACAUGCACUCAUGUAUGCACAUCCUUUUUACCCACACAUGCAUACACCCACAUAUAGUGCACGAUAUAUAUGUGUG-------- ....(((((.((....))..(.((((((..((((((((...................)))))))).........)))))).)........)))))-------- ( -18.61) >consensus CUUC_GCGCAACGUGUGCAACAUGCACUCAUGUAUGCACAUCCCCC_________C_______UUCCCACUCUCACAGGACGAGAUAUGGGUGUG________ .....(((((..((((((((((((....))))).)))))))..............................((((((........)))))))))))....... (-14.35 = -15.47 + 1.12)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:30:43 2006