
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 11,979,424 – 11,979,538 |
| Length | 114 |
| Max. P | 0.831307 |

| Location | 11,979,424 – 11,979,538 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.89 |
| Mean single sequence MFE | -23.52 |
| Consensus MFE | -13.65 |
| Energy contribution | -14.23 |
| Covariance contribution | 0.58 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.17 |
| Structure conservation index | 0.58 |
| SVM decision value | 0.44 |
| SVM RNA-class probability | 0.736714 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
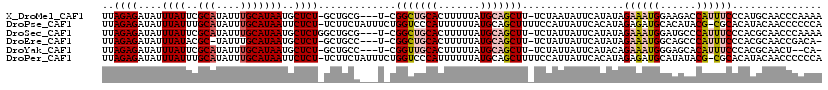
>X_DroMel_CAF1 11979424 114 + 22224390 UUAGAGAUAUUUAUUCGCAUAUUUGCAUAAUGCUCU-GCUGCG---U-CGGCUGCACUUUUUAUGCAGCUU-UCUAAUAUUCAUAUAGAAAUGGAAGACCAUUUCCCAUGCAACCCAAAA .(((((....((((..(((....)))))))..))))-).((((---(-.(((((((.......))))))).-...............(((((((....)))))))..)))))........ ( -27.40) >DroPse_CAF1 115118 118 + 1 UUAGAGAUAUUUAUUUGCAUAUUUGCAUAAUUCUCU-UCUUCUAUUUCUGGUCCCAUUUUUUAUGCAGCUUUUCCAUUAUUCACAUAGAGAUGCACAUACG-CGCACAUACAACCCCCCA ..(((((.(((....((((....)))).))))))))-............(((.....((((((((..................))))))))(((.(....)-.)))......)))..... ( -13.97) >DroSec_CAF1 19216 115 + 1 UUAGAGAUAUUUAUUCGCAUAUUUGCAUAAUGCUCUGGCUGCG---U-CGGCUGCACUUUUUAUGCAGCUU-UCUAUUAUUCAUAUAGAAAUGGAUGCCCAUUUCCCACGCAACCCAAAA ..((((....((((..(((....)))))))..))))((.((((---(-.(((((((.......))))))).-...............(((((((....)))))))..))))).))..... ( -31.40) >DroEre_CAF1 17952 112 + 1 UUAGAGAUAUUUAUACGC-UAUUUGCAUAAUGCUCU-GCUGCC---U-CGGCUGCACUUUUUAUGCAGCUU-UCUAUUAUUCAUAUAGAAAUGGCAGCCCAUUUCCCACGCAACCGACA- ..((((....((((..((-.....))))))..))))-((((((---.-.(((((((.......)))))))(-(((((.......))))))..)))))).....................- ( -29.10) >DroYak_CAF1 18876 111 + 1 UUAGAGAUAUUUAUUCGCAUAUUUGCAUAAUGCUCU-GCUGCC---U-CGGUUGCACUUUUUAUGCAGCUU-UCUAUUAUUCAUACAGAAAUGGGAGCACAUUUCCCACGCAACU--CA- ...(((.................(((((((.....(-((.((.---.-..)).)))....)))))))((((-(((...........)))))((((((.....)))))).))..))--).- ( -24.40) >DroPer_CAF1 119997 118 + 1 UUAGAGAUAUUUAUUUGCAUAUUUGCAUAAUUCUCU-UCUUCUAUUUCUGGUCCCAUUUUUUAUGCAGCUUUUCCAUUAUUCACAUAGAGAUGCAUAUACG-CGCACAUACAACCCCCCA ..(((((.(((....((((....)))).))))))))-........................((((((.((((..............)))).))))))....-.................. ( -14.84) >consensus UUAGAGAUAUUUAUUCGCAUAUUUGCAUAAUGCUCU_GCUGCC___U_CGGCUGCACUUUUUAUGCAGCUU_UCUAUUAUUCAUAUAGAAAUGGAAGCACAUUUCCCACGCAACCCACAA ..((((....((((..(((....)))))))..)))).............(((((((.......))))))).................((((((......))))))............... (-13.65 = -14.23 + 0.58)
| Location | 11,979,424 – 11,979,538 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.89 |
| Mean single sequence MFE | -29.98 |
| Consensus MFE | -15.74 |
| Energy contribution | -14.67 |
| Covariance contribution | -1.08 |
| Combinations/Pair | 1.37 |
| Mean z-score | -2.70 |
| Structure conservation index | 0.53 |
| SVM decision value | 0.72 |
| SVM RNA-class probability | 0.831307 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
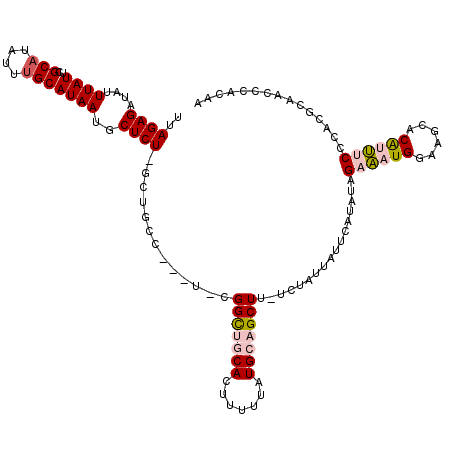
>X_DroMel_CAF1 11979424 114 - 22224390 UUUUGGGUUGCAUGGGAAAUGGUCUUCCAUUUCUAUAUGAAUAUUAGA-AAGCUGCAUAAAAAGUGCAGCCG-A---CGCAGC-AGAGCAUUAUGCAAAUAUGCGAAUAAAUAUCUCUAA ......(((((...((((((((....))))))))..............-..(((((((.....)))))))..-.---.)))))-((((..(((((((....)))..))))....)))).. ( -31.00) >DroPse_CAF1 115118 118 - 1 UGGGGGGUUGUAUGUGCG-CGUAUGUGCAUCUCUAUGUGAAUAAUGGAAAAGCUGCAUAAAAAAUGGGACCAGAAAUAGAAGA-AGAGAAUUAUGCAAAUAUGCAAAUAAAUAUCUCUAA ((((((.((((((((..(-((((((((((.((((((.......)))))...).)))))).....((....))...........-.......)))))..)))))))).......)))))). ( -23.80) >DroSec_CAF1 19216 115 - 1 UUUUGGGUUGCGUGGGAAAUGGGCAUCCAUUUCUAUAUGAAUAAUAGA-AAGCUGCAUAAAAAGUGCAGCCG-A---CGCAGCCAGAGCAUUAUGCAAAUAUGCGAAUAAAUAUCUCUAA .....((((((((.((((((((....)))))))...............-..(((((((.....)))))))).-)---)))))))((((..(((((((....)))..))))....)))).. ( -37.70) >DroEre_CAF1 17952 112 - 1 -UGUCGGUUGCGUGGGAAAUGGGCUGCCAUUUCUAUAUGAAUAAUAGA-AAGCUGCAUAAAAAGUGCAGCCG-A---GGCAGC-AGAGCAUUAUGCAAAUA-GCGUAUAAAUAUCUCUAA -.((...((((((((.......((((((.(((((((.......)))))-))(((((((.....)))))))..-.---))))))-......))))))))...-))................ ( -36.72) >DroYak_CAF1 18876 111 - 1 -UG--AGUUGCGUGGGAAAUGUGCUCCCAUUUCUGUAUGAAUAAUAGA-AAGCUGCAUAAAAAGUGCAACCG-A---GGCAGC-AGAGCAUUAUGCAAAUAUGCGAAUAAAUAUCUCUAA -..--..(((((((.(....((((((...(((((((.......)))))-))(((((................-.---.)))))-.))))))....)...))))))).............. ( -26.87) >DroPer_CAF1 119997 118 - 1 UGGGGGGUUGUAUGUGCG-CGUAUAUGCAUCUCUAUGUGAAUAAUGGAAAAGCUGCAUAAAAAAUGGGACCAGAAAUAGAAGA-AGAGAAUUAUGCAAAUAUGCAAAUAAAUAUCUCUAA ((((((.((((((((..(-((((((((((.((((((.......)))))...).)))))).....((....))...........-.......)))))..)))))))).......)))))). ( -23.80) >consensus UUGUGGGUUGCAUGGGAAAUGGGCUUCCAUUUCUAUAUGAAUAAUAGA_AAGCUGCAUAAAAAGUGCAACCG_A___AGCAGC_AGAGCAUUAUGCAAAUAUGCGAAUAAAUAUCUCUAA .....((((((((((((.......)))))).(((((.......))))).................)))))).............((((..((((((......)))..)))....)))).. (-15.74 = -14.67 + -1.08)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:28:00 2006