
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 11,773,617 – 11,773,803 |
| Length | 186 |
| Max. P | 0.929685 |

| Location | 11,773,617 – 11,773,731 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 89.27 |
| Mean single sequence MFE | -34.78 |
| Consensus MFE | -29.77 |
| Energy contribution | -29.63 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.86 |
| SVM decision value | 0.48 |
| SVM RNA-class probability | 0.753248 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11773617 114 - 22224390 CAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGUCAUCC------UAGGCAUGGCAACAGUCAUCCGCGGUAUCCAAGAGGUAUUUAUUGGCUC ...((((((((((.((((...(((((.((((......)))).)))))...)))))))).......------..((.(((((....)))))))..))))))...(((.((.....)).))) ( -34.90) >DroVir_CAF1 4137 110 - 1 CAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGACAUCC------UAGGCAUAGCAAUAGUCAUCAACGGUAUCCAAGAGGUAUUUAUCC---- ...((((...(.((((..((((((((..(((.((((((((.((((.((.((....)).)))))))------)....)))))).)))......))))))))..)))).)...)))).---- ( -31.80) >DroGri_CAF1 3757 110 - 1 CAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGCCAUCC------UAGGCAUGGCAACAGUCAUCCACGGUAUCCAAGAGGUAUUUAUCC---- ...((((...(.((((..((((((((...........(((.((((((..((....)).)))))))------))((.(((((....)))))))))))))))..)))).)...)))).---- ( -38.40) >DroWil_CAF1 7620 120 - 1 CAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGCCAUCCAGCAUCCAGGCAUGGCAACAGCCAUCCGCGUUAUCCAAGAGGUAUUUAUUAGUUU ..(((((...(.((((..((((.(((.....((((((....((((((..((....)).))))))))))))...((.(((((....)))))))))).))))..)))).)...))))).... ( -35.80) >DroMoj_CAF1 4176 113 - 1 CAUGAUAUCGGAUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGGCAUCC------UAGGCAUGGCAACAAUCAUCCACGGUAUCCAAGAGGUAUUUAACAGUU- ...((((((((.....))((((((((.....(((((.(((.((((.((.((....)).)))))))------)))))))(....)........)))))))).....))))))........- ( -31.40) >DroAna_CAF1 3255 114 - 1 CAUGAUAUAGGGUUCUCCGAUACCGUAUCUAAUGUCGUAGAGAUGGUCAAGGAGACUCGUCAUCC------UAGGCAUGGCAACAGCCAUCCACGGUAUCCCAGAGGUAGUUGUUGGAUG ...((((...(.((((..((((((((.....(((((.(((.(((((.(.((....)).)))))))------)))))))(((....)))....))))))))..)))).)...))))..... ( -36.40) >consensus CAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGCCAUCC______UAGGCAUGGCAACAGUCAUCCACGGUAUCCAAGAGGUAUUUAUCAG_U_ ...(((((((((.(((((...(((((.((((......)))).)))))...))))).))...............(((.((....))))).....))))))).................... (-29.77 = -29.63 + -0.14)



| Location | 11,773,657 – 11,773,771 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.41 |
| Mean single sequence MFE | -40.15 |
| Consensus MFE | -33.57 |
| Energy contribution | -32.85 |
| Covariance contribution | -0.72 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.50 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.20 |
| SVM RNA-class probability | 0.929685 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11773657 114 + 22224390 CCAUGCCUA------GGAUGACGAGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCGUCGAGGACUCGGAGAU .(((.(...------.)))).((((..((((((((.(((..((((((((((.....((((((((.....)))))))).)))))).))))..)))(((...)))))))))))))))..... ( -39.40) >DroVir_CAF1 4173 114 + 1 CUAUGCCUA------GGAUGUCGAGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCGCGUCGUCUAGGACUUGGCGAU (((..((((------((((((((((....)))).(((((..((((((((((.....((((((((.....)))))))).)))))).))))..)))))..)))))..)))))...))).... ( -37.70) >DroGri_CAF1 3793 114 + 1 CCAUGCCUA------GGAUGGCGAGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCGCGCCGUCUUGGACUUGGCGAU (((.(.(((------((((((((.(((.....)))((((..((((((((((.....((((((((.....)))))))).)))))).))))..))))....))))))))))).).))).... ( -45.50) >DroWil_CAF1 7660 120 + 1 CCAUGCCUGGAUGCUGGAUGGCGAGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGUCGUCGCGGACUCGGAGAU ((((.((........))))))((((((....((.(((((..((((((((((.....((((((((.....)))))))).)))))).))))..))))).))(((....)))))))))..... ( -42.00) >DroMoj_CAF1 4215 114 + 1 CCAUGCCUA------GGAUGCCGAGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAAUCCGAUAUCAUGAUGUAUGUAUUGAUGGCCGCGUCGUCUUGGACUUGGCGAU ...((((.(------(....(((((.(..(.((.(((((..((((((((((.....((((((((.....)))))))).)))))).))))..))))).)).)..).))))).)).)))).. ( -41.80) >DroAna_CAF1 3295 114 + 1 CCAUGCCUA------GGAUGACGAGUCUCCUUGACCAUCUCUACGACAUUAGAUACGGUAUCGGAGAACCCUAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCUUCGAGGGCUUGGCGAU ...((((.(------(((.((....))))))((.(((((..((((((((((((((.(((........)))...)))).)))))).))))..))))).)).((((....))))..)))).. ( -34.50) >consensus CCAUGCCUA______GGAUGACGAGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCGUCGAGGACUUGGCGAU .(((.((........))))).(((((((.(.((.(((((..((((((((((.....((((((((.....)))))))).)))))).))))..))))).)).).......)))))))..... (-33.57 = -32.85 + -0.72)


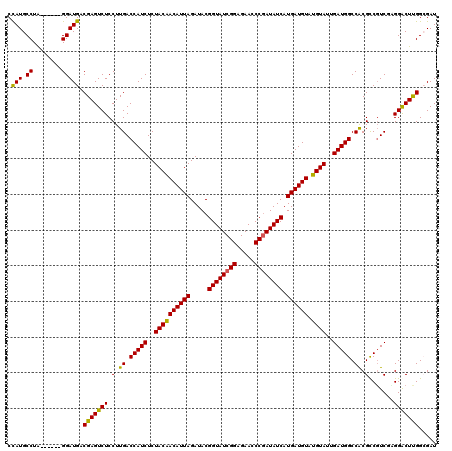
| Location | 11,773,657 – 11,773,771 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.41 |
| Mean single sequence MFE | -37.78 |
| Consensus MFE | -30.62 |
| Energy contribution | -30.90 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.25 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.28 |
| SVM RNA-class probability | 0.665292 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11773657 114 - 22224390 AUCUCCGAGUCCUCGACGGCGUGGCCAUCAAUACAUACAUCAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGUCAUCC------UAGGCAUGG .(((((..(((......))).((((((((..((((.((((...((((.(((..((...))..)))))))..))))))))..)))))))).)))))...(((....------..))).... ( -36.60) >DroVir_CAF1 4173 114 - 1 AUCGCCAAGUCCUAGACGACGCGGCCAUCAAUACAUACAUCAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGACAUCC------UAGGCAUAG ...(((..(((......)))..(((((((..((((.((((...((((.(((..((...))..)))))))..))))))))..))))))).((((.........)))------).))).... ( -35.30) >DroGri_CAF1 3793 114 - 1 AUCGCCAAGUCCAAGACGGCGCGGCCAUCAAUACAUACAUCAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGCCAUCC------UAGGCAUGG ...(((..(((...)))((((.(((((((..((((.((((...((((.(((..((...))..)))))))..))))))))..))))))).((....))))))....------..))).... ( -40.30) >DroWil_CAF1 7660 120 - 1 AUCUCCGAGUCCGCGACGACGUGGCCAUCAAUACAUACAUCAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGCCAUCCAGCAUCCAGGCAUGG .(((((..(((......))).((((((((..((((.((((...((((.(((..((...))..)))))))..))))))))..)))))))).)))))....((((((........)).)))) ( -39.60) >DroMoj_CAF1 4215 114 - 1 AUCGCCAAGUCCAAGACGACGCGGCCAUCAAUACAUACAUCAUGAUAUCGGAUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGGCAUCC------UAGGCAUGG ...(((..(((......)))(((((((((..((((.((((.(((.((((((.....)))))).))).....))))))))..))))))).((....))..))....------..))).... ( -37.80) >DroAna_CAF1 3295 114 - 1 AUCGCCAAGCCCUCGAAGGCGUGGCCAUCAAUACAUACAUCAUGAUAUAGGGUUCUCCGAUACCGUAUCUAAUGUCGUAGAGAUGGUCAAGGAGACUCGUCAUCC------UAGGCAUGG ...((((.(((......))).))))...............((((...(((((((((((...(((((.((((......)))).)))))...)))))......))))------))..)))). ( -37.10) >consensus AUCGCCAAGUCCUAGACGACGCGGCCAUCAAUACAUACAUCAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGCCAUCC______UAGGCAUGG ...(((..(((......)))..(((((((..((((.((((.(((.((((((.....)))))).))).....))))))))..))))))).((....))................))).... (-30.62 = -30.90 + 0.28)


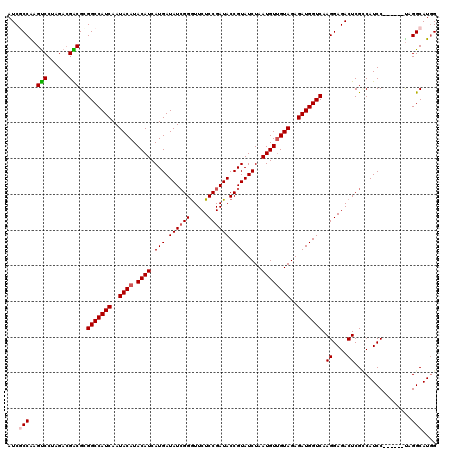
| Location | 11,773,691 – 11,773,803 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.20 |
| Mean single sequence MFE | -38.83 |
| Consensus MFE | -34.31 |
| Energy contribution | -34.73 |
| Covariance contribution | 0.42 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.08 |
| Structure conservation index | 0.88 |
| SVM decision value | 1.10 |
| SVM RNA-class probability | 0.914967 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11773691 112 + 22224390 CUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCGUCGAGGACUCGGAGAUCUGGCCAGUGGAGAGCCAUUGUGCCUGCACGA-------- .((((((((((.....((((((((.....)))))))).)))))).))))((((((((...))))))))....(((..(....(((((((((....)))))).)))..).)))-------- ( -39.60) >DroVir_CAF1 4207 119 + 1 CUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCGCGUCGUCUAGGACUUGGCGAUCUGGCCAAAGGAGAACCGUUGUGUCUGUGAUGGAA-CAAG .((((((((((.....((((((((.....)))))))).)))))).))))(((.((((((.((((((........)))))).))))))........((((..(....)..))))..-))). ( -41.20) >DroGri_CAF1 3827 119 + 1 CUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCGCGCCGUCUUGGACUUGGCGAUCUGGCCAAUGGAGAACCGUUGUGUCUGUAACAGAA-CAAC .((((((((((.....((((((((.....)))))))).)))))).))))....(((((.(((((.........)))))....)))))..((....))(((((.((((...)))))-)))) ( -38.20) >DroWil_CAF1 7700 116 + 1 CUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGUCGUCGCGGACUCGGAGAUCGGGCUAAUGGCGAGCCGUUAUGCCUAGAAAAAGCA---- .((((((((((.....((((((((.....)))))))).)))))).))))..(((..((..(((......)))..))..)))((((((((((....)))))).))))..........---- ( -36.50) >DroMoj_CAF1 4249 109 + 1 CUACAACAUUAGAUACGGUAUCGGAGAAUCCGAUAUCAUGAUGUAUGUAUUGAUGGCCGCGUCGUCUUGGACUUGGCGAUCUGGCCAAAGGAGAACCGUUGUGUCUGCA----------- .........(((((((((((((((.....))))))))................((((((.((((((........)))))).))))))..((....))...)))))))..----------- ( -38.60) >DroAna_CAF1 3329 113 + 1 CUACGACAUUAGAUACGGUAUCGGAGAACCCUAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCUUCGAGGGCUUGGCGAUCUGGCGAGCGGAGAGCCGUUAUGCCUGAAAAUA------- .((((((((((((((.(((........)))...)))).)))))).))))..(((.((((.((((....)))).)))).))).(((((((((....))))).))))........------- ( -38.90) >consensus CUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCGUCGAGGACUUGGCGAUCUGGCCAAUGGAGAACCGUUGUGCCUGCAAAA________ .((((((((((.....((((((((.....)))))))).)))))).))))..(((.((((.(((......))).)))).))).(((((((((....)))))).)))............... (-34.31 = -34.73 + 0.42)

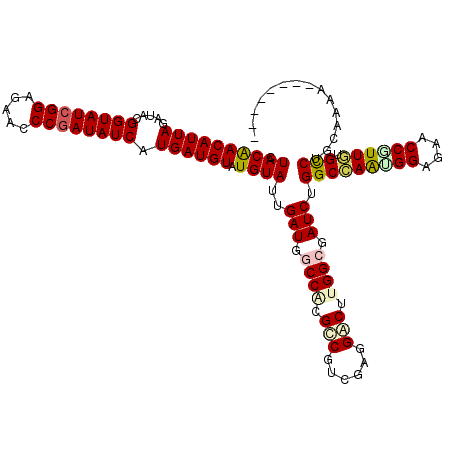
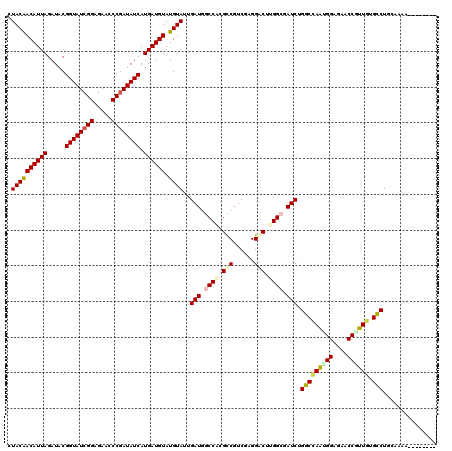
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:25:14 2006