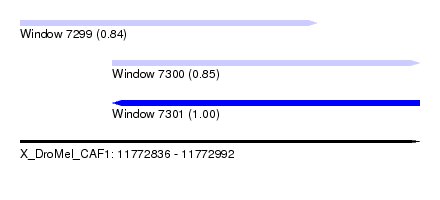
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 11,772,836 – 11,772,992 |
| Length | 156 |
| Max. P | 0.999233 |

| Location | 11,772,836 – 11,772,952 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.70 |
| Mean single sequence MFE | -30.97 |
| Consensus MFE | -28.11 |
| Energy contribution | -28.37 |
| Covariance contribution | 0.25 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.39 |
| Structure conservation index | 0.91 |
| SVM decision value | 0.76 |
| SVM RNA-class probability | 0.843329 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11772836 116 + 22224390 --UAACA--AGUGCUAAGGAUGUACCCAUAUCAAUCCCAUAACCAAGUCUGUCUCGUUCACGCAUCUCCUGAAGUCUAGCGGGACUCGGCGAUCGUGAUCUUUGACAUAGACUGGUUGUC --.....--.((((.......)))).............(((((((.((((((.(((.(((((.(((.((.((.((((....)))))))).))))))))....))).))))))))))))). ( -35.30) >DroGri_CAF1 2772 118 + 1 AACAACA--UGUGCUAAAGAUGUACCCAUAUCAAUCCCAUAACCAAGUCUGUCUCGUUCACGCAUCUCCUGAAGUCGAGCGGGACUCGGCGAUCGUGAUCUUUGACAUAAACUUGUUGUC .((((((--((((.(((((((.(((...(((.......))).....(((.(((((((((..((.((....)).)).)))))))))..)))....)))))))))).))))....)))))). ( -28.50) >DroWil_CAF1 6484 118 + 1 --UAAUUUUUGUGCUAAAGAUAUACCCAUAUCAAUCCCAUAACCAAGUCUGUCUCGUUCACGCAUCUCCUGAAGUCGGGCGGGACUCGGCGAUCGUGACCUUUGACAUAGACUUGUUGUC --......(((((.....(((((....))))).....))))).(((((((((.(((.(((((.(((.((.((.(((......))))))).))))))))....))).)))))))))..... ( -31.50) >DroMoj_CAF1 3136 118 + 1 UUUAACA--UGUGCCAAAGAUGUACCCAUAUCAAUCCCAUAACCAAGUCUGUCUCGUUCACGCAUCUCCUGAAGUCGAGCGGGACUCGGCGAUCGUGAUCUUUGACAUAAACUUGUUGUC .....((--((((((...(((((....)))))..................(((((((((..((.((....)).)).)))))))))..))))..))))......((((......))))... ( -27.40) >DroAna_CAF1 2590 118 + 1 --UAACAAAAAUGCUAAAAAUGUACCCAUAUUAAUCCCAUAACCAAGUCUGUCUCGUUCACGCAUCUCCUGAAGUCGAGCGGGACUCGGCGAUCGUGACCUUUGACAUAGACUGGUUGUC --....................................(((((((.((((((.(((.(((((.(((.((.((.(((......))))))).))))))))....))).))))))))))))). ( -30.90) >DroPer_CAF1 2882 118 + 1 --UAACAAGAGUGCUAAAGAUGUACCCAUAUCAAUCCCAUAACCAAGUCUGUCUCGUUCACGCAUCUCCUGAAGUCGAGCGGGACUCGGCGAUCGUGACCUUUGACAUAGACUUGUUGUC --..(((((.((((.......)))).)................(((((((((.(((.(((((.(((.((.((.(((......))))))).))))))))....))).))))))))))))). ( -32.20) >consensus __UAACA__AGUGCUAAAGAUGUACCCAUAUCAAUCCCAUAACCAAGUCUGUCUCGUUCACGCAUCUCCUGAAGUCGAGCGGGACUCGGCGAUCGUGACCUUUGACAUAGACUUGUUGUC ..................(((((....)))))......(((((.((((((((.(((.(((((.(((.((.((.(((......))))))).))))))))....))).))))))))))))). (-28.11 = -28.37 + 0.25)


| Location | 11,772,872 – 11,772,992 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.06 |
| Mean single sequence MFE | -41.03 |
| Consensus MFE | -37.27 |
| Energy contribution | -37.60 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.36 |
| Structure conservation index | 0.91 |
| SVM decision value | 0.78 |
| SVM RNA-class probability | 0.849225 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11772872 120 + 22224390 AACCAAGUCUGUCUCGUUCACGCAUCUCCUGAAGUCUAGCGGGACUCGGCGAUCGUGAUCUUUGACAUAGACUGGUUGUCCCAUAUGUGGGAUAAGGCAGUUGAGUGCGUUUACUGCGAG (((((.((((((.(((.(((((.(((.((.((.((((....)))))))).))))))))....))).)))))))))))((((((....))))))...(((((...........)))))... ( -44.00) >DroVir_CAF1 2673 120 + 1 AACCAAGUCUGUCUCGUUCACGCAUCUCCUGAAGUCGAGCGGGACUCGGCGAUCGUGAUCUUUGACAUAAACUUGUUGUCCCAUAUGUGGGAUAAGGCAGUUGAGUGCGUUUACUGCGAG ...(((((.((((....(((((.(((.((.((.(((......))))))).)))))))).....))))...)))))((((((((....)))))))).(((((...........)))))... ( -38.80) >DroGri_CAF1 2810 120 + 1 AACCAAGUCUGUCUCGUUCACGCAUCUCCUGAAGUCGAGCGGGACUCGGCGAUCGUGAUCUUUGACAUAAACUUGUUGUCCCAUAUGUGGGAUAAGGCAGUUGAGUGCGUUUACUGCGAG ...(((((.((((....(((((.(((.((.((.(((......))))))).)))))))).....))))...)))))((((((((....)))))))).(((((...........)))))... ( -38.80) >DroWil_CAF1 6522 120 + 1 AACCAAGUCUGUCUCGUUCACGCAUCUCCUGAAGUCGGGCGGGACUCGGCGAUCGUGACCUUUGACAUAGACUUGUUGUCCCAUAUGUGGGAUAAGGCAGUUGAGUGCGUUUACUGCGAG ...(((((((((.(((.(((((.(((.((.((.(((......))))))).))))))))....))).)))))))))((((((((....)))))))).(((((...........)))))... ( -43.40) >DroMoj_CAF1 3174 120 + 1 AACCAAGUCUGUCUCGUUCACGCAUCUCCUGAAGUCGAGCGGGACUCGGCGAUCGUGAUCUUUGACAUAAACUUGUUGUCCCAUAUGUGGGAUAAGGCAGUUGAGUGCGUUUACUGCGAG ...(((((.((((....(((((.(((.((.((.(((......))))))).)))))))).....))))...)))))((((((((....)))))))).(((((...........)))))... ( -38.80) >DroAna_CAF1 2628 120 + 1 AACCAAGUCUGUCUCGUUCACGCAUCUCCUGAAGUCGAGCGGGACUCGGCGAUCGUGACCUUUGACAUAGACUGGUUGUCCCAUAUGUGGGAUAAGGCAGUUGAGUGCGUUUACUGCGAG (((((.((((((.(((.(((((.(((.((.((.(((......))))))).))))))))....))).)))))))))))((((((....))))))...(((((...........)))))... ( -42.40) >consensus AACCAAGUCUGUCUCGUUCACGCAUCUCCUGAAGUCGAGCGGGACUCGGCGAUCGUGAUCUUUGACAUAAACUUGUUGUCCCAUAUGUGGGAUAAGGCAGUUGAGUGCGUUUACUGCGAG ...(((((.((((....(((((.(((.((.((.(((......))))))).)))))))).....))))...)))))((((((((....)))))))).(((((...........)))))... (-37.27 = -37.60 + 0.33)


| Location | 11,772,872 – 11,772,992 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.06 |
| Mean single sequence MFE | -37.22 |
| Consensus MFE | -36.23 |
| Energy contribution | -36.32 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.92 |
| Structure conservation index | 0.97 |
| SVM decision value | 3.45 |
| SVM RNA-class probability | 0.999233 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11772872 120 - 22224390 CUCGCAGUAAACGCACUCAACUGCCUUAUCCCACAUAUGGGACAACCAGUCUAUGUCAAAGAUCACGAUCGCCGAGUCCCGCUAGACUUCAGGAGAUGCGUGAACGAGACAGACUUGGUU ...(((((...........)))))....(((((....))))).(((((((((..(((...(.((((((((.(((((((......)))))..)).))).))))).)..))))))).))))) ( -37.90) >DroVir_CAF1 2673 120 - 1 CUCGCAGUAAACGCACUCAACUGCCUUAUCCCACAUAUGGGACAACAAGUUUAUGUCAAAGAUCACGAUCGCCGAGUCCCGCUCGACUUCAGGAGAUGCGUGAACGAGACAGACUUGGUU ...(((((...........)))))....(((((....)))))...((((((..((((...(.((((((((.(((((((......)))))..)).))).))))).)..))))))))))... ( -36.50) >DroGri_CAF1 2810 120 - 1 CUCGCAGUAAACGCACUCAACUGCCUUAUCCCACAUAUGGGACAACAAGUUUAUGUCAAAGAUCACGAUCGCCGAGUCCCGCUCGACUUCAGGAGAUGCGUGAACGAGACAGACUUGGUU ...(((((...........)))))....(((((....)))))...((((((..((((...(.((((((((.(((((((......)))))..)).))).))))).)..))))))))))... ( -36.50) >DroWil_CAF1 6522 120 - 1 CUCGCAGUAAACGCACUCAACUGCCUUAUCCCACAUAUGGGACAACAAGUCUAUGUCAAAGGUCACGAUCGCCGAGUCCCGCCCGACUUCAGGAGAUGCGUGAACGAGACAGACUUGGUU ...(((((...........)))))....(((((....)))))...(((((((..(((...(.((((((((.(((((((......)))))..)).))).))))).)..))))))))))... ( -38.10) >DroMoj_CAF1 3174 120 - 1 CUCGCAGUAAACGCACUCAACUGCCUUAUCCCACAUAUGGGACAACAAGUUUAUGUCAAAGAUCACGAUCGCCGAGUCCCGCUCGACUUCAGGAGAUGCGUGAACGAGACAGACUUGGUU ...(((((...........)))))....(((((....)))))...((((((..((((...(.((((((((.(((((((......)))))..)).))).))))).)..))))))))))... ( -36.50) >DroAna_CAF1 2628 120 - 1 CUCGCAGUAAACGCACUCAACUGCCUUAUCCCACAUAUGGGACAACCAGUCUAUGUCAAAGGUCACGAUCGCCGAGUCCCGCUCGACUUCAGGAGAUGCGUGAACGAGACAGACUUGGUU ...(((((...........)))))....(((((....))))).(((((((((..(((...(.((((((((.(((((((......)))))..)).))).))))).)..))))))).))))) ( -37.80) >consensus CUCGCAGUAAACGCACUCAACUGCCUUAUCCCACAUAUGGGACAACAAGUCUAUGUCAAAGAUCACGAUCGCCGAGUCCCGCUCGACUUCAGGAGAUGCGUGAACGAGACAGACUUGGUU ...(((((...........)))))....(((((....)))))...((((((..((((...(.((((((((.(((((((......)))))..)).))).))))).)..))))))))))... (-36.23 = -36.32 + 0.08)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:25:10 2006