| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 11,726,622 – 11,726,726 |
| Length | 104 |
| Max. P | 0.928819 |

| Location | 11,726,622 – 11,726,726 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 77.98 |
| Mean single sequence MFE | -27.93 |
| Consensus MFE | -18.53 |
| Energy contribution | -19.00 |
| Covariance contribution | 0.47 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.66 |
| SVM decision value | 1.19 |
| SVM RNA-class probability | 0.928819 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
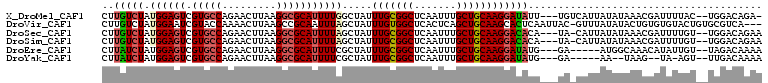
>X_DroMel_CAF1 11726622 104 - 22224390 CUUGUCUAUGGAGUCGUGCCAGAACUUAAGGCGCAUUUUGGCUAUUUGCGGCUCAAUUUGCUGCAAGGAUAUU---UGUCAUUAUAUAAACGAUUUUAC--UGGACAGA- .(((((((((((((((((((.........)))))((..((((..((((((((.......))))))))......---.))))..))......))))))).--))))))).- ( -33.50) >DroVir_CAF1 131 106 - 1 CUUGUCUAUGGAAUCGUACCAAAACUUAAGCCGCAAUUUAGCUAUUUGUGGCUCACUCAGCUGCAAGCACUCAAUUAC-GUUUAUAUACUGUGUGUACUGUGCGUCA--- ..........((..(((((.........((((((((.........))))))))(((.(((....((((..........-)))).....))).)))....))))))).--- ( -20.30) >DroSec_CAF1 8112 104 - 1 CUUGUCUAUGGAGUCGUGCCAGAACUUAAGGCGCAUUUUAGCUAUUUGCGGCUCAAUUUGCUGCAAGGACACA---UA-CAUUAUAUAAACGAUUUUGU--UGGACAGAA .(((((((..((((((((((.........)))))..........((((((((.......))))))))......---..-............)))))..)--.)))))).. ( -30.90) >DroSim_CAF1 556 104 - 1 CUUGUCUAUGGAGUCGUGCCAGAACUUAAGGCGCAUUUUAGCUAUUUGCGGCUCAAUUUGCUGCAAGGACACA---UA-CAUUAUAUAAACGAUUUUGU--UGGACAGAA .(((((((..((((((((((.........)))))..........((((((((.......))))))))......---..-............)))))..)--.)))))).. ( -30.90) >DroEre_CAF1 5670 100 - 1 CUUAUCUAUGGAGUCGUGCCAGAACUUAAGGCGCAUUUUCGCUAUUUGCGGCUCAAUUUGCUGCAAGGAUAUG---GA-----AUGGCAAACAUAUUGU--UAGACAAAA ..(((((..((((..(((((.........)))))..)))).....(((((((.......))))))))))))((---..-----.((((((.....))))--))..))... ( -26.10) >DroYak_CAF1 8528 95 - 1 CUUAUCUAUGGAGUCGUGCCAGAACUUAAGGCGCAUUUUCGCUAUUUGCGGCUCAAUUUGCUGCAAGGAUAUG---GA-----AA--UAAG--UA-AGU--UUGACAAAA ............((((((((.........)))))(((((((...((((((((.......))))))))....))---))-----))--)...--..-...--..))).... ( -25.90) >consensus CUUGUCUAUGGAGUCGUGCCAGAACUUAAGGCGCAUUUUAGCUAUUUGCGGCUCAAUUUGCUGCAAGGACACA___UA_CAUUAUAUAAACGAUUUUGU__UGGACAGAA ..(((((.((((((.(((((.........))))))))))).....(((((((.......))))))))))))....................................... (-18.53 = -19.00 + 0.47)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:24:59 2006