
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 11,653,469 – 11,653,615 |
| Length | 146 |
| Max. P | 0.999932 |

| Location | 11,653,469 – 11,653,589 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.50 |
| Mean single sequence MFE | -42.94 |
| Consensus MFE | -41.08 |
| Energy contribution | -40.60 |
| Covariance contribution | -0.48 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.54 |
| Structure conservation index | 0.96 |
| SVM decision value | 1.22 |
| SVM RNA-class probability | 0.932888 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
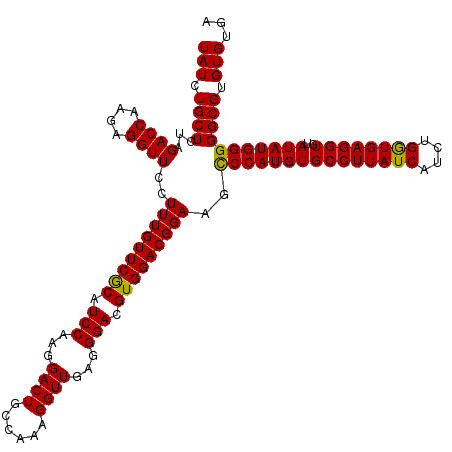
>X_DroMel_CAF1 11653469 120 + 22224390 UAUCUGCUCUAGACCAAGGGGUUCCUUUGUUCGCAUCCAAGGACUGCCAAAGGUUGAGGGACGUGGACGGAAGCCCAUGUGCCUUAUCAUCUGGUGAGGUGUAUAUGGGGGCGUGUGUGA (((.((((((.......(((.((((...((((((.(((...((((......))))...))).)))))))))).)))(((..(((((((....)))))))..)))...)))))).)))... ( -46.00) >DroSec_CAF1 26776 120 + 1 UAUCUGCUCUAGACCAAGAGGUUCCUUUGUUCGCAUCCAAGGACUGCCAAAGGUUGAGGGACGUGGACGGAAGCCCAUGUGCCUUAUCAUCUGGUGAGGUGUAUAUGGGGGCGUGUGUGA (((.((((...((((....))))..(((((((((.(((...((((......))))...))).)))))))))..(((((((((((((((....))))))))..))))))))))).)))... ( -41.00) >DroSim_CAF1 22857 120 + 1 UAUCUGCUCUAGACCAAGAGGUUCCUUUGUUCGCAUCCAAGGACUGCCAAAGGUUGAGGGACGUGGACGGAAGCCCAUGUGCCUUAUCAUCUGGUGAGGUGUAUAUGGGGGCGUGUGUGA (((.((((...((((....))))..(((((((((.(((...((((......))))...))).)))))))))..(((((((((((((((....))))))))..))))))))))).)))... ( -41.00) >DroEre_CAF1 28986 120 + 1 UAUCUGCUCUAGACCAAGGGGUUCCUUUGUUCACAUCCAAGGACUGCCAAAGGUUGAGGGACGUGGACGGAAGUCCAUGUGCCUUAUCAUCUGGUGAGGUGUAUAUGGGGGCGUGUGUGA (((.((((((.((.((((((...)))))).))(((((((.(((((.....))(.(((((.((((((((....)))))))).))))).).)))..)).))))).....)))))).)))... ( -45.10) >DroYak_CAF1 26251 120 + 1 UAUCUGCUCUAGACCAAGAGGUUCCUUUGUUCGCAUCCAAGGACUGCCAAAGGUUGAGGGACGUGGACGGAAGCCCAUGUGCCUUAUCAUCUGAUGAGGUGUAUAUGGGGGCGUGUGUGA (((.((((...((((....))))..(((((((((.(((...((((......))))...))).)))))))))..(((((((((((((((....))))))))..))))))))))).)))... ( -41.60) >consensus UAUCUGCUCUAGACCAAGAGGUUCCUUUGUUCGCAUCCAAGGACUGCCAAAGGUUGAGGGACGUGGACGGAAGCCCAUGUGCCUUAUCAUCUGGUGAGGUGUAUAUGGGGGCGUGUGUGA (((.((((...((((....))))..(((((((((.(((...((((......))))...))).)))))))))..(((((((((((((((....))))))))..))))))))))).)))... (-41.08 = -40.60 + -0.48)

| Location | 11,653,509 – 11,653,615 |
|---|---|
| Length | 106 |
| Sequences | 5 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 92.86 |
| Mean single sequence MFE | -41.22 |
| Consensus MFE | -36.54 |
| Energy contribution | -36.06 |
| Covariance contribution | -0.48 |
| Combinations/Pair | 1.11 |
| Mean z-score | -4.08 |
| Structure conservation index | 0.89 |
| SVM decision value | 4.64 |
| SVM RNA-class probability | 0.999932 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
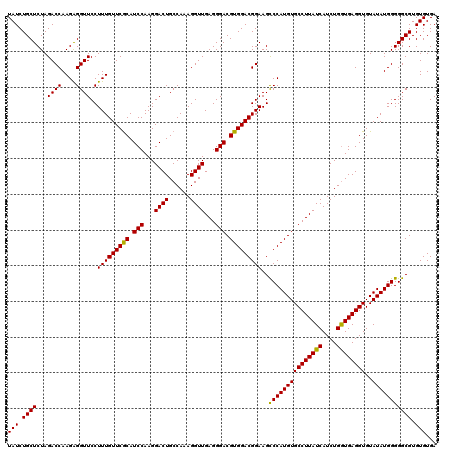
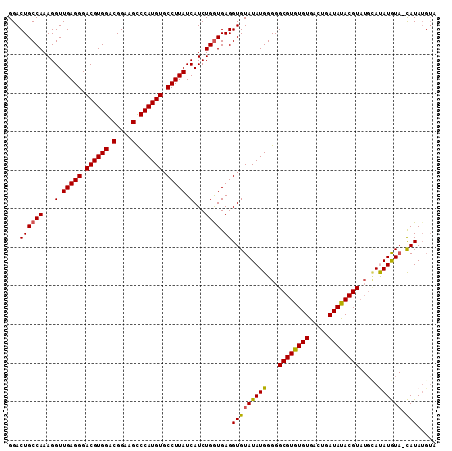
>X_DroMel_CAF1 11653509 106 + 22224390 GGACUGCCAAAGGUUGAGGGACGUGGACGGAAGCCCAUGUGCCUUAUCAUCUGGUGAGGUGUAUAUGGGGGCGUGUGUGACUGAUAUACGUAUGCAUAUGUA-CAUA---- ...((((((...(.(((((.((((((.(....).)))))).))))).)...)))).))(((((((((...((((((((.....)))))))).....))))))-))).---- ( -41.30) >DroSec_CAF1 26816 110 + 1 GGACUGCCAAAGGUUGAGGGACGUGGACGGAAGCCCAUGUGCCUUAUCAUCUGGUGAGGUGUAUAUGGGGGCGUGUGUGACUGAUAUACGUAUGCAUAUGUA-CAUAUGUA ...((((((...(.(((((.((((((.(....).)))))).))))).)...)))).))(((((((((...((((((((.....)))))))).....))))))-)))..... ( -41.30) >DroSim_CAF1 22897 110 + 1 GGACUGCCAAAGGUUGAGGGACGUGGACGGAAGCCCAUGUGCCUUAUCAUCUGGUGAGGUGUAUAUGGGGGCGUGUGUGACUGAUAUACGUAUGCAUAUGUA-CAUAUGUA ...((((((...(.(((((.((((((.(....).)))))).))))).)...)))).))(((((((((...((((((((.....)))))))).....))))))-)))..... ( -41.30) >DroEre_CAF1 29026 111 + 1 GGACUGCCAAAGGUUGAGGGACGUGGACGGAAGUCCAUGUGCCUUAUCAUCUGGUGAGGUGUAUAUGGGGGCGUGUGUGACUGAUACACGUAUGUACAUAUAGUAUAUGUA ...((((((...(.(((((.((((((((....)))))))).))))).)...)))).))((((((((....((((((((.....))))))))))))))))............ ( -45.10) >DroYak_CAF1 26291 106 + 1 GGACUGCCAAAGGUUGAGGGACGUGGACGGAAGCCCAUGUGCCUUAUCAUCUGAUGAGGUGUAUAUGGGGGCGUGUGUGAAUGAUAUACGU----ACAUAU-GUAUAUGUA ((....))..(((((((((.((((((.(....).)))))).)))))..))))....(.(((((((((...((((((((.....))))))))----...)))-)))))).). ( -37.10) >consensus GGACUGCCAAAGGUUGAGGGACGUGGACGGAAGCCCAUGUGCCUUAUCAUCUGGUGAGGUGUAUAUGGGGGCGUGUGUGACUGAUAUACGUAUGCAUAUGUA_CAUAUGUA ...((((((...(.(((((.((((((.(....).)))))).))))).)...)))).))(((((((((...((((((((.....)))))))).....)))))).)))..... (-36.54 = -36.06 + -0.48)


| Location | 11,653,509 – 11,653,615 |
|---|---|
| Length | 106 |
| Sequences | 5 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 92.86 |
| Mean single sequence MFE | -20.03 |
| Consensus MFE | -17.15 |
| Energy contribution | -16.67 |
| Covariance contribution | -0.48 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.86 |
| SVM decision value | 0.82 |
| SVM RNA-class probability | 0.857991 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
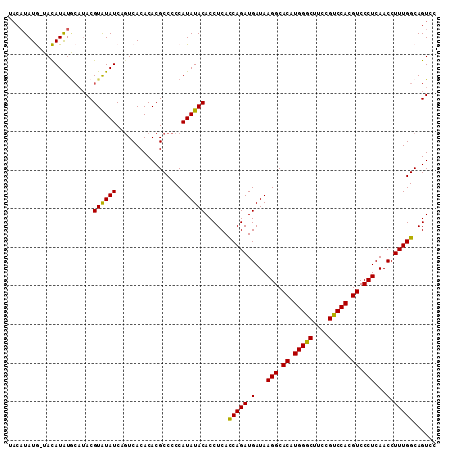
>X_DroMel_CAF1 11653509 106 - 22224390 ----UAUG-UACAUAUGCAUACGUAUAUCAGUCACACACGCCCCCAUAUACACCUCACCAGAUGAUAAGGCACAUGGGCUUCCGUCCACGUCCCUCAACCUUUGGCAGUCC ----((((-((....)))))).((((((.................))))))......(((((.(...(((.((.(((((....))))).)).)))...).)))))...... ( -19.53) >DroSec_CAF1 26816 110 - 1 UACAUAUG-UACAUAUGCAUACGUAUAUCAGUCACACACGCCCCCAUAUACACCUCACCAGAUGAUAAGGCACAUGGGCUUCCGUCCACGUCCCUCAACCUUUGGCAGUCC (((.((((-((....)))))).)))................................(((((.(...(((.((.(((((....))))).)).)))...).)))))...... ( -20.00) >DroSim_CAF1 22897 110 - 1 UACAUAUG-UACAUAUGCAUACGUAUAUCAGUCACACACGCCCCCAUAUACACCUCACCAGAUGAUAAGGCACAUGGGCUUCCGUCCACGUCCCUCAACCUUUGGCAGUCC (((.((((-((....)))))).)))................................(((((.(...(((.((.(((((....))))).)).)))...).)))))...... ( -20.00) >DroEre_CAF1 29026 111 - 1 UACAUAUACUAUAUGUACAUACGUGUAUCAGUCACACACGCCCCCAUAUACACCUCACCAGAUGAUAAGGCACAUGGACUUCCGUCCACGUCCCUCAACCUUUGGCAGUCC (((((((...)))))))....(((((.........))))).................(((((.(...(((.((.(((((....))))).)).)))...).)))))...... ( -22.20) >DroYak_CAF1 26291 106 - 1 UACAUAUAC-AUAUGU----ACGUAUAUCAUUCACACACGCCCCCAUAUACACCUCAUCAGAUGAUAAGGCACAUGGGCUUCCGUCCACGUCCCUCAACCUUUGGCAGUCC ...((((((-......----..))))))...........(((..............(((....))).(((.((.(((((....))))).)).)))........)))..... ( -18.40) >consensus UACAUAUG_UACAUAUGCAUACGUAUAUCAGUCACACACGCCCCCAUAUACACCUCACCAGAUGAUAAGGCACAUGGGCUUCCGUCCACGUCCCUCAACCUUUGGCAGUCC ......................((((((.................))))))......(((((.(...(((.((.(((((....))))).)).)))...).)))))...... (-17.15 = -16.67 + -0.48)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:24:35 2006