
| Sequence ID | X_DroMel_CAF1 |
|---|---|
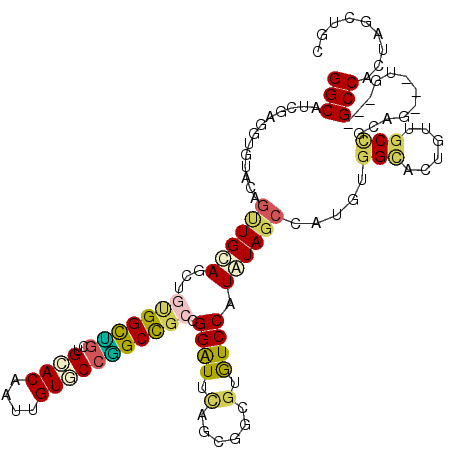
| Location | 11,564,492 – 11,564,606 |
| Length | 114 |
| Max. P | 0.857706 |

| Location | 11,564,492 – 11,564,606 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 72.57 |
| Mean single sequence MFE | -48.95 |
| Consensus MFE | -23.94 |
| Energy contribution | -23.92 |
| Covariance contribution | -0.02 |
| Combinations/Pair | 1.50 |
| Mean z-score | -1.63 |
| Structure conservation index | 0.49 |
| SVM decision value | 0.09 |
| SVM RNA-class probability | 0.577312 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11564492 114 + 22224390 GGCAUCGAGGUGUAUAGCUGCAGUUGUGGUGCCUGGACAAUGGUUCCUGCCGCCGGAUUUAGUGGAGCGUCCAUAUAACCAUGUGGCAAAGUGGCCGCAG---UG---GCCAUUGACGCA .((.((.(((..((((((....))))).)..))).))(((((((..((((.((((......((((.....))))....((....)).....)))).))))---..---)))))))..)). ( -43.80) >DroPse_CAF1 7786 117 + 1 GGCAUCGAGGUGUACAGUUGCAGCUGUGGCUGCUGCACGAUUGUGCCGGCCGCCGGAUUCAGCGGCGUGUCCAUGUAGCCAUGGGGAACUGUUGCCGCAGCCGUG---GCCGCUAGCUGC .((((....)))).(((((((.((..(((((((.((((....)))).((((((((.......)))))(.((((((....)))))).)......))))))))))..---)).)).))))). ( -54.50) >DroGri_CAF1 9759 111 + 1 GGCAUGGAGGUGUACAGUUGUAAC---GGUUGCUGCACUAUGGUGCCAGCUGCUGGAUUCAGGGGUGUGUCCAUAUAGCCGUGUGGUGCUGCUGCUGCUG---GC---GCCGUUGGUUGC ((((((..((..(((..(((...(---(((.(((((((....))).)))).))))....)))..)))..))..))).)))....((((((((....)).)---))---)))......... ( -40.80) >DroWil_CAF1 29425 106 + 1 GGCAUUGAUGUAUACAAUUGCAA---UGGCUGCUGCACAAUUGUUCCGGCCGCUGGAUUCAAUGGUGUAUCCAUAUAGCCAUGUGGCACAGAAGC---UG---UG---GCCACUGCCA-- ((((....((((......))))(---((((((.(((((.((((.(((((...)))))..)))).)))))......)))))))(((((((((...)---))---).---))))))))).-- ( -38.50) >DroAna_CAF1 5253 117 + 1 GGCAUGGAGGUGUAGAGCUGCAGCUGCGGCGGCUGUACAAUAGUUCCCGCCGCCGGGUUGAGGGGCGUGUCCAUGUAGCCGUGCGGCAAGGUGGCUGCUG---UGGCGGCCACGAAUGCC (((((((((.((((.((((((.......)))))).))))....)))).((((((((.(....(((....)))....).))).)))))...((((((((..---..))))))))..))))) ( -58.80) >DroPer_CAF1 7605 117 + 1 GGCAUCGAGGUGUACAGUUGCAGCUGUGGCUGCUGCACGAUUGUGCCGGCCGCCGGAUUCAGCGGCGUGUCCAUGUAGCCAUGGGGUACUGUUGCCGCAGCCGUG---GCCGCCAGCUGC .((((....)))).(((((((.((..(((((((.(((.....(((((.(((((........)))))....(((((....))))))))))...))).)))))))..---)).).)))))). ( -57.30) >consensus GGCAUCGAGGUGUACAGUUGCAGCUGUGGCUGCUGCACAAUUGUGCCGGCCGCCGGAUUCAGCGGCGUGUCCAUAUAGCCAUGUGGCACUGUUGCCGCAG___UG___GCCACUAGCUGC (((.............((((((...(((((((..((((....))))))))))).((((.(......).)))).)))))).....(((......)))............)))......... (-23.94 = -23.92 + -0.02)


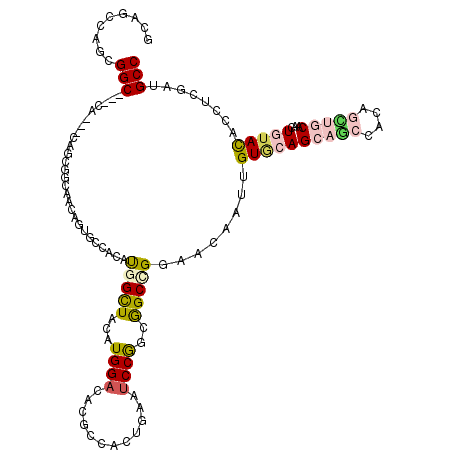
| Location | 11,564,492 – 11,564,606 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 72.57 |
| Mean single sequence MFE | -42.00 |
| Consensus MFE | -13.63 |
| Energy contribution | -14.47 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.41 |
| Mean z-score | -2.30 |
| Structure conservation index | 0.32 |
| SVM decision value | 0.81 |
| SVM RNA-class probability | 0.857706 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11564492 114 - 22224390 UGCGUCAAUGGC---CA---CUGCGGCCACUUUGCCACAUGGUUAUAUGGACGCUCCACUAAAUCCGGCGGCAGGAACCAUUGUCCAGGCACCACAACUGCAGCUAUACACCUCGAUGCC .(((((.(((((---((---.((.(((......))).)))))))))...)))))............(((.((((.......(((....)))......)))).)))............... ( -32.72) >DroPse_CAF1 7786 117 - 1 GCAGCUAGCGGC---CACGGCUGCGGCAACAGUUCCCCAUGGCUACAUGGACACGCCGCUGAAUCCGGCGGCCGGCACAAUCGUGCAGCAGCCACAGCUGCAACUGUACACCUCGAUGCC ((((((.(((((---....)))))))).((((((..(((((....)))))....(((((((....)))))))..((((....)))).(((((....))))))))))).........))). ( -52.80) >DroGri_CAF1 9759 111 - 1 GCAACCAACGGC---GC---CAGCAGCAGCAGCACCACACGGCUAUAUGGACACACCCCUGAAUCCAGCAGCUGGCACCAUAGUGCAGCAACC---GUUACAACUGUACACCUCCAUGCC (((......((.---((---((((.((...(((........)))...((((..((....))..)))))).)))))).))...((((((.....---.......)))))).......))). ( -33.10) >DroWil_CAF1 29425 106 - 1 --UGGCAGUGGC---CA---CA---GCUUCUGUGCCACAUGGCUAUAUGGAUACACCAUUGAAUCCAGCGGCCGGAACAAUUGUGCAGCAGCCA---UUGCAAUUGUAUACAUCAAUGCC --.(((((((((---.(---((---(...))))))))).(((((...(((((.((....)).)))))..)))))..((((((((((....))..---..)))))))).........)))) ( -36.50) >DroAna_CAF1 5253 117 - 1 GGCAUUCGUGGCCGCCA---CAGCAGCCACCUUGCCGCACGGCUACAUGGACACGCCCCUCAACCCGGCGGCGGGAACUAUUGUACAGCCGCCGCAGCUGCAGCUCUACACCUCCAUGCC ((((...(((((.((..---..)).)))))..))))....(((...(((((..............(((((((..(.((....)).).))))))).(((....))).......)))))))) ( -42.80) >DroPer_CAF1 7605 117 - 1 GCAGCUGGCGGC---CACGGCUGCGGCAACAGUACCCCAUGGCUACAUGGACACGCCGCUGAAUCCGGCGGCCGGCACAAUCGUGCAGCAGCCACAGCUGCAACUGUACACCUCGAUGCC ((((((((((((---....)))))(((.........(((((....)))))....(((((((....)))))))..((((....))))....))).)))))))................... ( -54.10) >consensus GCAGCCAGCGGC___CA___CAGCGGCAACAGUGCCACAUGGCUACAUGGACACGCCACUGAAUCCGGCGGCCGGAACAAUUGUGCAGCAGCCACAGCUGCAACUGUACACCUCGAUGCC .........(((...........................(((((...((((............))))..)))))........((((((((((....)))))...)))))........))) (-13.63 = -14.47 + 0.84)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:23:52 2006