
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 11,527,067 – 11,527,237 |
| Length | 170 |
| Max. P | 0.999983 |

| Location | 11,527,067 – 11,527,184 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 96.41 |
| Mean single sequence MFE | -52.82 |
| Consensus MFE | -49.64 |
| Energy contribution | -50.44 |
| Covariance contribution | 0.80 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.67 |
| Structure conservation index | 0.94 |
| SVM decision value | 4.35 |
| SVM RNA-class probability | 0.999878 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11527067 117 + 22224390 GGCCAUGUGAGCAAACACCAUCAGAUGCUGCUCGCCGAGCAGAUGCAUCUGUUGCAGCACCAGGAGCAGCUGGAGCUGGAGCAGAUGCACCAGCUGGGGCUGGCGCCGGAGCAGCAC ((...(((......)))))......(((((((..(((.((...((((((((((.((((.((((......)))).)))).))))))))))(((((....))))).)))))))))))). ( -58.20) >DroSec_CAF1 8331 117 + 1 GGCCAUGUGAGCAAACACCAUCAGAUGCUGCUCGCCGAGCAGAUGCACCUGUUGCAGCACCAGGAGCAGCUGGACCUGGAGCAGAUGCACCAGCUGGGACUGGCGCCGGAGCAGCAC ((...(((......)))))......((((((((.((.(((.(.((((.(((((.(((..((((......))))..))).))))).)))).).))).))..((....)))))))))). ( -45.30) >DroSim_CAF1 9486 117 + 1 GGCCAUGUGAGCAAACACCAUCAGAUGCUGCUCGCCGAGCAGAUGCACCUGUUGCAGCACCAGGAGCAGCUGGAGCUGGAGCAGAUGCACCAGCUGGGGCUGGCGCCGGAGCAGCAC ((...(((......)))))......(((((((..(((.((...((((.(((((.((((.((((......)))).)))).))))).))))(((((....))))).)))))))))))). ( -53.60) >DroEre_CAF1 8182 117 + 1 GGCCAUGUGAGCAAACACCACCAGAUGCUGCUCGCCGAGCAGAUGCACCUGUUGCAGCACCAGGAGCAACUGGAGCUGGAGCAGAUGCACCAGCUGGGGCUGGCACCGGAGCAGCAU ((...(((......)))...))..((((((((((((.(((...((((.(((((.((((.((((......)))).)))).))))).))))((....)).))))))....))))))))) ( -53.30) >DroYak_CAF1 8800 117 + 1 GGCCAUGUGAGCAAACACCACCAGAUGCUGCUCGCCGAGCAGAUGCACCUGUUGCAGCACCAGGAGCAACUGGAGCUGGAGCAAAUGCACCAGCUGGGGCUGGUGCCGGAGCAGCAU ((...(((......)))...))..((((((((..(((.(((..((((..((((.((((.((((......)))).)))).))))..))))(((((....))))))))))))))))))) ( -53.70) >consensus GGCCAUGUGAGCAAACACCAUCAGAUGCUGCUCGCCGAGCAGAUGCACCUGUUGCAGCACCAGGAGCAGCUGGAGCUGGAGCAGAUGCACCAGCUGGGGCUGGCGCCGGAGCAGCAC ((...(((......)))))......(((((((..(((.((...((((.(((((.((((.((((......)))).)))).))))).))))(((((....))))).)))))))))))). (-49.64 = -50.44 + 0.80)

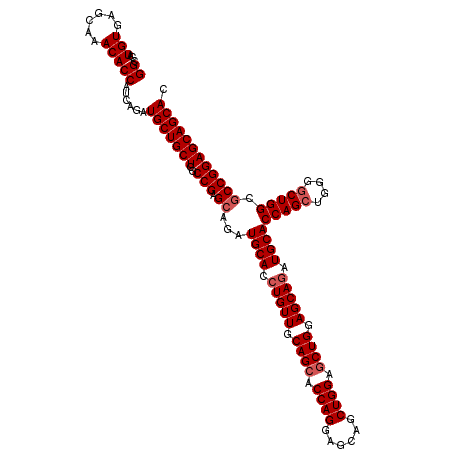
| Location | 11,527,067 – 11,527,184 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 96.41 |
| Mean single sequence MFE | -56.62 |
| Consensus MFE | -55.18 |
| Energy contribution | -54.94 |
| Covariance contribution | -0.24 |
| Combinations/Pair | 1.10 |
| Mean z-score | -3.56 |
| Structure conservation index | 0.97 |
| SVM decision value | 5.32 |
| SVM RNA-class probability | 0.999983 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11527067 117 - 22224390 GUGCUGCUCCGGCGCCAGCCCCAGCUGGUGCAUCUGCUCCAGCUCCAGCUGCUCCUGGUGCUGCAACAGAUGCAUCUGCUCGGCGAGCAGCAUCUGAUGGUGUUUGCUCACAUGGCC ..((((...))))(((((((..(((.((((((((((...((((.((((......)))).))))...)))))))))).))).)))((((((((((....)))).))))))...)))). ( -58.80) >DroSec_CAF1 8331 117 - 1 GUGCUGCUCCGGCGCCAGUCCCAGCUGGUGCAUCUGCUCCAGGUCCAGCUGCUCCUGGUGCUGCAACAGGUGCAUCUGCUCGGCGAGCAGCAUCUGAUGGUGUUUGCUCACAUGGCC ..((((...))))(((((.((.(((.((((((((((...(((..((((......))))..)))...)))))))))).))).)))((((((((((....)))).))))))...)))). ( -51.80) >DroSim_CAF1 9486 117 - 1 GUGCUGCUCCGGCGCCAGCCCCAGCUGGUGCAUCUGCUCCAGCUCCAGCUGCUCCUGGUGCUGCAACAGGUGCAUCUGCUCGGCGAGCAGCAUCUGAUGGUGUUUGCUCACAUGGCC ..((((...))))(((((((..(((.((((((((((...((((.((((......)))).))))...)))))))))).))).)))((((((((((....)))).))))))...)))). ( -58.20) >DroEre_CAF1 8182 117 - 1 AUGCUGCUCCGGUGCCAGCCCCAGCUGGUGCAUCUGCUCCAGCUCCAGUUGCUCCUGGUGCUGCAACAGGUGCAUCUGCUCGGCGAGCAGCAUCUGGUGGUGUUUGCUCACAUGGCC (((((((((.(....).(((..(((.((((((((((...((((.((((......)))).))))...)))))))))).))).))))))))))))..(((.((((......)))).))) ( -57.70) >DroYak_CAF1 8800 117 - 1 AUGCUGCUCCGGCACCAGCCCCAGCUGGUGCAUUUGCUCCAGCUCCAGUUGCUCCUGGUGCUGCAACAGGUGCAUCUGCUCGGCGAGCAGCAUCUGGUGGUGUUUGCUCACAUGGCC (((((((((.(((....)))(((((.((((((((((...((((.((((......)))).))))...)))))))))).))).)).)))))))))..(((.((((......)))).))) ( -56.60) >consensus GUGCUGCUCCGGCGCCAGCCCCAGCUGGUGCAUCUGCUCCAGCUCCAGCUGCUCCUGGUGCUGCAACAGGUGCAUCUGCUCGGCGAGCAGCAUCUGAUGGUGUUUGCUCACAUGGCC ..((((...))))(((((((..(((.((((((((((...((((.((((......)))).))))...)))))))))).))).)))((((((((((....)))).))))))...)))). (-55.18 = -54.94 + -0.24)


| Location | 11,527,107 – 11,527,224 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 96.24 |
| Mean single sequence MFE | -49.60 |
| Consensus MFE | -45.96 |
| Energy contribution | -46.40 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.31 |
| Structure conservation index | 0.93 |
| SVM decision value | 3.57 |
| SVM RNA-class probability | 0.999397 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11527107 117 + 22224390 AGAUGCAUCUGUUGCAGCACCAGGAGCAGCUGGAGCUGGAGCAGAUGCACCAGCUGGGGCUGGCGCCGGAGCAGCACCAUCAUGUGACCGUCGACGAGAUCAUCGAGCUGCCCACAU ...((((((((((.((((.((((......)))).)))).))))))))))(((((....)))))....((.(((((....((....))...((((.(....).))))))))))).... ( -53.50) >DroSec_CAF1 8371 117 + 1 AGAUGCACCUGUUGCAGCACCAGGAGCAGCUGGACCUGGAGCAGAUGCACCAGCUGGGACUGGCGCCGGAGCAGCACCAUCAUGUGACCGUCGACGAGAUCAUCGAGCUGCCCACAU ....((.((.(((.((((.((((......))))...(((.((....)).))))))).))).)).)).((.(((((....((....))...((((.(....).))))))))))).... ( -42.10) >DroSim_CAF1 9526 117 + 1 AGAUGCACCUGUUGCAGCACCAGGAGCAGCUGGAGCUGGAGCAGAUGCACCAGCUGGGGCUGGCGCCGGAGCAGCACCAUCAUGUGACCGUCGACGAGAUCAUCGAGCUGCCCACAU ...((((.(((((.((((.((((......)))).)))).))))).))))(((((....)))))....((.(((((....((....))...((((.(....).))))))))))).... ( -48.90) >DroEre_CAF1 8222 117 + 1 AGAUGCACCUGUUGCAGCACCAGGAGCAACUGGAGCUGGAGCAGAUGCACCAGCUGGGGCUGGCACCGGAGCAGCAUCAUCACGUCACCGUCGACGAGAUCAUCGAGCUGCCCACAU ...((((.(((((.((((.((((......)))).)))).))))).))))(((((....)))))....((.(((((.(((((.((((......)))).)))....))))))))).... ( -53.40) >DroYak_CAF1 8840 117 + 1 AGAUGCACCUGUUGCAGCACCAGGAGCAACUGGAGCUGGAGCAAAUGCACCAGCUGGGGCUGGUGCCGGAGCAGCAUCAUCAUGUGACCGUCGACGAGAUCAUCGAGCUGCCCACAU .........((((.((((.((((......)))).)))).))))...((((((((....)))))))).((.(((((.((((...))))...((((.(....).))))))))))).... ( -50.10) >consensus AGAUGCACCUGUUGCAGCACCAGGAGCAGCUGGAGCUGGAGCAGAUGCACCAGCUGGGGCUGGCGCCGGAGCAGCACCAUCAUGUGACCGUCGACGAGAUCAUCGAGCUGCCCACAU ...((((.(((((.((((.((((......)))).)))).))))).))))(((((....)))))....((.(((((....(((((....))).))(((.....))).))))))).... (-45.96 = -46.40 + 0.44)


| Location | 11,527,107 – 11,527,224 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 96.24 |
| Mean single sequence MFE | -53.64 |
| Consensus MFE | -50.64 |
| Energy contribution | -50.44 |
| Covariance contribution | -0.20 |
| Combinations/Pair | 1.13 |
| Mean z-score | -3.01 |
| Structure conservation index | 0.94 |
| SVM decision value | 4.83 |
| SVM RNA-class probability | 0.999954 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11527107 117 - 22224390 AUGUGGGCAGCUCGAUGAUCUCGUCGACGGUCACAUGAUGGUGCUGCUCCGGCGCCAGCCCCAGCUGGUGCAUCUGCUCCAGCUCCAGCUGCUCCUGGUGCUGCAACAGAUGCAUCU .((.((((.(.((((((....)))))))..........((((((((...))))))))))))))...((((((((((...((((.((((......)))).))))...)))))))))). ( -55.50) >DroSec_CAF1 8371 117 - 1 AUGUGGGCAGCUCGAUGAUCUCGUCGACGGUCACAUGAUGGUGCUGCUCCGGCGCCAGUCCCAGCUGGUGCAUCUGCUCCAGGUCCAGCUGCUCCUGGUGCUGCAACAGGUGCAUCU (((((((((((.(((((....)))))((.(((....))).)))))))))(((((((((...(((((((...(((((...))))))))))))...)))))))))........)))).. ( -52.00) >DroSim_CAF1 9526 117 - 1 AUGUGGGCAGCUCGAUGAUCUCGUCGACGGUCACAUGAUGGUGCUGCUCCGGCGCCAGCCCCAGCUGGUGCAUCUGCUCCAGCUCCAGCUGCUCCUGGUGCUGCAACAGGUGCAUCU .((.((((.(.((((((....)))))))..........((((((((...))))))))))))))...((((((((((...((((.((((......)))).))))...)))))))))). ( -54.90) >DroEre_CAF1 8222 117 - 1 AUGUGGGCAGCUCGAUGAUCUCGUCGACGGUGACGUGAUGAUGCUGCUCCGGUGCCAGCCCCAGCUGGUGCAUCUGCUCCAGCUCCAGUUGCUCCUGGUGCUGCAACAGGUGCAUCU .((.(((((((.(((((....)))))(((....)))......)))))))))..((((((....))))))(((((((...((((.((((......)))).))))...))))))).... ( -55.50) >DroYak_CAF1 8840 117 - 1 AUGUGGGCAGCUCGAUGAUCUCGUCGACGGUCACAUGAUGAUGCUGCUCCGGCACCAGCCCCAGCUGGUGCAUUUGCUCCAGCUCCAGUUGCUCCUGGUGCUGCAACAGGUGCAUCU ((((((.(.(.((((((....)))))))).))))))...(((((.......((((((((....))))))))(((((...((((.((((......)))).))))...)))))))))). ( -50.30) >consensus AUGUGGGCAGCUCGAUGAUCUCGUCGACGGUCACAUGAUGGUGCUGCUCCGGCGCCAGCCCCAGCUGGUGCAUCUGCUCCAGCUCCAGCUGCUCCUGGUGCUGCAACAGGUGCAUCU .((.((((.(.((((((....)))))))..........((((((((...))))))))))))))...((((((((((...((((.((((......)))).))))...)))))))))). (-50.64 = -50.44 + -0.20)

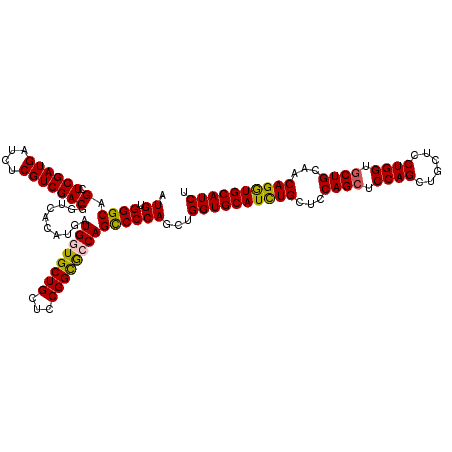

| Location | 11,527,144 – 11,527,237 |
|---|---|
| Length | 93 |
| Sequences | 5 |
| Columns | 93 |
| Reading direction | reverse |
| Mean pairwise identity | 96.77 |
| Mean single sequence MFE | -40.46 |
| Consensus MFE | -38.74 |
| Energy contribution | -38.70 |
| Covariance contribution | -0.04 |
| Combinations/Pair | 1.13 |
| Mean z-score | -1.72 |
| Structure conservation index | 0.96 |
| SVM decision value | 2.29 |
| SVM RNA-class probability | 0.991879 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11527144 93 - 22224390 UGUCCCGCCGUCGAUGUGGGCAGCUCGAUGAUCUCGUCGACGGUCACAUGAUGGUGCUGCUCCGGCGCCAGCCCCAGCUGGUGCAUCUGCUCC .....(((((((.((((((.(.(.((((((....)))))))).)))))))))))))..((....((((((((....))))))))....))... ( -42.60) >DroSec_CAF1 8408 93 - 1 UGUCCCGCCGUCGAUGUGGGCAGCUCGAUGAUCUCGUCGACGGUCACAUGAUGGUGCUGCUCCGGCGCCAGUCCCAGCUGGUGCAUCUGCUCC .....(((((((.((((((.(.(.((((((....)))))))).)))))))))))))..((....((((((((....))))))))....))... ( -40.90) >DroSim_CAF1 9563 93 - 1 UGUCCCGCCGUCGAUGUGGGCAGCUCGAUGAUCUCGUCGACGGUCACAUGAUGGUGCUGCUCCGGCGCCAGCCCCAGCUGGUGCAUCUGCUCC .....(((((((.((((((.(.(.((((((....)))))))).)))))))))))))..((....((((((((....))))))))....))... ( -42.60) >DroEre_CAF1 8259 93 - 1 UGUCCCGCCGUCGAUGUGGGCAGCUCGAUGAUCUCGUCGACGGUGACGUGAUGAUGCUGCUCCGGUGCCAGCCCCAGCUGGUGCAUCUGCUCC .........((.((((((((((((.(((((....)))))(((....)))......))))).))...((((((....))))))))))).))... ( -38.40) >DroYak_CAF1 8877 93 - 1 UGUCCCGCCGUCGAUGUGGGCAGCUCGAUGAUCUCGUCGACGGUCACAUGAUGAUGCUGCUCCGGCACCAGCCCCAGCUGGUGCAUUUGCUCC (((...((((((((((.(..((......))..).)))))))))).)))..........((....((((((((....))))))))....))... ( -37.80) >consensus UGUCCCGCCGUCGAUGUGGGCAGCUCGAUGAUCUCGUCGACGGUCACAUGAUGGUGCUGCUCCGGCGCCAGCCCCAGCUGGUGCAUCUGCUCC .....(((((((.((((((.(.(.((((((....)))))))).)))))))))))))..((....((((((((....))))))))....))... (-38.74 = -38.70 + -0.04)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:23:38 2006