
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 11,511,724 – 11,511,875 |
| Length | 151 |
| Max. P | 0.968793 |

| Location | 11,511,724 – 11,511,841 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.94 |
| Mean single sequence MFE | -44.72 |
| Consensus MFE | -34.74 |
| Energy contribution | -35.38 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.16 |
| Mean z-score | -3.92 |
| Structure conservation index | 0.78 |
| SVM decision value | 1.50 |
| SVM RNA-class probability | 0.959540 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11511724 117 + 22224390 UACAAAUGUGGAUGCACCGGAUAGAUGACAUUCG---GUUGUCAACCAGAGUGAUAACAACAAUUCGGUGGUUAUCGUCCUGGUUGACAACAAGCCCAGCUCGAUGAUCUGCAAGGAUAG ......((..(((.((.(((...((......)).---((((((((((((..(((((((.((......)).)))))))..)))))))))))).........))).)))))..))....... ( -41.10) >DroVir_CAF1 26856 115 + 1 --UG---AUCGAGGGGCCGGACAGAUGACAUUCGGCGGCGGUCAAUCAGAGCGAGAACAACAAUUCGGUUGUGAUUGUUCUGGUUGACAACAAGCCCAGCUCGAUGAUCUGCAAGGAGGG --..---((((((..((((((.........))))))(((.((((((((((((((..(((((......)))))..)))))))))))))).....)))...))))))..(((....)))... ( -47.30) >DroPse_CAF1 10513 114 + 1 UCUG---ACGGAUGGCCCGGACAGAUGACAUUCG---GUUGUCAAUCAGAGCGAUAACAACAACUCUGUGGUUAUCGUUCUGGUUGACAACAAGCCCAGUUCGAUGAUCUGCAAGGACAG (((.---.(((((.(..(((((.((......)).---(((((((((((((((((((((.(((....))).))))))))))))))))))))).......))))).).)))))..))).... ( -48.20) >DroWil_CAF1 8386 113 + 1 UCCAUA----GCCAUAGAGGACAGAUGACAUUCG---GUUGUCAAUCAGAGUGAUAACAACAAUUCGGUGGUUAUCGUUCUGGUUGACAAUAAACCCAGCUCCAUGAUAUGCAAAGAGGG ..((((----..(((.(((....((......)).---(((((((((((((((((((((.((......)).)))))))))))))))))))))........))).))).))))......... ( -35.10) >DroMoj_CAF1 25202 113 + 1 -------AUCGAUGCGCCGGACAGAUGACAUUCGGCGGUUGUCAACCAGAGCGAUAACAACAACUCGGUGGUUAUCGUGCUGGUUGACAACAAGCCCAGCUCGAUGAUCUGCAAGGAGGG -------(((((.((((((((.........)))))).((((((((((((.((((((((.((......)).)))))))).)))))))))))).......)))))))..(((....)))... ( -48.40) >DroPer_CAF1 10801 114 + 1 UCUG---ACGGAUGGCCCGGACAGAUGACAUUCG---GUUGUCAAUCAGAGCGAUAACAACAACUCUGUGGUUAUCGUUCUGGUUGACAACAAGCCCAGUUCGAUGAUCUGCAAGGACAG (((.---.(((((.(..(((((.((......)).---(((((((((((((((((((((.(((....))).))))))))))))))))))))).......))))).).)))))..))).... ( -48.20) >consensus UCUG___AUGGAUGGACCGGACAGAUGACAUUCG___GUUGUCAAUCAGAGCGAUAACAACAACUCGGUGGUUAUCGUUCUGGUUGACAACAAGCCCAGCUCGAUGAUCUGCAAGGAGAG ................((...(((((...........(((((((((((((((((((((.((......)).))))))))))))))))))))).....((......)))))))...)).... (-34.74 = -35.38 + 0.64)



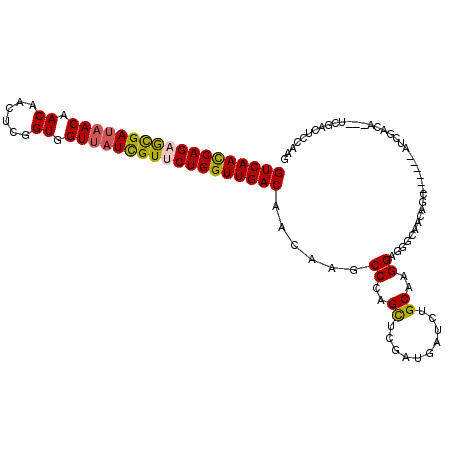
| Location | 11,511,724 – 11,511,841 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.94 |
| Mean single sequence MFE | -36.00 |
| Consensus MFE | -25.59 |
| Energy contribution | -27.15 |
| Covariance contribution | 1.56 |
| Combinations/Pair | 1.16 |
| Mean z-score | -2.39 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.57 |
| SVM RNA-class probability | 0.785675 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11511724 117 - 22224390 CUAUCCUUGCAGAUCAUCGAGCUGGGCUUGUUGUCAACCAGGACGAUAACCACCGAAUUGUUGUUAUCACUCUGGUUGACAAC---CGAAUGUCAUCUAUCCGGUGCAUCCACAUUUGUA ....(((.((.(.....)..)).)))...(((((((((((((..((((((.((......)).))))))..)))))))))))))---(((((((((((.....)))).....))))))).. ( -36.90) >DroVir_CAF1 26856 115 - 1 CCCUCCUUGCAGAUCAUCGAGCUGGGCUUGUUGUCAACCAGAACAAUCACAACCGAAUUGUUGUUCUCGCUCUGAUUGACCGCCGCCGAAUGUCAUCUGUCCGGCCCCUCGAU---CA-- ...........((((...(.(((((((..((.(((((.((((.(....(((((......)))))....).)))).))))).))....((......)).))))))).)...)))---).-- ( -29.10) >DroPse_CAF1 10513 114 - 1 CUGUCCUUGCAGAUCAUCGAACUGGGCUUGUUGUCAACCAGAACGAUAACCACAGAGUUGUUGUUAUCGCUCUGAUUGACAAC---CGAAUGUCAUCUGUCCGGGCCAUCCGU---CAGA ..((((..((((((((......))(((..((((((((.((((.(((((((.((......)).))))))).)))).))))))))---.....)))))))))..)))).......---.... ( -37.90) >DroWil_CAF1 8386 113 - 1 CCCUCUUUGCAUAUCAUGGAGCUGGGUUUAUUGUCAACCAGAACGAUAACCACCGAAUUGUUGUUAUCACUCUGAUUGACAAC---CGAAUGUCAUCUGUCCUCUAUGGC----UAUGGA .........((((((((((((...(((....((((((.((((..((((((.((......)).))))))..)))).))))))))---)((......))....)))))))).----)))).. ( -30.80) >DroMoj_CAF1 25202 113 - 1 CCCUCCUUGCAGAUCAUCGAGCUGGGCUUGUUGUCAACCAGCACGAUAACCACCGAGUUGUUGUUAUCGCUCUGGUUGACAACCGCCGAAUGUCAUCUGUCCGGCGCAUCGAU------- ...((..(((.(.....)..(((((((..((((((((((((..(((((((.((......)).)))))))..))))))))))))....((......)).))))))))))..)).------- ( -43.40) >DroPer_CAF1 10801 114 - 1 CUGUCCUUGCAGAUCAUCGAACUGGGCUUGUUGUCAACCAGAACGAUAACCACAGAGUUGUUGUUAUCGCUCUGAUUGACAAC---CGAAUGUCAUCUGUCCGGGCCAUCCGU---CAGA ..((((..((((((((......))(((..((((((((.((((.(((((((.((......)).))))))).)))).))))))))---.....)))))))))..)))).......---.... ( -37.90) >consensus CCCUCCUUGCAGAUCAUCGAGCUGGGCUUGUUGUCAACCAGAACGAUAACCACCGAAUUGUUGUUAUCGCUCUGAUUGACAAC___CGAAUGUCAUCUGUCCGGCCCAUCCAU___CAGA ..................(.(((((((..((((((((.((((.(((((((.((......)).))))))).)))).))))))))....((......)).))))))).)............. (-25.59 = -27.15 + 1.56)

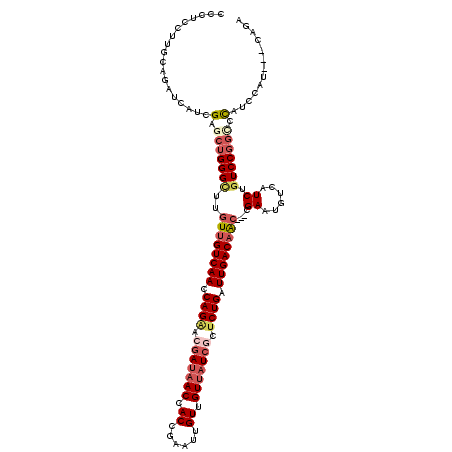

| Location | 11,511,761 – 11,511,875 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 81.11 |
| Mean single sequence MFE | -42.12 |
| Consensus MFE | -29.07 |
| Energy contribution | -28.82 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.21 |
| Mean z-score | -3.95 |
| Structure conservation index | 0.69 |
| SVM decision value | 1.63 |
| SVM RNA-class probability | 0.968793 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11511761 114 + 22224390 GUCAACCAGAGUGAUAACAACAAUUCGGUGGUUAUCGUCCUGGUUGACAACAAGCCCAGCUCGAUGAUCUGCAAGGAUAGCAAGGGCGGCAACAUCGAUGUGCUCGAAUCGCAG (((((((((..(((((((.((......)).)))))))..))))))))).....(((..((((....((((....)))).....)))))))....((((.....))))....... ( -40.70) >DroVir_CAF1 26891 105 + 1 GUCAAUCAGAGCGAGAACAACAAUUCGGUUGUGAUUGUUCUGGUUGACAACAAGCCCAGCUCGAUGAUCUGCAAGGAGGGCAGCCGU------GUCGACA---UCGAUUCGCAG ((((((((((((((..(((((......)))))..))))))))))))))..........((((((((.((.(((.((.......)).)------)).))))---))))...)).. ( -41.40) >DroGri_CAF1 8777 105 + 1 GUCAAUCAGAGUGAUAACAACAAUUCGGUGGUUAUCGUUCUGGUUGACAACAAGCCCAGCUCGAUGAUCUGCAAGGAGGGCAGCAGC------AUCGAUA---UCGACUCCAAG ((((((((((((((((((.((......)).))))))))))))))))))............((((((..((((.......))))...)------)))))..---........... ( -37.00) >DroMoj_CAF1 25235 105 + 1 GUCAACCAGAGCGAUAACAACAACUCGGUGGUUAUCGUGCUGGUUGACAACAAGCCCAGCUCGAUGAUCUGCAAGGAGGGCAGCCGC------GUGGACA---UCGACUCCAAG (((((((((.((((((((.((......)).)))))))).))))))))).........((.((((((.((..(..((.......))..------)..))))---))))))..... ( -41.90) >DroAna_CAF1 9119 107 + 1 GUCAACCAGAAUGAUAACAACAACUCGGUGGUUAUCGUUCUGGUUGACAACAAGCCCAGCUCGAUGAUCUGCAAGGAUGGGAAGAGC---CACAUCGACAUGGAG----GGCGU ((((((((((((((((((.((......)).)))))))))))))))))).....((((.((((..(.((((....)))).)...))))---..(((....)))..)----))).. ( -48.10) >DroPer_CAF1 10835 111 + 1 GUCAAUCAGAGCGAUAACAACAACUCUGUGGUUAUCGUUCUGGUUGACAACAAGCCCAGUUCGAUGAUCUGCAAGGACAGCAAGAGU---AAUCUCGACAUCAUCGAUCCCAAG ((((((((((((((((((.(((....))).))))))))))))))))))............((((((((.(((.......))).(((.---...)))...))))))))....... ( -43.60) >consensus GUCAACCAGAGCGAUAACAACAACUCGGUGGUUAUCGUUCUGGUUGACAACAAGCCCAGCUCGAUGAUCUGCAAGGAGGGCAACAGC______AUCGACA___UCGACUCCAAG ((((((((((((((((((.((......)).))))))))))))))))))......((..((..........))..))...................................... (-29.07 = -28.82 + -0.25)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:23:29 2006