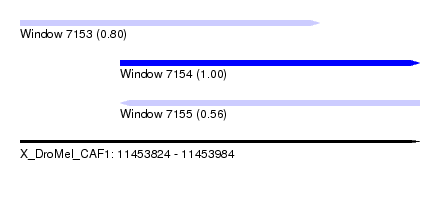
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 11,453,824 – 11,453,984 |
| Length | 160 |
| Max. P | 0.999737 |

| Location | 11,453,824 – 11,453,944 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 89.50 |
| Mean single sequence MFE | -29.18 |
| Consensus MFE | -23.04 |
| Energy contribution | -22.88 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.73 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.61 |
| SVM RNA-class probability | 0.797882 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
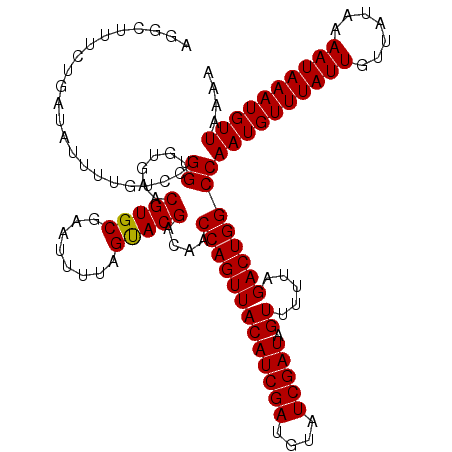
>X_DroMel_CAF1 11453824 120 + 22224390 AAUCUUUCUGAUAUUUUGUCCGGUGUGAACGUGCGAAUUUUAGUACGACAACCAGUUACAUCGAUGUAUCGAUAGUUUUUAGACUGGCCAAUGUUUAUUGUUAUAAAAUAAAUGUUACAA .(((.....))).....(.(((((.(((((((((........)))))............(((((....)))))....)))).))))).)((((((((((.......)))))))))).... ( -24.20) >DroSec_CAF1 7926 120 + 1 AGGUUUUCUGAUACUUCGUCCGGUGUGAACGUGCGAAUUUUAGUACGACAACCAGUUACAUCGAUGUAUCGAUAGUUUUUAGACUGGCCAAUGUUUAUUGUUAUAAAAUAAAUGUUAAAA .((((.(((((.((((((..((((((((.(((((........)))))........))))))))......))).)))..)))))..))))((((((((((.......)))))))))).... ( -28.20) >DroSim_CAF1 7326 120 + 1 AGGUUUUCUGAUACUUCGUCCGGUGUGAACGUGCGAAUUUUAGUACGACAACCAGUUACAUCGAUGUAUCGAUAGUUUUUAGACUGGCCAAUGUUUAUUGUUAUAAAAUAAAUGUUAAAA .((((.(((((.((((((..((((((((.(((((........)))))........))))))))......))).)))..)))))..))))((((((((((.......)))))))))).... ( -28.20) >DroEre_CAF1 11308 120 + 1 AUGCUACCAGAUAUGCUGUCCGGUGGAAACGUGCGAAUUUCAGUACGUCAGCCAGUUACAUCGAUCUAUCGAUAGUUUUUAGACUGGCCAAUGUUUAUUGUUAUAAAAUAAAUGUUAAAA ...(((((.(((.....))).)))))..((((((........))))))..((((((((((((((....))))).)).....))))))).((((((((((.......)))))))))).... ( -33.40) >DroYak_CAF1 11424 120 + 1 GUGCUUACAGAUAUGCUGUACGGUGAGGACGUGCGAGUUUUAGCACGAUAGCCAGUUACAUCGAUCUAUCGAUAGUUAAUAGACUGGCCAAUGUUUAUUGUUAUAAAAUAAAUGUUAAAA ..((((((((.....))))).))).....(((((........)))))...((((((((((((((....))))).)).....))))))).((((((((((.......)))))))))).... ( -31.90) >consensus AGGCUUUCUGAUAUUUUGUCCGGUGUGAACGUGCGAAUUUUAGUACGACAACCAGUUACAUCGAUGUAUCGAUAGUUUUUAGACUGGCCAAUGUUUAUUGUUAUAAAAUAAAUGUUAAAA .....................((......(((((........)))))....(((((((((((((....))))).)).....))))))))((((((((((.......)))))))))).... (-23.04 = -22.88 + -0.16)


| Location | 11,453,864 – 11,453,984 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.50 |
| Mean single sequence MFE | -22.94 |
| Consensus MFE | -21.19 |
| Energy contribution | -21.03 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.46 |
| Structure conservation index | 0.92 |
| SVM decision value | 3.98 |
| SVM RNA-class probability | 0.999737 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
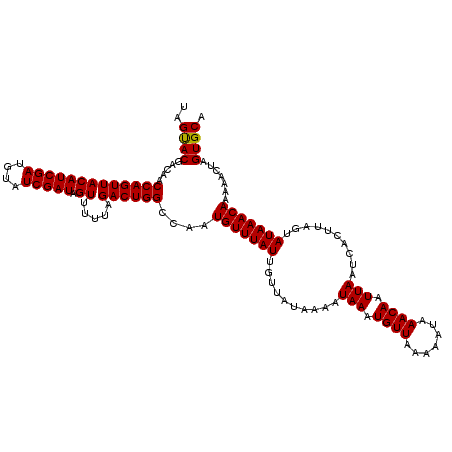
>X_DroMel_CAF1 11453864 120 + 22224390 UAGUACGACAACCAGUUACAUCGAUGUAUCGAUAGUUUUUAGACUGGCCAAUGUUUAUUGUUAUAAAAUAAAUGUUACAAAUAAACAAUUAAUCACUUAGUAUAAACAAAAAUAAGUGCA ..((((.....(((((((((((((....))))).)).....))))))....((((((((((.(((.......))).)).))))))))............................)))). ( -20.80) >DroSec_CAF1 7966 120 + 1 UAGUACGACAACCAGUUACAUCGAUGUAUCGAUAGUUUUUAGACUGGCCAAUGUUUAUUGUUAUAAAAUAAAUGUUAAAAAUAAACAAUUAAUCACUUAGUAUAAACAAAAACUAGUGCA ..((((.....(((((((((((((....))))).)).....))))))....(((((((..........(((.((((.......)))).)))..........))))))).......)))). ( -20.75) >DroSim_CAF1 7366 120 + 1 UAGUACGACAACCAGUUACAUCGAUGUAUCGAUAGUUUUUAGACUGGCCAAUGUUUAUUGUUAUAAAAUAAAUGUUAAAAAUAAACAAUUAAUCACUUAGUAUAAACAAAAACUAGUGCA ..((((.....(((((((((((((....))))).)).....))))))....(((((((..........(((.((((.......)))).)))..........))))))).......)))). ( -20.75) >DroEre_CAF1 11348 120 + 1 CAGUACGUCAGCCAGUUACAUCGAUCUAUCGAUAGUUUUUAGACUGGCCAAUGUUUAUUGUUAUAAAAUAAAUGUUAAAAAUAAACAAUUAAUCACUUAAUAUAAACAAAAACUAGUGCA ..((((....((((((((((((((....))))).)).....)))))))...(((((((.((((.....(((.((((.......)))).)))......))))))))))).......)))). ( -25.20) >DroYak_CAF1 11464 120 + 1 UAGCACGAUAGCCAGUUACAUCGAUCUAUCGAUAGUUAAUAGACUGGCCAAUGUUUAUUGUUAUAAAAUAAAUGUUAAAAAUAAACAAUUAAUCACUUAAUAUAAACAAAAACUAGUGCA ..((((....((((((((((((((....))))).)).....)))))))...(((((((.((((.....(((.((((.......)))).)))......))))))))))).......)))). ( -27.20) >consensus UAGUACGACAACCAGUUACAUCGAUGUAUCGAUAGUUUUUAGACUGGCCAAUGUUUAUUGUUAUAAAAUAAAUGUUAAAAAUAAACAAUUAAUCACUUAGUAUAAACAAAAACUAGUGCA ..((((.....(((((((((((((....))))).)).....))))))....(((((((..........(((.((((.......)))).)))..........))))))).......)))). (-21.19 = -21.03 + -0.16)

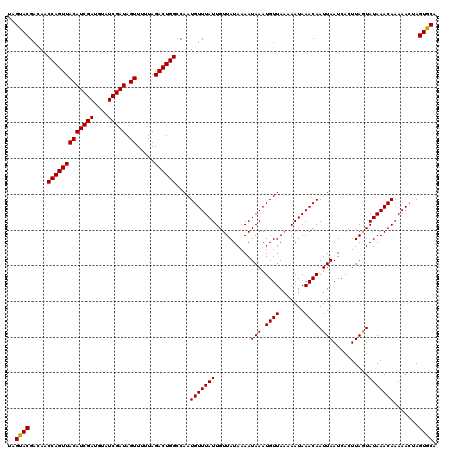
| Location | 11,453,864 – 11,453,984 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.50 |
| Mean single sequence MFE | -21.98 |
| Consensus MFE | -19.20 |
| Energy contribution | -18.64 |
| Covariance contribution | -0.56 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.24 |
| Structure conservation index | 0.87 |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.564201 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11453864 120 - 22224390 UGCACUUAUUUUUGUUUAUACUAAGUGAUUAAUUGUUUAUUUGUAACAUUUAUUUUAUAACAAUAAACAUUGGCCAGUCUAAAAACUAUCGAUACAUCGAUGUAACUGGUUGUCGUACUA ..((((((.............))))))......(((((((((((((........)))))..))))))))..(((((((.((.....((((((....)))))))))))))))......... ( -19.02) >DroSec_CAF1 7966 120 - 1 UGCACUAGUUUUUGUUUAUACUAAGUGAUUAAUUGUUUAUUUUUAACAUUUAUUUUAUAACAAUAAACAUUGGCCAGUCUAAAAACUAUCGAUACAUCGAUGUAACUGGUUGUCGUACUA (((....(((((((((.....((((((.((((..........))))))))))......))))).))))...(((((((.((.....((((((....)))))))))))))))...)))... ( -19.10) >DroSim_CAF1 7366 120 - 1 UGCACUAGUUUUUGUUUAUACUAAGUGAUUAAUUGUUUAUUUUUAACAUUUAUUUUAUAACAAUAAACAUUGGCCAGUCUAAAAACUAUCGAUACAUCGAUGUAACUGGUUGUCGUACUA (((....(((((((((.....((((((.((((..........))))))))))......))))).))))...(((((((.((.....((((((....)))))))))))))))...)))... ( -19.10) >DroEre_CAF1 11348 120 - 1 UGCACUAGUUUUUGUUUAUAUUAAGUGAUUAAUUGUUUAUUUUUAACAUUUAUUUUAUAACAAUAAACAUUGGCCAGUCUAAAAACUAUCGAUAGAUCGAUGUAACUGGCUGACGUACUG (((....(((((((((.(((.((((((.((((..........))))))))))...)))))))).)))).(..((((((.((.....((((((....))))))))))))))..).)))... ( -23.90) >DroYak_CAF1 11464 120 - 1 UGCACUAGUUUUUGUUUAUAUUAAGUGAUUAAUUGUUUAUUUUUAACAUUUAUUUUAUAACAAUAAACAUUGGCCAGUCUAUUAACUAUCGAUAGAUCGAUGUAACUGGCUAUCGUGCUA .((((..(((((((((.(((.((((((.((((..........))))))))))...)))))))).))))..((((((((.(((.....(((((....))))))))))))))))..)))).. ( -28.80) >consensus UGCACUAGUUUUUGUUUAUACUAAGUGAUUAAUUGUUUAUUUUUAACAUUUAUUUUAUAACAAUAAACAUUGGCCAGUCUAAAAACUAUCGAUACAUCGAUGUAACUGGUUGUCGUACUA (((.........(((((((.....((((.(((.((((.......)))).)))..))))....))))))).((((((((.((.....((((((....))))))))))))))))..)))... (-19.20 = -18.64 + -0.56)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:22:57 2006