
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 11,281,156 – 11,281,316 |
| Length | 160 |
| Max. P | 0.762795 |

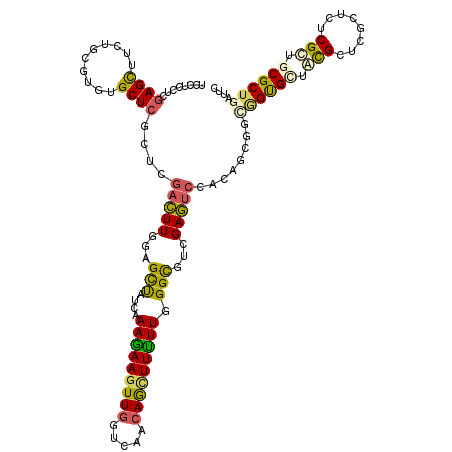
| Location | 11,281,156 – 11,281,276 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.04 |
| Mean single sequence MFE | -42.45 |
| Consensus MFE | -23.93 |
| Energy contribution | -23.08 |
| Covariance contribution | -0.85 |
| Combinations/Pair | 1.50 |
| Mean z-score | -1.86 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.51 |
| SVM RNA-class probability | 0.762795 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11281156 120 + 22224390 UCCUCCUCGAGCUUCUGCGUGUGCUCGCUACUCUUGGAGCUGUCAAAGAAGUUGGUCAACAGCUUUUUGGGCGUAGAGUCCACAGCGUUGGUGGUGCGUUCGCUUUCGCUGCGCUGAUUC ..((((..(((.....(((......)))..)))..))))..(((.(((((((((.....))))))))).)))...((((((.(((((..(((((.....)))))..))))).)..))))) ( -41.10) >DroVir_CAF1 12987 114 + 1 UCCUCCUCGAGUUUCUGUGUGUGCUCCGUCGAUUUUGAGUUAUCAAAGAAAUUUGUUAACAACUUUUUGGGUGUGGAAUCAACGGAGGCAGCGCUAUGC------UCGUUGCGCUGAUUU .(((((..((((..(.....).)))).((.((((((..((((.((((....)))).)))).(((.....)))..)))))).)))))))((((((.((..------..)).)))))).... ( -33.20) >DroPse_CAF1 12358 120 + 1 UCCUCCUCGAGCUUCUGCGUUAGCUCGCUGGACUUGGAGCCCUCAAAGAAGUUGGUCAACAGCUUCUUGGGCGUCGAGUCCAGCGCUUGGGUGUUCCGCUCGCUCUCGCUGCGCUGAUUC ......((.(((....(((..(((.(((((((((((..((((...(((((((((.....)))))))))))))..)))))))))))...(((((...))))))))..)))...)))))... ( -56.50) >DroWil_CAF1 13556 120 + 1 UCCUCCUCCAGUUUCUGGGUAUGCUCAUUCGAUUUCGAGCUAUCAAAGAAGUUGGUCAACAGUUUUUUGGGCGUCGAAUCAACAGCCGCUGUGCUACGUUCGUUCUCGUUACGCUGAUUC .......(((..((((.((((.((((..........))))))))..))))..)))....((((.(..((((...(((((..((((...)))).....)))))..))))..).)))).... ( -27.50) >DroMoj_CAF1 11666 114 + 1 UCCUCCUCGAGCUUCUGUGUGUGCUCCGUCGACUUGGAGUUAUCGAAAAAGUUGGUCAACAGCUUUUUGGGUGUGGAGUCCACGGAGGCGGCGCUAUGC------UCGUUGCGCUGAUUC .(((((..(..((((.......((((((......))))))((((.(((((((((.....))))))))).)))).))))..)..)))))((((((.((..------..)).)))))).... ( -43.40) >DroPer_CAF1 12401 120 + 1 UCCUCCUCGAGCUUCUGCGUUAGCUCGCUGGACUUGGAGCCCUCAAAGAAGUUGGUCAACAGCUUCUUGGGCGUCGAGUCCAGAGCUUGGGUGUUCCGCUCGCUCUCGCUGCGCUGAUUC .......((((((........))))))(((((((((..((((...(((((((((.....)))))))))))))..)))))))))(((..(((((.......)))))..))).......... ( -53.00) >consensus UCCUCCUCGAGCUUCUGCGUGUGCUCGCUCGACUUGGAGCUAUCAAAGAAGUUGGUCAACAGCUUUUUGGGCGUCGAGUCCACAGCGGCGGUGCUACGCUCGCUCUCGCUGCGCUGAUUC ........((((..........))))....(((((...(((....(((((((((.....))))))))).)))...)))))........((((((.(((........))).)))))).... (-23.93 = -23.08 + -0.85)


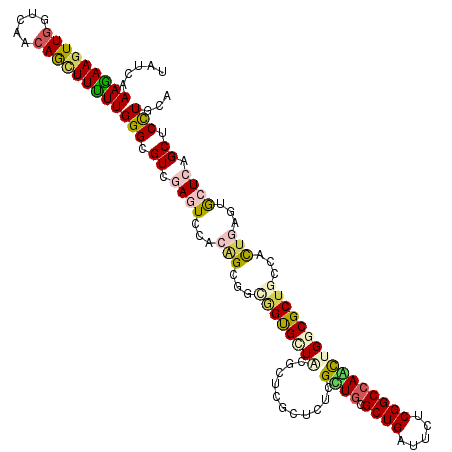
| Location | 11,281,196 – 11,281,316 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.49 |
| Mean single sequence MFE | -45.13 |
| Consensus MFE | -26.64 |
| Energy contribution | -27.20 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.43 |
| Mean z-score | -1.74 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.25 |
| SVM RNA-class probability | 0.652074 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11281196 120 + 22224390 UGUCAAAGAAGUUGGUCAACAGCUUUUUGGGCGUAGAGUCCACAGCGUUGGUGGUGCGUUCGCUUUCGCUGCGCUGAUUCUCGGCCAAUUGACGCUGCCACUGGCUGCUCAGCUCCUGCA .(((((.(((((((.....)))))))((((.((.(((((((.(((((..(((((.....)))))..))))).)..)))))))).)))))))))((.(((...))).))...((....)). ( -47.60) >DroVir_CAF1 13027 114 + 1 UAUCAAAGAAAUUUGUUAACAACUUUUUGGGUGUGGAAUCAACGGAGGCAGCGCUAUGC------UCGUUGCGCUGAUUUUCGGCCAAUUGGCGCUGCCACUGGGUGCUCAGCUCCUGCA ...((((....)))).............(((.((.((..((.(((.((((((((((...------..((((.((((.....)))))))))))))))))).)))..)).)).)).)))... ( -38.50) >DroPse_CAF1 12398 120 + 1 CCUCAAAGAAGUUGGUCAACAGCUUCUUGGGCGUCGAGUCCAGCGCUUGGGUGUUCCGCUCGCUCUCGCUGCGCUGAUUCUCGGCCAGCUGGCGCUGCCACUGAGAGCUGAGCUCCUGAA ..((((((((((((.....)))))))))((((.((((((..(((((....)))))..))))((((((((((.((((.....))))))))((((...))))..)))))).)))))).))). ( -52.80) >DroWil_CAF1 13596 120 + 1 UAUCAAAGAAGUUGGUCAACAGUUUUUUGGGCGUCGAAUCAACAGCCGCUGUGCUACGUUCGUUCUCGUUACGCUGAUUCUCGGCCAACUGCCGCUGCCAUUGAGUACUCAGCUCUUGCA ...(((((((.(((.....))))))))))(((...((((((((((...))))((.(((........)))...)))))))).((((.....))))..)))...((((.....))))..... ( -28.40) >DroMoj_CAF1 11706 114 + 1 UAUCGAAAAAGUUGGUCAACAGCUUUUUGGGUGUGGAGUCCACGGAGGCGGCGCUAUGC------UCGUUGCGCUGAUUCUCGGCCAGCUGGCGCUGCCAUUGGCUGCUCAGCUCCUGCA .(((.(((((((((.....))))))))).)))((((...))))(((((((((((((...------..((((.((((.....))))))))))))))))))....(((....)))))).... ( -48.90) >DroPer_CAF1 12441 120 + 1 CCUCAAAGAAGUUGGUCAACAGCUUCUUGGGCGUCGAGUCCAGAGCUUGGGUGUUCCGCUCGCUCUCGCUGCGCUGAUUCUCGGCCAGCUGGCGCUGCCACUGAGAGCUGAGCUCCUGAA ..((((((((((((.....)))))))))((((.....)))).(((((((((((...)))))((((((((((.((((.....))))))))((((...))))..)))))).)))))).))). ( -54.60) >consensus UAUCAAAGAAGUUGGUCAACAGCUUUUUGGGCGUCGAGUCCACAGCGGCGGUGCUACGCUCGCUCUCGCUGCGCUGAUUCUCGGCCAACUGGCGCUGCCACUGAGUGCUCAGCUCCUGCA .....(((((((((.....)))))))))(((.((.((((...(((...((((((((...........((((.((((.....))))))))))))))))...)))...)))).)).)))... (-26.64 = -27.20 + 0.56)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:21:14 2006