
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 1,247,575 – 1,247,713 |
| Length | 138 |
| Max. P | 0.819380 |

| Location | 1,247,575 – 1,247,678 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 77.55 |
| Mean single sequence MFE | -43.97 |
| Consensus MFE | -23.67 |
| Energy contribution | -25.08 |
| Covariance contribution | 1.42 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.54 |
| SVM decision value | 0.64 |
| SVM RNA-class probability | 0.807220 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
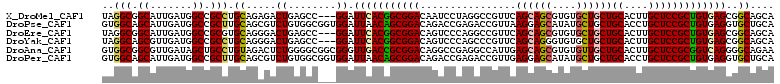
>X_DroMel_CAF1 1247575 103 + 22224390 CA--GCAAUGGCGGCUCCAGUGCCAGUGGAGUGCUGCCGCUCACAGCGGAGCAAGUGCAGCAGCACACGCUGCUGAACGGCCUAGGAUUGUCCGCCGUGAAUCC---G ..--...(((((((((((((.(((.......((((.((((.....)))))))).((.(((((((....))))))).))))))).)))..).)))))))......---. ( -47.70) >DroPse_CAF1 9183 108 + 1 AAUGGGAACAGCAGCUCCAACAACGCCGGAGUGCAGCACCUCACAGCGGAGCAGGUGCAGCAGCAUAUGCUCCUUAACGGUCUCGGUCUGUCCGCUGUUAAUCCACCG ..((((((((((.(((((.........)))))((((.(((..((...((((((.((((....)))).))))))......))...)))))))..))))))..))))... ( -38.80) >DroEre_CAF1 3603 103 + 1 CA--GCAAUGGCGGCCCCAAUGCCAGUGGAGUGCUGCCGCUCACAGCGGAGCAAGUGCAGCAGCACACGCUGCUGAACGGCCUGGGACUGUCCGCCGUGAAUCC---G ..--...((((((((((((..(((.......((((.((((.....)))))))).((.(((((((....))))))).))))).))))...).)))))))......---. ( -47.90) >DroYak_CAF1 3162 103 + 1 CA--GCAAUGGCGGCCCCAAUGCCAGUGGAGUGCUGCCGCUCACAGCGGAGCAAGUGCAGCAGCACACCCUGCUGAACGGGCUGGGACUGUCCGCCGUGAAUCC---G ..--...((((((((((((..(((.......((((.((((.....)))))))).((.((((((......)))))).)).)))))))...).)))))))......---. ( -43.30) >DroAna_CAF1 1003 100 + 1 CA--AC------GGGGUCAACACCAAUGGAGUUCUGCCCCUGACCGCGGAGCAAGUGCAGCAACACACGCUGCUCAAUGGCCUCGGCCUGUCCGCGGUCAACCCGCCG ..--..------(((((.(((.((...)).)))..)))))((((((((((.((.(.(((((.......))))).)...(((....)))))))))))))))........ ( -45.10) >DroPer_CAF1 9518 108 + 1 AAUGGGAACAGCAGCUCCAACAGCGCCGGAGUGCAGCACCUCACAGCGGAGCAGGUGCAGCAGCAUAUGCUCCUCAACGGUCUCGGUCUGUCCGCUGUUAAUCCACCG ....(((..((((((....((((.((((((.((..((........))((((((.((((....)))).))))))....)).)).))))))))..))))))..))).... ( -41.00) >consensus CA__GCAAUGGCGGCUCCAACACCAGUGGAGUGCUGCCCCUCACAGCGGAGCAAGUGCAGCAGCACACGCUGCUGAACGGCCUCGGACUGUCCGCCGUGAAUCC___G ..........(((((.(((.......)))...)))))...((((.(((((((....))((((((....))))))................))))).))))........ (-23.67 = -25.08 + 1.42)

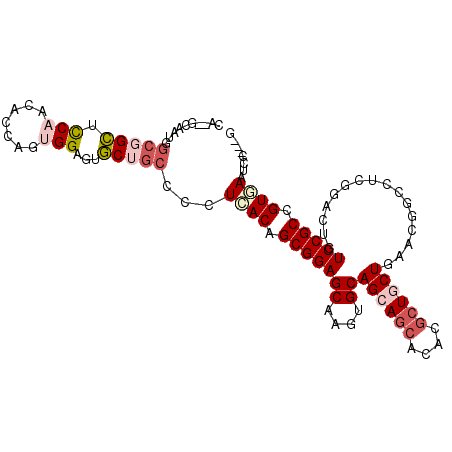
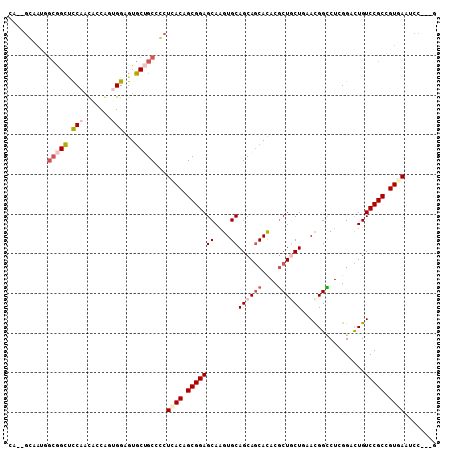
| Location | 1,247,575 – 1,247,678 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 77.55 |
| Mean single sequence MFE | -46.10 |
| Consensus MFE | -28.34 |
| Energy contribution | -28.32 |
| Covariance contribution | -0.03 |
| Combinations/Pair | 1.27 |
| Mean z-score | -1.60 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.48 |
| SVM RNA-class probability | 0.753152 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1247575 103 - 22224390 C---GGAUUCACGGCGGACAAUCCUAGGCCGUUCAGCAGCGUGUGCUGCUGCACUUGCUCCGCUGUGAGCGGCAGCACUCCACUGGCACUGGAGCCGCCAUUGC--UG .---........(((((....(((.(((((((.(((((((....))))))).)).((((((((.....)))).)))).......))).))))).))))).....--.. ( -46.90) >DroPse_CAF1 9183 108 - 1 CGGUGGAUUAACAGCGGACAGACCGAGACCGUUAAGGAGCAUAUGCUGCUGCACCUGCUCCGCUGUGAGGUGCUGCACUCCGGCGUUGUUGGAGCUGCUGUUCCCAUU .(((((...((((((((.((((((...((.((...((((((..(((....)))..)))))))).))..))).)))..(((((((...))))))))))))))).))))) ( -42.90) >DroEre_CAF1 3603 103 - 1 C---GGAUUCACGGCGGACAGUCCCAGGCCGUUCAGCAGCGUGUGCUGCUGCACUUGCUCCGCUGUGAGCGGCAGCACUCCACUGGCAUUGGGGCCGCCAUUGC--UG .---........(((((....(((((((((((.(((((((....))))))).)).((((((((.....)))).)))).......))).))))))))))).....--.. ( -48.10) >DroYak_CAF1 3162 103 - 1 C---GGAUUCACGGCGGACAGUCCCAGCCCGUUCAGCAGGGUGUGCUGCUGCACUUGCUCCGCUGUGAGCGGCAGCACUCCACUGGCAUUGGGGCCGCCAUUGC--UG .---........(((((.((((.(((((((........))).((((((((((..(..(......)..)))))))))))....)))).))))...))))).....--.. ( -45.30) >DroAna_CAF1 1003 100 - 1 CGGCGGGUUGACCGCGGACAGGCCGAGGCCAUUGAGCAGCGUGUGUUGCUGCACUUGCUCCGCGGUCAGGGGCAGAACUCCAUUGGUGUUGACCCC------GU--UG (((((((.(((((((((((((((....))).....((((((.....))))))...)).))))))))))((((.....))))............)))------))--)) ( -50.50) >DroPer_CAF1 9518 108 - 1 CGGUGGAUUAACAGCGGACAGACCGAGACCGUUGAGGAGCAUAUGCUGCUGCACCUGCUCCGCUGUGAGGUGCUGCACUCCGGCGCUGUUGGAGCUGCUGUUCCCAUU .(((((...((((((((.((((((...((.((...((((((..(((....)))..)))))))).))..))).)))..(((((((...))))))))))))))).))))) ( -42.90) >consensus C___GGAUUCACGGCGGACAGUCCGAGGCCGUUCAGCAGCGUGUGCUGCUGCACUUGCUCCGCUGUGAGCGGCAGCACUCCACUGGCAUUGGAGCCGCCAUUGC__UG ....((.(((((((((((................((((((....))))))((....)))))))))))))........(((((.......)))))))............ (-28.34 = -28.32 + -0.03)

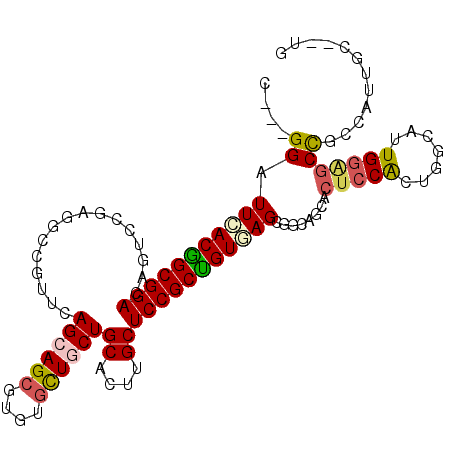
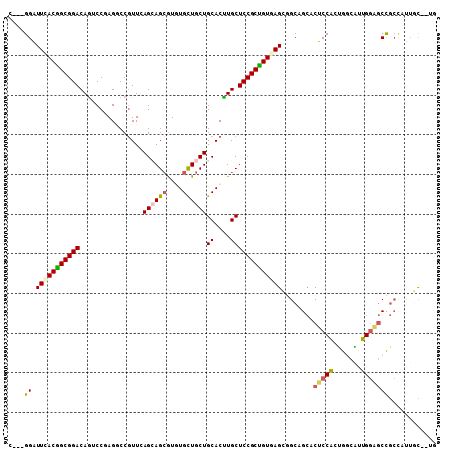
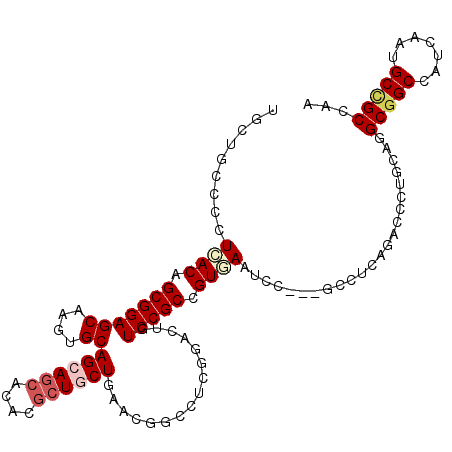
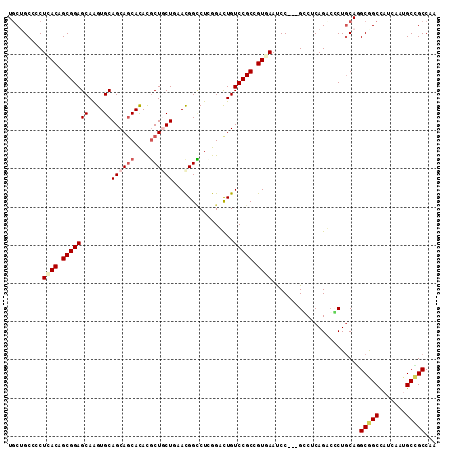
| Location | 1,247,604 – 1,247,713 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 82.11 |
| Mean single sequence MFE | -48.67 |
| Consensus MFE | -25.38 |
| Energy contribution | -26.22 |
| Covariance contribution | 0.83 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.14 |
| Structure conservation index | 0.52 |
| SVM decision value | 0.68 |
| SVM RNA-class probability | 0.819380 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1247604 109 + 22224390 UGCUGCCGCUCACAGCGGAGCAAGUGCAGCAGCACACGCUGCUGAACGGCCUAGGAUUGUCCGCCGUGAAUCC---GGCUCAGUCUCUGCAGGCGGCCAUCAAUGCCGCCUA ((((.((((.....)))))))).((.(((((((....))))))).)).((...((((((...((((......)---))).))))))..))(((((((.......))))))). ( -52.50) >DroPse_CAF1 9214 112 + 1 UGCAGCACCUCACAGCGGAGCAGGUGCAGCAGCAUAUGCUCCUUAACGGUCUCGGUCUGUCCGCUGUUAAUCCACCGCCACAGACGCUGCAAGCGGCCAUCAAUGCUGCCAC .((((((.........((((((.((((....)))).)))))).....((((...((((((..((.((......)).)).))))))((.....)))))).....))))))... ( -42.50) >DroEre_CAF1 3632 109 + 1 UGCUGCCGCUCACAGCGGAGCAAGUGCAGCAGCACACGCUGCUGAACGGCCUGGGACUGUCCGCCGUGAAUCC---GGCUCAGUCCCUGCACGCGGCCAUCAAUGCCGCCUA ((((.((((.....)))))))).((.(((((((....))))))).)).((..(((((((...((((......)---))).))))))).))..(((((.......)))))... ( -56.20) >DroYak_CAF1 3191 109 + 1 UGCUGCCGCUCACAGCGGAGCAAGUGCAGCAGCACACCCUGCUGAACGGGCUGGGACUGUCCGCCGUGAAUCC---GGCUCAGUCCCUGCAGGCGGCCAUCAACGCUGCCUA ((((.((((.....)))))))).((.((((((......)))))).))..((.(((((((...((((......)---))).))))))).))(((((((.......))))))). ( -55.80) >DroAna_CAF1 1026 112 + 1 UUCUGCCCCUGACCGCGGAGCAAGUGCAGCAACACACGCUGCUCAAUGGCCUCGGCCUGUCCGCGGUCAACCCGCCGCCCCAGAGUCUACAGGCAGCUAUCAACGCCGCCAC ....((...((((((((((.((.(.(((((.......))))).)...(((....)))))))))))))))....))................(((.((.......)).))).. ( -42.10) >DroPer_CAF1 9549 112 + 1 UGCAGCACCUCACAGCGGAGCAGGUGCAGCAGCAUAUGCUCCUCAACGGUCUCGGUCUGUCCGCUGUUAAUCCACCGCCACAGACGCUGCAAGCGGCCAUCAAUGCUGCCAC .((((((.......(.((((((.((((....)))).)))))).)...((((...((((((..((.((......)).)).))))))((.....)))))).....))))))... ( -42.90) >consensus UGCUGCCCCUCACAGCGGAGCAAGUGCAGCAGCACACGCUGCUGAACGGCCUCGGACUGUCCGCCGUGAAUCC___GCCUCAGACCCUGCAGGCGGCCAUCAAUGCCGCCAA .........((((.(((((((....))((((((....))))))................))))).)))).......................(((((.......)))))... (-25.38 = -26.22 + 0.83)

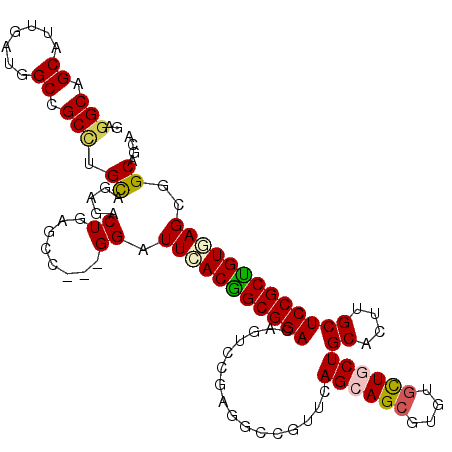
| Location | 1,247,604 – 1,247,713 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 82.11 |
| Mean single sequence MFE | -52.65 |
| Consensus MFE | -29.57 |
| Energy contribution | -29.52 |
| Covariance contribution | -0.05 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.22 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.04 |
| SVM RNA-class probability | 0.552087 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1247604 109 - 22224390 UAGGCGGCAUUGAUGGCCGCCUGCAGAGACUGAGCC---GGAUUCACGGCGGACAAUCCUAGGCCGUUCAGCAGCGUGUGCUGCUGCACUUGCUCCGCUGUGAGCGGCAGCA .(((((((.......)))))))((.....(((.(((---(......))))((.....)))))((((((((.(((((.((((....))))......))))))))))))).)). ( -53.10) >DroPse_CAF1 9214 112 - 1 GUGGCAGCAUUGAUGGCCGCUUGCAGCGUCUGUGGCGGUGGAUUAACAGCGGACAGACCGAGACCGUUAAGGAGCAUAUGCUGCUGCACCUGCUCCGCUGUGAGGUGCUGCA ...(((((((.........((..((((((((((..((.((......)).)).))))))............((((((..(((....)))..))))))))))..))))))))). ( -48.40) >DroEre_CAF1 3632 109 - 1 UAGGCGGCAUUGAUGGCCGCGUGCAGGGACUGAGCC---GGAUUCACGGCGGACAGUCCCAGGCCGUUCAGCAGCGUGUGCUGCUGCACUUGCUCCGCUGUGAGCGGCAGCA ...(((((.......))))).(((.(((((((.(((---(......))))...)))))))..((((((((.(((((.((((....))))......))))))))))))).))) ( -60.90) >DroYak_CAF1 3191 109 - 1 UAGGCAGCGUUGAUGGCCGCCUGCAGGGACUGAGCC---GGAUUCACGGCGGACAGUCCCAGCCCGUUCAGCAGGGUGUGCUGCUGCACUUGCUCCGCUGUGAGCGGCAGCA .((((.((.......)).))))((.(((((((.(((---(......))))...))))))).((((...((((.((((((((....))))..)))).))))...).))).)). ( -54.10) >DroAna_CAF1 1026 112 - 1 GUGGCGGCGUUGAUAGCUGCCUGUAGACUCUGGGGCGGCGGGUUGACCGCGGACAGGCCGAGGCCAUUGAGCAGCGUGUGUUGCUGCACUUGCUCCGCGGUCAGGGGCAGAA ......((.......(((((((.(((...))))))))))...((((((((((((((((....))).....((((((.....))))))...)).)))))))))))..)).... ( -51.00) >DroPer_CAF1 9549 112 - 1 GUGGCAGCAUUGAUGGCCGCUUGCAGCGUCUGUGGCGGUGGAUUAACAGCGGACAGACCGAGACCGUUGAGGAGCAUAUGCUGCUGCACCUGCUCCGCUGUGAGGUGCUGCA ...(((((((.........((..((((((((((..((.((......)).)).))))))............((((((..(((....)))..))))))))))..))))))))). ( -48.40) >consensus GAGGCAGCAUUGAUGGCCGCCUGCAGAGACUGAGCC___GGAUUCACGGCGGACAGUCCGAGGCCGUUCAGCAGCGUGUGCUGCUGCACUUGCUCCGCUGUGAGCGGCAGCA ..(((.((.......)).))).((.....((........)).(((((((((((................((((((....))))))((....)))))))))))))..)).... (-29.57 = -29.52 + -0.05)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:44:20 2006