
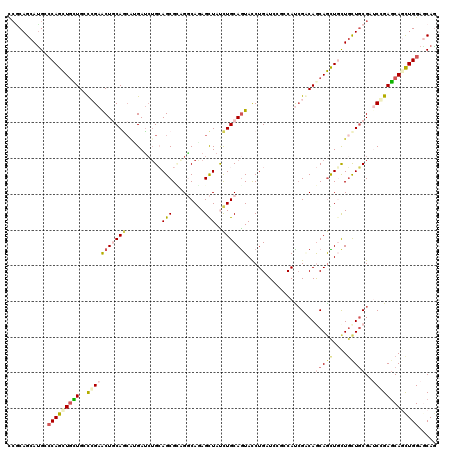
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 11,177,747 – 11,177,867 |
| Length | 120 |
| Max. P | 0.780416 |

| Location | 11,177,747 – 11,177,867 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.28 |
| Mean single sequence MFE | -50.92 |
| Consensus MFE | -23.57 |
| Energy contribution | -24.63 |
| Covariance contribution | 1.06 |
| Combinations/Pair | 1.39 |
| Mean z-score | -2.20 |
| Structure conservation index | 0.46 |
| SVM decision value | 0.56 |
| SVM RNA-class probability | 0.780416 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11177747 120 + 22224390 CCACAGCAUGGCCAGCUUCUGGCCGAACUGCAGCGGGAUCUGGAGCGACGACAAAGCUACCUGCAGUACCUGAUGCGACACAGGCAACAGCUCCUGCUGCGAUCGGAGCAAUUGGAGCAG .....((.(((((((...)))))))...((((((((((.(((..((.........((.....)).....(((........)))))..))).)))))))))).((.((....)).)))).. ( -43.90) >DroVir_CAF1 5412 120 + 1 CCGCAGCAUGCCCAGCUGCUGCUGGAACUCCAGCAUGAUAUGCAGCGCAGGCAGAGCUAUCUGCAGUACCUGCUCGUGCAUCGACAGCAGCUUCUGUUGCGCGCCGAGCAGCUGGAACAG ..((.....))((((((((((((((....)))))..........((((..(((((....))))).((...(((....)))...)).(((((....)))))))))..)))))))))..... ( -52.80) >DroGri_CAF1 5448 120 + 1 CCGCAGCAUGCGCAGCUGUUGCUCGAACUCCAAAAUGAUCGGCAGCGUCGCCAGAGUUACCUGCAGUACCUGCUCGUCCAUCGACAGCAGCUGCUGCUGCGGUCCGAGCAGCUGGAGCAG ((((((((...((((((((((.((((.........((..((((((.(((..(((......)))..).)))))).))..))))))))))))))))))))))))((((......)))).... ( -49.50) >DroWil_CAF1 5264 120 + 1 UGCCAGCAUGCCCAGUUGUUGGCCGAACUGCAACGUGAUCUGCAGCGUAGGCAAAGCUAUUUGCAAUAUCUGAUGCGUCAUCGCCAGCAAUUGCUGCUGCGUACAGAACAGCUGGAUCAG .(((((((........)))))))............(((((..((((....(((((....)))))....((((.(((((....((.(((....))))).)))))))))...))))))))). ( -38.80) >DroMoj_CAF1 5712 120 + 1 CCGCAGCAUGGCCAGCUGCUGCUGGAGCUGCAGCACGAUCUGCAGCGCAGGCAGAGCUAUCUGCAGUACCUGCUCGUGCAUCGCCAGCAGCUGUUGCUGCGCUCCGAGCAGCUGGAGCAA ..((((((....((((((((((((..(((((((......))))))).)))(((((....))))).((((......)))).....))))))))).))))))((((((......)))))).. ( -62.10) >DroAna_CAF1 4527 120 + 1 CCGCAGCACGGCCAGCUGCUGGCCGAACUGCAGCUGGACCUGGAGCGGCGGCAGAGCUAUCUGCAGUACCUGAUGCGCCACCGGCAACAGUUGCUCCUCCGAUCGGAGCAACUGGAGCAG ((((..((.((((((((((.(......).)))))))).))))..))))..(((((....))))).........((((((...)))..((((((((((.......))))))))))..))). ( -58.40) >consensus CCGCAGCAUGCCCAGCUGCUGCCCGAACUGCAGCAUGAUCUGCAGCGCAGGCAGAGCUAUCUGCAGUACCUGAUCCGCCAUCGACAGCAGCUGCUGCUGCGAUCCGAGCAGCUGGAGCAG ...........(((((((((..(((((((((((..........(((.........)))..)))))))...................(((((....)))))..)))))))))))))..... (-23.57 = -24.63 + 1.06)


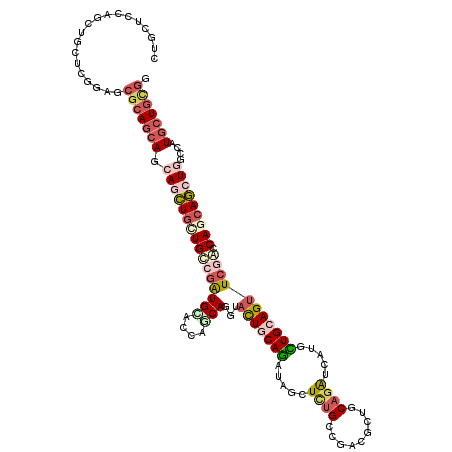
| Location | 11,177,747 – 11,177,867 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.28 |
| Mean single sequence MFE | -52.00 |
| Consensus MFE | -25.64 |
| Energy contribution | -27.65 |
| Covariance contribution | 2.01 |
| Combinations/Pair | 1.35 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.49 |
| SVM decision value | 0.24 |
| SVM RNA-class probability | 0.649334 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11177747 120 - 22224390 CUGCUCCAAUUGCUCCGAUCGCAGCAGGAGCUGUUGCCUGUGUCGCAUCAGGUACUGCAGGUAGCUUUGUCGUCGCUCCAGAUCCCGCUGCAGUUCGGCCAGAAGCUGGCCAUGCUGUGG ..(((((..((((.......))))..))))).((((((((((..((.....))..))))))))))...(..(((......)))..).(..((((..((((((...))))))..))))..) ( -45.20) >DroVir_CAF1 5412 120 - 1 CUGUUCCAGCUGCUCGGCGCGCAACAGAAGCUGCUGUCGAUGCACGAGCAGGUACUGCAGAUAGCUCUGCCUGCGCUGCAUAUCAUGCUGGAGUUCCAGCAGCAGCUGGGCAUGCUGCGG .....((((((((((((((.(((........)))))))))((((.(.(((((((..((.....))..))))))).))))).....((((((....))))))))))))))((.....)).. ( -56.00) >DroGri_CAF1 5448 120 - 1 CUGCUCCAGCUGCUCGGACCGCAGCAGCAGCUGCUGUCGAUGGACGAGCAGGUACUGCAGGUAACUCUGGCGACGCUGCCGAUCAUUUUGGAGUUCGAGCAACAGCUGCGCAUGCUGCGG ......(((((((((((((((((((.((((((((((((....))).)))))...))))..........(....))))))(((.....)))).))))))))...)))))(((.....))). ( -46.30) >DroWil_CAF1 5264 120 - 1 CUGAUCCAGCUGUUCUGUACGCAGCAGCAAUUGCUGGCGAUGACGCAUCAGAUAUUGCAAAUAGCUUUGCCUACGCUGCAGAUCACGUUGCAGUUCGGCCAACAACUGGGCAUGCUGGCA .....((((((((((.((..((..((((....))))((((((.(......).)))))).....))...(((...(((((((......)))))))..))).....)).))))).))))).. ( -39.30) >DroMoj_CAF1 5712 120 - 1 UUGCUCCAGCUGCUCGGAGCGCAGCAACAGCUGCUGGCGAUGCACGAGCAGGUACUGCAGAUAGCUCUGCCUGCGCUGCAGAUCGUGCUGCAGCUCCAGCAGCAGCUGGCCAUGCUGCGG ..(((((........)))))((((((.(((((((((.(.........(((((((..((.....))..)))))))(((((((......)))))))....))))))))))....)))))).. ( -64.60) >DroAna_CAF1 4527 120 - 1 CUGCUCCAGUUGCUCCGAUCGGAGGAGCAACUGUUGCCGGUGGCGCAUCAGGUACUGCAGAUAGCUCUGCCGCCGCUCCAGGUCCAGCUGCAGUUCGGCCAGCAGCUGGCCGUGCUGCGG ..((..((((((((((.......))))))))))..))((((((((((........)))(((....))))))))))............(((((((.(((((((...))))))).))))))) ( -60.60) >consensus CUGCUCCAGCUGCUCGGAGCGCAGCAGCAGCUGCUGCCGAUGCACCAGCAGGUACUGCAGAUAGCUCUGCCGACGCUGCAGAUCAUGCUGCAGUUCGACCAGCAGCUGGCCAUGCUGCGG ...................(((((((.((((((((((((((((....)))...(((((((.....((((.........)))).....))))))))))).)))))))))....))))))). (-25.64 = -27.65 + 2.01)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:20:25 2006