
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 11,174,179 – 11,174,339 |
| Length | 160 |
| Max. P | 0.865908 |

| Location | 11,174,179 – 11,174,299 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.44 |
| Mean single sequence MFE | -45.93 |
| Consensus MFE | -28.77 |
| Energy contribution | -29.17 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.44 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.78 |
| SVM RNA-class probability | 0.850122 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
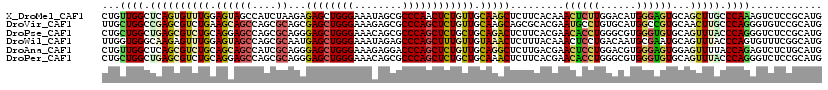
>X_DroMel_CAF1 11174179 120 + 22224390 CAUGCGGAGACUUUGGGCAAGCUGCACUCCCAUGUCCAAGAGUUUGUGAAGAGCUUGCAACAGAGUUGGGCGCUAUUUCCCAGCUCUCUUAGAUGGCUACUCCAAACACUGAGCCAACAG ((((.((((.(....)((.....)).)))))))).......(((((.((..((((......(((((((((........))))))))).......))))..))))))).(((......))) ( -35.12) >DroVir_CAF1 1333 120 + 1 CAUGCGGACACCCUGGGCAAGUUGCACGGCCAUGCACAGGCAUUCGUGCGCUGCUUGCAACAGAGCUGGGCGCUCUUUCCCAGCUCGCUGCGCUGGCUGCUUCAGACGCUCGGCCAGCAA ...((((.(.......(((((((((((((..((((....)))))))))))..))))))....((((((((........)))))))))))))(((((((((.......))..))))))).. ( -55.60) >DroPse_CAF1 3617 120 + 1 CAUGCGGAGACCCUGGGUAAACUGCACACCCACGCCCAGGUGUUCGUGAAGAGUCUGCAGCAGAGCUGGGCGCUGUUUCCCAGCUCCCUGCGCUGGCUCCUGCAGACGCUCAGCCAGCAG ...(((((.(((.(((((...............)))))))).))))).........((((..((((((((........)))))))).))))(((((((...((....))..))))))).. ( -52.96) >DroWil_CAF1 1380 120 + 1 CAUGCCGAAACACUGGGUAAACUGCAUUCGCAUUGUCAGGAGUUUGUAAAGAGUUUACAACAAAGCUGGGCUCUAUUUCCCAGCUCAUUGCGCUGGCUACUCCAAACUCUUGCCCACCAA .............(((((((.(((((.......)).)))(((((((....((((...((.((((((((((........)))))))..)))...))...)))))))))))))))))).... ( -35.00) >DroAna_CAF1 912 120 + 1 CAUGCAGAGACUCUGGUAAAACUCCACUCCCACGUCCAGGAGUUCGUCAAGAGCCUGCAACAGAGCUGGGUCCUCUUUCCCAGCUCCCUGCGAUGGCUGCUGCAGACGCUGAGCCAACAG ..(((((.(.((((.....((((((.............)))))).....))))))))))...((((((((........)))))))).(((...(((((.(.((....)).)))))).))) ( -43.92) >DroPer_CAF1 3499 120 + 1 CAUGCGGAGACCCUGGGUAAACUGCACACCCACGCCCAGGUGUUCGUGAAGAGUUUGCAGCAGAGCUGGGCGCUGUUUCCCAGCUCCCUGCGCUGGCUCCUGCAGACGCUCAGCCAGCAG ...(((((.(((.(((((...............)))))))).))))).........((((..((((((((........)))))))).))))(((((((...((....))..))))))).. ( -52.96) >consensus CAUGCGGAGACCCUGGGUAAACUGCACUCCCACGCCCAGGAGUUCGUGAAGAGCUUGCAACAGAGCUGGGCGCUAUUUCCCAGCUCCCUGCGCUGGCUACUGCAGACGCUCAGCCAGCAG ...((((.........(((((((.(((....((((....))))..)))...)))))))....((((((((........)))))))).))))(.(((((.............))))).).. (-28.77 = -29.17 + 0.40)


| Location | 11,174,179 – 11,174,299 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.44 |
| Mean single sequence MFE | -47.43 |
| Consensus MFE | -30.77 |
| Energy contribution | -30.83 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.50 |
| Mean z-score | -1.30 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.00 |
| SVM RNA-class probability | 0.535044 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11174179 120 - 22224390 CUGUUGGCUCAGUGUUUGGAGUAGCCAUCUAAGAGAGCUGGGAAAUAGCGCCCAACUCUGUUGCAAGCUCUUCACAAACUCUUGGACAUGGGAGUGCAGCUUGCCCAAAGUCUCCGCAUG .......((.((.((((((((.(((....(((.((((.((((........)))).)))).)))...))).))).))))).)).)).((((((((....((((.....)))))))).)))) ( -35.60) >DroVir_CAF1 1333 120 - 1 UUGCUGGCCGAGCGUCUGAAGCAGCCAGCGCAGCGAGCUGGGAAAGAGCGCCCAGCUCUGUUGCAAGCAGCGCACGAAUGCCUGUGCAUGGCCGUGCAACUUGCCCAGGGUGUCCGCAUG ..((((((...((.......)).))))))(((((((((((((........)))))))).))))).....(((.(((....((((.(((..((...))....))).)))).))).)))... ( -52.20) >DroPse_CAF1 3617 120 - 1 CUGCUGGCUGAGCGUCUGCAGGAGCCAGCGCAGGGAGCUGGGAAACAGCGCCCAGCUCUGCUGCAGACUCUUCACGAACACCUGGGCGUGGGUGUGCAGUUUACCCAGGGUCUCCGCAUG ..(((((((..((....))...)))))))(((((((((((((........))))))))).))))(((((((..(((..(....)..)))(((((.......))))))))))))....... ( -55.30) >DroWil_CAF1 1380 120 - 1 UUGGUGGGCAAGAGUUUGGAGUAGCCAGCGCAAUGAGCUGGGAAAUAGAGCCCAGCUUUGUUGUAAACUCUUUACAAACUCCUGACAAUGCGAAUGCAGUUUACCCAGUGUUUCGGCAUG ..(((((((..(((((((((((...(((((....((((((((........)))))))))))))...))))....))))))).......(((....))))))))))..((((....)))). ( -38.50) >DroAna_CAF1 912 120 - 1 CUGUUGGCUCAGCGUCUGCAGCAGCCAUCGCAGGGAGCUGGGAAAGAGGACCCAGCUCUGUUGCAGGCUCUUGACGAACUCCUGGACGUGGGAGUGGAGUUUUACCAGAGUCUCUGCAUG .(((.(((((((.(((((((((.((....))..(((((((((........)))))))))))))))))).)).(((..((((((......))))))...)))......)))))...))).. ( -51.90) >DroPer_CAF1 3499 120 - 1 CUGCUGGCUGAGCGUCUGCAGGAGCCAGCGCAGGGAGCUGGGAAACAGCGCCCAGCUCUGCUGCAAACUCUUCACGAACACCUGGGCGUGGGUGUGCAGUUUACCCAGGGUCUCCGCAUG ..(((((((..((....))...)))))))((.((((((((((........)))))))).((((((.(((...((((..(....)..))))))).))))))....)).((....))))... ( -51.10) >consensus CUGCUGGCUGAGCGUCUGCAGCAGCCAGCGCAGGGAGCUGGGAAACAGCGCCCAGCUCUGUUGCAAACUCUUCACGAACUCCUGGGCAUGGGAGUGCAGUUUACCCAGGGUCUCCGCAUG ...((((.((((((((((.((((((....))...((((((((........)))))))))))).))))).........((((((......))))))...))))).))))............ (-30.77 = -30.83 + 0.06)


| Location | 11,174,219 – 11,174,339 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.28 |
| Mean single sequence MFE | -52.05 |
| Consensus MFE | -32.10 |
| Energy contribution | -33.35 |
| Covariance contribution | 1.25 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.60 |
| SVM RNA-class probability | 0.794408 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11174219 120 + 22224390 AGUUUGUGAAGAGCUUGCAACAGAGUUGGGCGCUAUUUCCCAGCUCUCUUAGAUGGCUACUCCAAACACUGAGCCAACAGCUCCGGCAGUCGUUGCGCCACGAGGAGCAGGAGAUCCGCC .(((((.((..((((......(((((((((........))))))))).......))))..)))))))...((((.....)))).(((.(((.((((.((....)).))))..)))..))) ( -41.42) >DroVir_CAF1 1373 120 + 1 CAUUCGUGCGCUGCUUGCAACAGAGCUGGGCGCUCUUUCCCAGCUCGCUGCGCUGGCUGCUUCAGACGCUCGGCCAGCAACUGCGUCAGACGCAACGUUACAGCGACUAUGAGAUACGUC ..(((((((((((...(((...((((((((........))))))))..)))(((((((((.......))..)))))))...((((.....))))......))))).).)))))....... ( -47.70) >DroPse_CAF1 3657 120 + 1 UGUUCGUGAAGAGUCUGCAGCAGAGCUGGGCGCUGUUUCCCAGCUCCCUGCGCUGGCUCCUGCAGACGCUCAGCCAGCAGCUGCGGCAGACGCAUCGCCACAAAGAGCAGGAGAUCCGGC (((((((((.(.((((((.((((.((.(((.((((.....)))).))).))(((((((...((....))..)))))))..)))).)))))).).))))......)))))((....))... ( -56.50) >DroMoj_CAF1 2121 120 + 1 GCUUUGUGCGCAGCCUGCAGCAGAGCUGGGCCUUGUUUCCCAGCUCGCUGCGCUGGCUGCUGCAGACGCUCGGGCAGCAGCUGCGGCAGACGCAGCGCCACAGCGACCACGAGAUACGCC ((..(.(((((((((((((((.((((((((........))))))))))))))..)))))).))).)..((((((..((.((((((.....))))))......))..)).))))....)). ( -62.80) >DroAna_CAF1 952 120 + 1 AGUUCGUCAAGAGCCUGCAACAGAGCUGGGUCCUCUUUCCCAGCUCCCUGCGAUGGCUGCUGCAGACGCUGAGCCAACAGCUCCGGCAGAGCUUGCGCUACGAGGAGCAGGAGAUACGGC ...((((......(((((....((((((((........))))))))((((((..((((.((((.((.((((......))))))..))))))))..)))....))).)))))....)))). ( -50.00) >DroPer_CAF1 3539 120 + 1 UGUUCGUGAAGAGUUUGCAGCAGAGCUGGGCGCUGUUUCCCAGCUCCCUGCGCUGGCUCCUGCAGACGCUCAGCCAGCAGCUGCGGCAGACGCAUCGCCACAAAGAGCAGGAGAUCCGGC (((((((((.(.((((((.((((.((.(((.((((.....)))).))).))(((((((...((....))..)))))))..)))).)))))).).))))......)))))((....))... ( -53.90) >consensus AGUUCGUGAAGAGCCUGCAACAGAGCUGGGCGCUGUUUCCCAGCUCCCUGCGCUGGCUGCUGCAGACGCUCAGCCAGCAGCUGCGGCAGACGCAGCGCCACAAAGAGCAGGAGAUACGGC .((((.....((((..(((...((((((((........))))))))..)))(((((((...((....))..))))))).)))).(((.........))).....))))............ (-32.10 = -33.35 + 1.25)



| Location | 11,174,219 – 11,174,339 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.28 |
| Mean single sequence MFE | -54.55 |
| Consensus MFE | -33.48 |
| Energy contribution | -33.73 |
| Covariance contribution | 0.26 |
| Combinations/Pair | 1.37 |
| Mean z-score | -2.25 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.85 |
| SVM RNA-class probability | 0.865908 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 11174219 120 - 22224390 GGCGGAUCUCCUGCUCCUCGUGGCGCAACGACUGCCGGAGCUGUUGGCUCAGUGUUUGGAGUAGCCAUCUAAGAGAGCUGGGAAAUAGCGCCCAACUCUGUUGCAAGCUCUUCACAAACU (((((.((...(((.((....)).)))..))))))).((((.....))))...((((((((.(((....(((.((((.((((........)))).)))).)))...))).))).))))). ( -43.10) >DroVir_CAF1 1373 120 - 1 GACGUAUCUCAUAGUCGCUGUAACGUUGCGUCUGACGCAGUUGCUGGCCGAGCGUCUGAAGCAGCCAGCGCAGCGAGCUGGGAAAGAGCGCCCAGCUCUGUUGCAAGCAGCGCACGAAUG .............((((((((....(((((.....)))))..((((((...((.......)).))))))(((((((((((((........)))))))).)))))..)))))).))..... ( -52.40) >DroPse_CAF1 3657 120 - 1 GCCGGAUCUCCUGCUCUUUGUGGCGAUGCGUCUGCCGCAGCUGCUGGCUGAGCGUCUGCAGGAGCCAGCGCAGGGAGCUGGGAAACAGCGCCCAGCUCUGCUGCAGACUCUUCACGAACA ((.((....)).))..(((((((.((...((((((.((.((.(((((((..((....))...)))))))))..(((((((((........))))))))))).)))))))).))))))).. ( -56.70) >DroMoj_CAF1 2121 120 - 1 GGCGUAUCUCGUGGUCGCUGUGGCGCUGCGUCUGCCGCAGCUGCUGCCCGAGCGUCUGCAGCAGCCAGCGCAGCGAGCUGGGAAACAAGGCCCAGCUCUGCUGCAGGCUGCGCACAAAGC (((((.((..(..((.(((((((((.......))))))))).))..)..)))))))(((.((((((...(((((((((((((........)))))))).))))).)))))))))...... ( -67.30) >DroAna_CAF1 952 120 - 1 GCCGUAUCUCCUGCUCCUCGUAGCGCAAGCUCUGCCGGAGCUGUUGGCUCAGCGUCUGCAGCAGCCAUCGCAGGGAGCUGGGAAAGAGGACCCAGCUCUGUUGCAGGCUCUUGACGAACU ((((...(....(((((.((.(((....))).))..))))).).))))((((.(((((((((.((....))..(((((((((........)))))))))))))))))).).)))...... ( -53.50) >DroPer_CAF1 3539 120 - 1 GCCGGAUCUCCUGCUCUUUGUGGCGAUGCGUCUGCCGCAGCUGCUGGCUGAGCGUCUGCAGGAGCCAGCGCAGGGAGCUGGGAAACAGCGCCCAGCUCUGCUGCAAACUCUUCACGAACA ((.((..(((((((((.....)).(((((.((.(((((....)).))).))))))).))))))))).))(((((((((((((........))))))))).))))................ ( -54.30) >consensus GCCGGAUCUCCUGCUCCUUGUGGCGCUGCGUCUGCCGCAGCUGCUGGCUGAGCGUCUGCAGCAGCCAGCGCAGGGAGCUGGGAAACAGCGCCCAGCUCUGCUGCAAGCUCUUCACGAACA ................((((((((.......(((.(((.(((((((............)))))))..))))))(((((((((........)))))))))))))))))............. (-33.48 = -33.73 + 0.26)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:20:21 2006